2010年6月14日
独立行政法人 理化学研究所
ナノグラムレベルのRNAから遺伝子発現をキャッチする新解析手法を確立
-2つの新たな解析手法がRNAの網羅的な機能解析を促進-
ポイント
- nanoCAGE法でナノグラムを解析、CAGEscan法で5´と3´側の両配列を決定
- 未知のトランスクリプトーム解析や細胞核と細胞質のRNAの比較を実現
- RNAの研究を促進させ、がんの個別診療など医療分野への応用に期待
要旨
独立行政法人理化学研究所(野依良治理事長)は、イタリアのSISSA※1(Scuola Internazionale Superiore di Studi Avanzati)と共同で、従来のCAGE法※2の感度を1,000倍以上向上させ、ナノグラムレベル(ng:1ngは10-9グラム)のmRNAを解析して遺伝子の転写開始点を決定することができるnanoCAGE法と、mRNA鎖の5´末端※3と3´側※3の両方の配列を一度に解析することができるCAGEscan法という2つの高感度な新遺伝子発現解析方法を開発しました。理研オミックス基盤研究領域(OSC、林崎良英領域長)のピエロ・カルニンチ チームリーダーと SISSA神経生物学部門のステファノ・グスティンチッチ(Stefano Gustincich)博士らの共同研究による成果です。
nanoCAGE法は、現行のCAGE法などの遺伝子発現解析法がマイクログラムレベル(μg:1μgは10-6グラム)のRNAの量を必要とするのに対し、ngレベルのRNAの量でも解析できるように設計した技術で、DNAの断片であるプライマー配列の工夫とcDNAの特異的な増幅法の導入によって、CAGE法と比較して1,000倍以上の感度向上を実現しました。このため、単一の細胞に存在している微量なmRNAの5´末端を検出し、関連するプロモーター※4をゲノム上で同定することができます。このことは、個々の細胞内の遺伝子発現がどのように制御されているかを網羅的に知ることで、例えば、神経細胞やがんを発症している細胞などで発現する微量なRNAを解析することを可能にします。
一方CAGEscan法は、nanoCAGE法と同様に微量なmRNAを解析できる上に、次世代シーケンサーの能力を活用し、5´末端とその反対側である3´側の塩基配列の両方を同時に解析できるというメリットがあります。その結果、5´末端が、自分自身やほかのmRNAの3´側の塩基配列に結合する様子を明らかにして、mRNA間の相互作用を理解することができるようになります。
さらに、CAGE法では、すべてのRNAを逆転写した後、mRNAだけを分離する必要があるため、解析終了までに約5日の日数を費やしますが、nanoCAGE法とCAGEscan法は、mRNA だけを逆転写することができるため、解析に要する時間を2~3日と短縮することができます。
両手法を導入することで、研究グループが「RNA新大陸※5」と名付けた全遺伝子の半分以上を占めるncRNA※6に関する知見を増やし、これまで、ほとんど不明確なまま取り残されてきたRNAの理解が飛躍的に向上すると期待できます。
この成果は、英国の科学雑誌『Nature Method』の7月号に掲載されるに先立ち、オンライン版(6月13日付:日本時間6月14日)に掲載されました。
背景
研究グループは、ヒトをはじめとするほ乳類のゲノム解読の結果を基に、従来100個ぐらいしか知られていなかったncRNAが、実は23,000個以上、つまり、全遺伝子の半分以上(53%)を占めているという新しい事実が明らかにしてきました(2005年9月2日プレス発表)。「RNA新大陸」と名付けたこの発見は、タンパク質に翻訳されない膨大なRNAの重要性を明らかにしたもので、転写産物全体が織り成すネットワークの複雑さを再認識させるものでした。
この発見に研究グループが用いた独自技術のCAGE法では、まず、転写されたRNA(mRNA、rRNA、tRNA、ncRNAなど)を逆転写後、mRNAだけを分離・増幅してcDNAライブラリーを作製します。次に、先頭部分(Capサイト)である5´末端の20~27塩基を切り出し、その配列を次世代シーケンサーで解析します。この解析結果をゲノム配列と比較することで、ゲノム上の転写開始点を正確に同定することができると同時に、その転写開始点ごとの発現量をほぼ定量的に計測することもできます。しかし、CAGE法で解析するには、μgレベルのRNA量が必要になるとともに、5´末端の反対側である3´側の塩基配列を解析することができないため、全RNAを網羅的に解析するためには不十分でした。
研究手法
研究グループは、CAGE法の解析能力の向上を図り、ngレベルの量のRNAの解析と3´側の塩基配列の同時解析、さらに解析スピードの向上を目指しました。
具体的には、rRNAやtRNAなどを含めたすべての種類のRNAのうち、mRNAからのcDNAだけを特異的に逆転写することが可能なTemplate switch法(図1)と、この方法で逆転写したcDNAだけを増幅するSemisuppressive法(図2)を導入しました。その結果、ngレベルという非常に少ない量のmRNAから5´末端の27塩基のタグ配列を切り出し、次世代シーケンサーを用いて大規模に転写開始点を決定することができるnanoCAGE法の開発に成功しました(図3)。
さらに、次世代シーケンサーの解析能力の向上にあわせて150塩基以上のcDNAライブラリーを作製し、CAGEscan法を確立しました(図4)。5´末端と同時に3´側の塩基配列も解析できるようになり、着目したmRNAがどの遺伝子のどの領域に結合しているかを解析できるようになりました。
研究成果
研究グループは、ヒト白血病K562細胞にnanoCAGE法を適用し、約100~500ngという微量ではあるものの、核内にある核小体、核細胞質、染色体やrRNAの構成要素であり、mRNAの末端を意味するpolyAテール※7を持たないポリソームRNA※8などの転写開始点を調べることに成功しました。このように、これまで未知であった部分のRNAという転写産物全体の解析が可能となるだけでなく、生命の維持活動に欠かせない神経細胞などが産出する微量なmRNAの機能も解析することができるようになりました。
また、CAGEscan法を使って、ヒト肝細胞がんHep G2細胞の細胞核と細胞質にあるmRNAを解析したところ、細胞核内にあるmRNAは細胞質にあるmRNAに比べ、タンパク質に翻訳されないゲノム領域と多く結合していることが明らかになりました。このことは、細胞核内ではタンパク質翻訳以外の機能を持つゲノム領域も重要な役割を果たしていることを意味します。
これらの2つの新手法を用いることにより、発見されたばかりで謎の多いRNA新大陸がもたらす生体内のメカニズムをより深く理解することができると考えています。
今後の期待
nanoCAGE法はmRNAを特異的に増幅するため、これまで解析できなかった微量なmRNAを容易に解析できるようになります。一方CAGEscan法は、mRNAの両側の配列情報を明らかにするため、mRNA間の相互作用が明らかになり、例えば、ヒトのがん細胞など無秩序な増殖のメカニズム解明に貢献すると期待されます。
今後、これらの2つの新たな生物学的解析方法を駆使すると、RNAの制御メカニズムに関する詳細な研究を展開することができるようになるとともに、将来的には、がんに対する個別診療など、医療分野への応用に貢献する手法になると考えています。
発表者
理化学研究所
オミックス基盤研究領域 ゲノム機能研究チーム
チームリーダー Piero Carninci(ピエロ・カルニンチ)
Tel: 045-503-9222 / Fax: 045-503-9216
お問い合わせ先
横浜研究推進部 企画課Tel: 045-503-9117 / Fax: 045-503-9113
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.SISSA
イタリアにおける代表的な研究機関(Scuola Internazionale Superiore di Studi Avanzati)で、国際的にも著名な機関。大学院レベルの研究に特化。物理学、数学、神経学の分野で最先端の研究を行っている。また、イタリアで最初に博士号を授与した教育機関でもあり、注目すべき多大な成果をあげている。1978年の創立以来、数多くの博士を研究および教育分野に輩出している。 - 2.CAGE法
Cap Analysis of Gene Expressionの略。理研オミックス基盤研究領域が開発した方法で、耐熱性逆転写酵素やcap-trapper法を組み合わせて、5´末端から20塩基のタグ配列を切り出し、塩基配列を決定する実験技法。この塩基配列を読み取ってゲノム配列と照らし合わせ、どの部分がコピーされているかを調べることができる。 - 3.5´末端、3´側
DNA、RNAは塩基の糖の部分の5´側から3´側に向かって合成が進み、mRNAの5´末端の塩基にはCAP構造が、3´末端の塩基には例外を除いてほとんどの場合polyAテール配列が付加されている。ここでいう3´側とは、このpolyAテールを除いた塩基配列の部分を指す。 - 4.プロモーター
mRNA合成の開始に関与するDNA上の特定領域の短い塩基配列のこと。プロモーター領域にRNAを合成する酵素であるRNAポリメラーゼが結合し、転写が開始される。 - 5.RNA新大陸
多様な細胞内RNA集団の莫大(ばくだい)な可能性を示す比喩的表現。2005年9月2日のプレス発表で、遺伝子の定義を新たに提案した際に用いた新用語。細胞が生産するRNAに関し、今までにない大規模なスケールで調べたところ、従来100個ぐらいしか知られていなかったncRNAが、実は23,000個以上、つまり、全遺伝子の半分以上(53%)を占めているという新しい事実を示したものだった。このことは、タンパク質がゲノムにコードされている最終生理活性物質であるというこれまでの常識を覆し、予想を凌ぐトランスクリプトームの複雑さを認識させるもので、ほ乳動物ゲノムの情報内容に対するこれまでの理解(「遺伝子」という領域が散在しているゲノムのイメージ)を根幹から変えてしまうものだった。 - 6.ncRNA
非タンパク質コードRNA(non-coding RNA)のことで、このRNAからはタンパク質は翻訳されない。 - 7.polyAテール
mRNAの3´末端に50~200塩基ほどのアデニン(A)ヌクレオチドが付加されており、これをpolyAテールと呼ぶ。mRNAに安定性を与え、翻訳を促進する働きがあると考えられている。 - 8.ポリソームRNA
リボソームは転写されているmRNAに速やかに集まり翻訳を開始する。一本のmRNAに複数のリボソームが連結した状態をポリソーム(polysome)と呼ぶ。
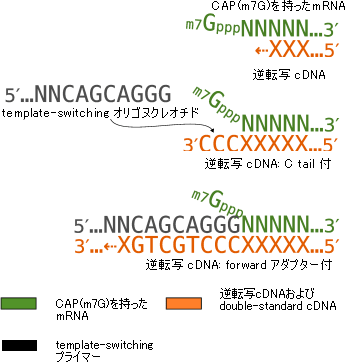
(図1)Template switching法
5´末端にCAP(m7G)を持ったmRNA(緑)が逆転写反応を起こしてcDNA(オレンジ)に逆転写されるとき(上)、逆転写酵素はCAPと相補鎖のシトシン(C)を付加する(中)。それぞれのcDNAの5´末端に付加されたシトシンは、リボグアノシン(G)というCAPと疑似的な配列を持つtemplate-switchingプライマー(黒)と相補鎖を作るため、cDNAの両末端は違う配列が増幅される(下)。この時点では長さの調整はできず、150塩基以下の短いものも、それ以上の長いものも逆転写される。長さの調整は図2のTemplate switchingで行われる。
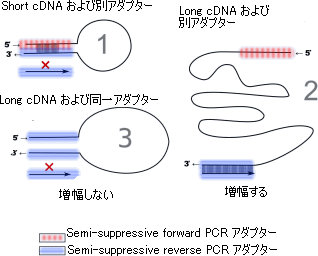
(図2)Semisuppressive PCR反応
150塩基以下の短いcDNAテンプレートは、末端の塩基同士が容易に相補鎖を形成するため(1)、プライマーがcDNAテンプレートと相補鎖を形成できずPCR反応が進まない。また、150塩基以上のcDNAテンプレートでも、逆転写反応中に両末端が template-switching オリゴヌクレオチドに置換されてしまったもの(3)はプライマーが相補鎖を形成できず、PCR反応が進まない。template-switching法で相補鎖を形成したcDNAテンプレート(逆転写反応で末端が別々のプライマーに置換された150塩基以上のcDNAテンプレート)だけがPCR反応で増幅される(2)。
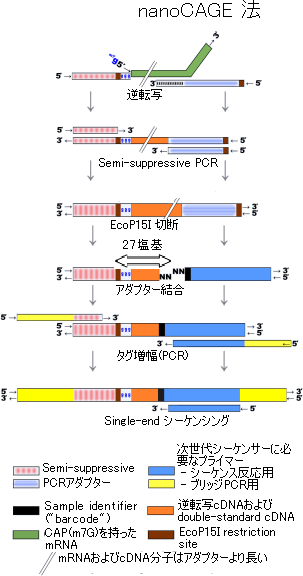
(図3)nanoCAGE法
Template switching反応とSemisuppressive PCR反応後、EcoP15I酵素で切断した27塩基のタグに、次世代シーケンサーIllumina Genome AnalyzerSingle read kitで配列を読むためのアダプターとプライマーを付加し、解析する。
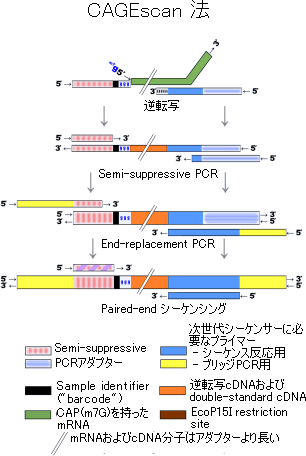
(図4)CAGEscan法
nanoCAGE法と同様、Template switching反応とSemisuppressive PCR反応を行う。その後、EcoP15I酵素で切断することなく、次世代シーケンサーIllumina Genome Analyzer Paird End kitで、5´末端と3´側の両方を解析する。
