2012年1月31日
独立行政法人 理化学研究所
1分子DNAシーケンサーを使ったCAGE解析の自動化に成功
-遺伝子発現を定量解析するCAGE解析のスピードが約5倍向上-
ポイント
- 自動化システム構築により、サンプル調製日数を42日間から8日間に短縮
- ヒューマンエラーの解消により、解析データの信頼性向上に期待
- 技術支援組織GeNASを通じて、外部の研究者にも技術を提供
要旨
独立行政法人理化学研究所(野依良治理事長)は、1分子DNAシーケンサー※1を活用した「HeliScopeCAGE法」のサンプル調製工程を自動化し、調製期間を従来の実働42日間から8日間に短縮しました。その結果、遺伝子転写開始点を解析するCAGE法※2の解析スピードが約5倍向上しました。これは理研オミックス基盤研究領域(OSC:林崎良英領域長)LSAシステム構築グループ オミックス制御研究ユニットの伊藤昌可ユニットリーダーらによる成果です。
2011年にOSCは、ゲノム全体の遺伝子転写開始点とその発現量の解析が可能な独自技術のCAGE法を1分子DNAシーケンサーに適用し、より正確な解析が可能な「HeliScopeCAGE法」を開発しました。しかし、サンプル調製工程は手動であり、実働42日間という長時間を要するため、これを短縮できるシステムの構築が求められていました。
研究ユニットは、1度で解析するサンプル数を増やすために、8個のチューブが連結した従来の8連チューブストリップから96穴(8×12)プレートに変更し、また、さまざまな温度調節が可能なサンプル調製用プラットフォームと、独立して8本の分注を同時に行うことができるロボットアーム(マニピュレーター)を新たに採用しました。その結果、サンプル調製の全工程を自動化することに成功し、調製にかかる日数を従来の実働42日間から8日間に短縮しました。手動と自動化による解析結果の違いを比較するために、同じサンプルを用いてCAGE解析を行ったところ、両者に違いはなく高い再現性を確認しました。また、自動化によってヒューマンエラーがなくなり、データの信頼性向上も期待できるようになりました。
開発した技術は他のシーケンサーにも展開が可能で、理研OSCの技術支援サービスGeNAS※3を通じて、外部のさまざまな研究機関へも提供していきます。
本研究成果は、米国のオンライン雑誌『PLoS ONE』(1月30日付け)に掲載されます。
背景
DNA塩基配列を高速かつ正確に同定するDNAシーケンサーの技術が急速に進歩し、ゲノムの大規模解析は、遺伝学や医療などのライフサイエンス分野に限らず、環境など他の分野でも頻繁に使われる研究手法となりました。
2011年に研究ユニットは、たった1個のDNAから塩基配列を同定する1分子DNAシーケンサーであるヘリコスバイオサイエンス社製「HeliScope」に独自技術であるCAGE法を最適化し、わずか100ナノグラムのRNAサンプルからPCRなどの増幅なしで遺伝子発現量を定量的に測定する「HeliScopeCAGE法」を開発しました(2011年5月19日プレスリリース)。しかし、最新のシーケンサーが高速で塩基配列を同定するのに対し、そのサンプル調製には長い時間と手間がかかるという課題がありました。
研究グループは、解析のさらなるスピードアップとデータの高い再現性を目的に、サンプル調製工程の自動化に取り組みました。
研究手法と成果
HeliScopeでは1回に48サンプルの解析が可能ですが、従来の手動技術では、1人が16サンプルを調製するのに7日間を要し、48サンプルを調製するには21日間必要でした。そこで、1度に解析するサンプル数を増やすため、8種類しか調製できなかった8連チューブストリップから96種類を調製できる96穴プレートに変更しました。また、サンプル調製工程のうち、液体サンプルの操作、インキュベーション、サンプル精製の工程を自動化するために、テカン社製Freedom Evo 150プラットフォームをもとに、1度に8本の試薬を独立して分注可能なロボットアームを採用しました。架台には、一定の温度調節が可能な場所と、液体保存部分を備えました。また、RNAを相補的なDNAに変換する逆転写反応には複雑な温度調節プログラムが必要となるため、自動開閉加熱ボンネットを装着したプログラム可能な自動温度調節インキュベーターを併設しました。さらに、調製したcDNAを定量するための自動計測可能な蛍光プレートリーダーも装備しました(図1)。こうして、CAGE法のサンプル調製に必要な全ての工程(逆転写反応、酸化反応、ビオチン化反応、cap捕捉技術、核酸の精製操作、cDNAの定量など)を自動化することができました(図2)。さらに、同じサンプルを自動で調製したものと手動で調製したものを用意し、CAGE解析して比較した結果、転写開始点の位置の同定やその発現量に違いはなく、高い再現性を確認することができました(図3)。
サンプル調製工程の自動化で、1人が96サンプルを8日間で調製できるようになり、手動で要する実働42日間と比べ約5倍効率化したことになります。また、サンプルの取り違えなどヒューマンエラーを未然に防ぐことができ、生産するデータの信頼性向上が期待できます。
今後の期待
このサンプル調製工程の自動化は、機種が異なる次世代シーケンサーに適用したCAGE法へも展開が可能です。また、RNAの塩基配列を同定するRNA-seqや、転写物の全ての情報を含む完全長cDNA解析などの遺伝子解析法にも応用できます。今後、さまざまな遺伝子解析のスピードアップとデータの信頼性向上に貢献すると同時に、理研OSCの技術支援サービスGeNASを通じて、外部のさまざまな研究機関へも提供していきます。
原論文情報
- Masayoshi Itoh, Miki Kojima, Sayaka Nagao-Sato, Eri Saijo, Timo Lassmann, Mutsumi Kanamori-Katayama, Ai Kaiho, Marina Lizio, Hideya Kawaji, Piero Carninci, Alistair R R Forrest, Yoshihide Hayashizaki. “Automated workflow for preparation of cDNA for cap analysis of gene expression on a single molecule sequencer”. PLoS ONE, 2012, doi: 10.1371/journal.pone.0030809
発表者
理化学研究所
オミックス基盤研究領域
LSAシステム構築グループ オミックス制御研究ユニット
ユニットリーダー 伊藤 昌可(いとう まさよし)
Tel: 045-503-9222 / Fax: 045-503-9216
お問い合わせ先
横浜研究推進部 企画課
Tel: 045-503-9117 / Fax: 045-503-9113
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.1分子DNAシーケンサー
DNAを増幅することなしに、たった1個のDNAから塩基配列を決定することができるシーケンサー。 - 2.CAGE法
Cap Analysis of Gene Expressionの略。理研オミックス基盤研究領域が開発した方法で、耐熱性逆転写酵素やcap捕捉技術を組み合わせて、5´末端の塩基配列を決定する実験技法。この塩基配列を読み取ってゲノム配列と照らし合わせ、どこからどの位コピーが始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる世界唯一の解析技術。 - 3.GeNAS
Gene Network Analysis Serviceの略。理研オミックス基盤研究領域が構築した網羅的なゲノム・トランスクリプトーム解析技術からなる解析システム『ライフサイエンスアクセラレーター(LSA)』のもと、その要素技術である独自のサンプル調製技術、超高速大規模シーケンス基盤および情報処理パイプラインを受益者負担で技術支援するサービス。2009年9月に開始。

図1 自動化したサンプル調製プラットフォーム
テカン社製Freedom Evo 150をもとに開発。8チャンネル独立分注可能なロボットアーム、温度調節可能部位と液体保存部分を設置した架台、自動開閉加熱ボンネット付プログラム可能インキュベーター、蛍光プレートリーダーが付属する。
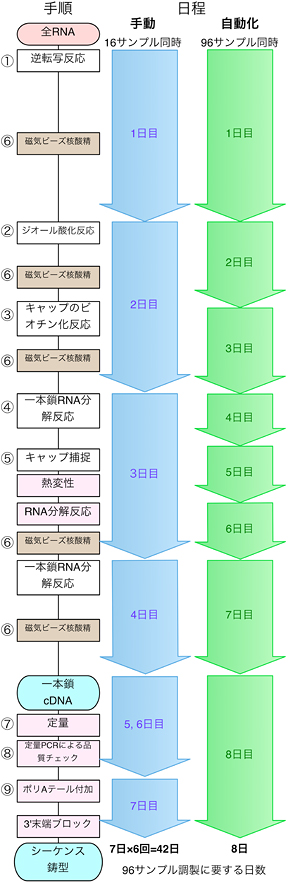
図2 自動と手動のサンプル調製法の手順の比較
CAGE法の手動と自動化工程の時系列比較。①RNAからcDNAを作製する逆転写反応、②キャップ構造のジオール基の酸化反応、③キャップ構造にビオチンを付加するビオチン化反応、④1本鎖RNA部分を消化するRNase I反応、⑤ビオチン化されたキャップ構造を有するRNA/DNA2本鎖を特異的に選別するキャップ捕捉技術、⑥磁気ビーズを用いた核酸精製、⑦1本鎖DNAを定量するOliGreenアッセイ、⑧品質をチェックする定量PCR、そして⑨HeliScopeに供するために必要なポリAテールを付加する工程からなる。手動では一度に16サンプルを7日間で調製するため、48サンプルの調製に実働21日かかるが、自動化工程では一度に96サンプルを8日間で調製できる。
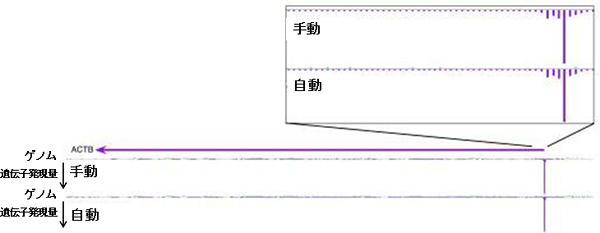
図3 ゲノム上の転写開始点の位置と遺伝子発現量の比較
手動の場合も自動の場合も、転写開始点の位置と発現量に違いは無く、再現性を確認した。横軸はゲノム上の位置、縦軸は遺伝子発現量(下向き)。
