ポイント
- ミナトカモジグサの完全長cDNAを大規模に解析
- コムギ、オオムギのゲノム情報と統合し、相互比較を可能にするデータベースを公開
- 麦類およびバイオマス植物研究を加速する研究基盤を構築
要旨
理化学研究所(理研、野依良治理事長)は、麦類研究とバイオマス研究のモデル植物であるミナトカモジグサ(Brachypodium distachyon、ブラキポディウム)の完全長cDNA[1]を大規模に解析し、約1万種類の遺伝子に対応する完全長cDNAを同定するとともに、これまでの遺伝子構造予測を大幅に刷新しました。また、同定した完全長cDNAをデータベース化し公開しました。これは理研環境資源科学研究センター(篠崎一雄センター長)バイオマス工学連携部門バイオマス研究基盤チームの篠崎一雄チームリーダー、持田恵一副チームリーダー(横浜市立大学木原生物学研究所 客員准教授)らの研究チームによる研究成果です。
植物研究には、作物増産技術の開発による食糧問題の解決だけでなく、循環型社会の構築に向けたバイオマスなどの再生可能資源の開発にも大きな期待が寄せられています。ミナトカモジグサは、食糧資源であるコムギやオオムギと同じイネ科の植物です。実験室で短期間に育成できることなどから麦類の研究やバイオマス研究に非常に有用だと考えられているモデル植物です。
今回、ミナトカモジグサの花や種子などの器官で発現する遺伝子について完全長cDNAライブラリを作成し、約1万種の完全長cDNAの完全配列を同定することができました。そして、同定した完全長cDNA配列情報を用いて、これまでに予測されていたミナトカモジグサの遺伝子構造の9,000カ所以上を修正しました。これまで別々であったコムギとオオムギのゲノム配列情報を統合し、ミナトカモジグサの情報も併せて相互比較できるデータベース(RIKEN Brachypodium Full-length cDNA Database (RBFLDB)(英語))を構築し、全ての研究者に利用できるよう公開しました。今回の成果は、麦類やバイオマス資源植物[2]を改良するために有用な遺伝子の探索やその機能の解明に貢献すると期待できます。また、ミナトカモジグサ完全長cDNAクローンは理研バイオリソースセンター(BRC)に寄託しており、10月10日よりBRCからクローンの入手が可能となります。
本研究は、理研社会知創成事業バイオマス工学研究プログラムの一環として行われました。成果は、米国の科学雑誌『PLOS ONE』オンライン版(10月9日付け:日本時間10月10日)に掲載されます。
背景
人口増加による食糧問題の解決のため、作物の増産技術の開発は世界の重要な課題です。コムギとオオムギの生産量は、トウモロコシ、イネに次いで多く(出典:2012年国連食料農業機関統計)主要な食糧資源として、病気や環境変動に強い麦品種の開発などにより生産性を維持・向上することが課題となっています。一方で、化石燃料の使用により生じた温室効果ガスの増加による地球の温暖化や気候変動が危惧され、バイオマスなどの再生可能資源を利用する循環型社会の構築に期待が寄せられています。バイオマス資源植物として、スイッチグラスやミスカンサス(ススキ)といったイネ科植物に有望なものがあり、その生産性や利用性を高めることが課題となっています。
ミナトカモジグサは、コムギやオオムギと同じイネ科の植物です(図1)。ゲノムサイズが272メガ塩基対と高等植物の中では比較的小さく、簡単に遺伝的性質を変化させることができ、また実験室内で育成可能など、モデル実験植物としての特徴を備えています。そのため、近縁であるコムギやオオムギなどの麦類にとっては品種改良に有用遺伝子の探索を推進するためのモデル実験植物として、またイネ科のバイオマス資源植物にとっては、バイオマス生産性や利用性を向上するための有用遺伝子の探索を推進するためのモデル実験植物として注目されています。
完全長cDNAは、メッセンジャーRNA(mRNA)の完全なコピーで、タンパク質の翻訳に必要な情報、つまり遺伝子と同じ情報をすべて備えています。遺伝子の機能情報がすべて保持されているため、有用遺伝子の探索や遺伝子機能を解明する上で必要不可欠な基盤情報となっています。ミナトカモジグサの完全長cDNAが大規模に解析され、その情報がデータベースとして公開されることは、麦類とバイオマス資源植物の双方の研究者から期待されていました。
研究手法と成果
研究チームは、様々な器官や時期、環境の変化応じて発現する遺伝子を網羅的に解析し、ミナトカモジグサの完全長cDNAライブラリを作製するために、乾燥条件や塩ストレス条件などの環境ストレス処理したミナトカモジグサの花や種子などの器官から合計21種類の試料を準備しました。まず、集めた試料からmRNAを取り出し、完全長cDNAライブラリを作製し、そこから約4万種の完全長cDNAを単離しました。約4万種の完全長cDNAの両端部分の塩基配列を解読し、EST[3]の約8万種が得ました。次に、ESTを解析し、約1万6,000種完全長cDNAの完全配列を決定しました。得られた完全長cDNA配列の重複を除いた結果、約1万種のミナトカモジグサの完全長cDNAを同定することができました。
完全長cDNAとゲノム配列を比べることで、ゲノム上の遺伝子領域を正確に定めることができます。今回の結果と従来のミナトカモジグサのゲノム情報と比べたところ、ミナトカモジグサ遺伝子領域の構造を9,000カ所以上修正することができ、より正確な遺伝子領域情報となりました(図2)。また、今回修正した遺伝子領域の構造情報を使うと、遺伝子配列の中からmRNAが合成される位置も正確に知ることができるため、その直前に位置する合成の制御領域も調べることが可能です。
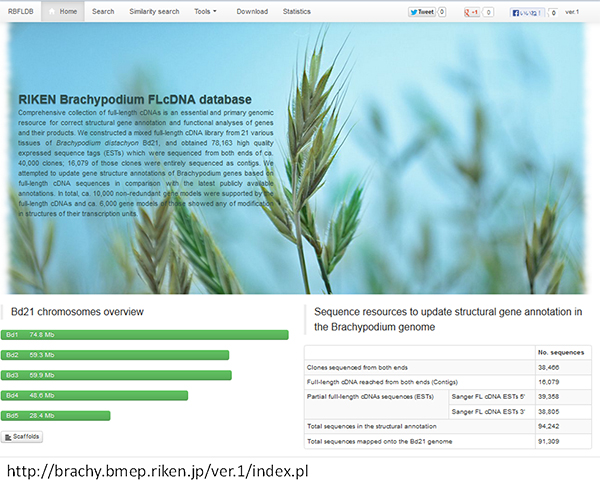
コムギやオオムギのゲノム配列とミナトカモジグサのゲノム配列が相互比較できるようになることで、ミナトカモジグサでの知見をすばやくコムギなどの麦類の研究に活用することができるようになります。そこで、ミナトカモジグサとコムギとオオムギの完全長cDNAやゲノム配列情報を統合し、一体的に閲覧できるデータベース(RBFLDB(英語))を構築し、全ての研究者に利用可能な形でインターネット上に公開しました(図3)。また、ミナトカモジグサ完全長cDNAクローンは理研バイオリソースセンター(BRC)に寄託しており、全てのクローンはBRCから入手可能となる予定です。
今後の期待
今回のミナトカモジグサ完全長cDNAの大規模収集により、世界のミナトカモジグサ研究や、近縁の麦類や同じイネ科植物のバイオマス植物の研究に大きく貢献する成果といえます。また、今回、麦類のゲノム情報と統合したことで、コムギ、オオムギ、ミナトカモジグサの知見を相互に見比べながら、効率よく有用遺伝子の探索を進めることができるため、病気や環境変動に強い麦品種の開発につながります。
原論文情報
- Keiichi Mochida, Yukiko Uehara-Yamaguchi, Fuminori Takahashi, Takuhiro Yoshida, Tetsuya Sakurai and Kazuo Shinozaki. Large-scale collection and analysis of full-length cDNAs from Brachypodium distachyon and integration with Pooideae sequence resources. Plos One 10.1371/journal.pone.0075265
発表者
理化学研究所
環境資源科学研究センター バイオマス工学連携部門 バイオマス研究基盤チーム
チームリーダー 篠崎 一雄 (しのざき かずお)
副チームリーダー 持田 恵一 (もちだ けいいち)
お問い合わせ先
環境資源科学研究推進室
Tel: 045-503-9471 / Fax: 045-503-9113
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.完全長cDNA
cDNAとは、ゲノムDNAのうちタンパク質をコードする配列のみの遺伝情報物質であるmRNA(メッセンジャーRNA)を鋳型にして作られたDNAのこと。cDNAはmRNAから作られるが、その際mRNAの全ての領域をカバーできずに不完全なものになることが多い。完全長cDNAは、mRNAの全領域をカバーするように作られているため、その配列情報からは遺伝子の完全な構造がわかり、そのクローンを翻訳してタンパク質を合成したり、遺伝子機能を細かく調べたりすることができる。 - 2.バイオマス資源植物
バイオマス資源として利用される植物。イネ科植物には、トウモロコシやサトウキビはバイオ燃料やバイオプラスチックの原料となっている。食料との競合をさけるために、これまで雑草として扱われていた大型の草であるミスカンサス(ススキ)やスイッチグラスなどをバイオマス用に利用しやすくする研究が進められている。バイオマス植物は、元来大型であるため実験室内で栽培しにくく、イネ科のバイオマス植物と細胞壁の構造が似ているミナトカモジグサは、バイオマス植物研究のスピードアップにも役立つと期待されている。 - 3.EST
Expressed Sequence Tagの略。cDNAライブラリからランダムに選んだクローンの両側末端部分から数百塩基程度の部分配列を決定したもの。解読した配列に相当する領域がゲノムDNAからmRNAに合成されていることがわかり、その遺伝子が実際に発現していることの証拠が得られる。

図1 ミナトカモジグサ
左上と左中央は茎、左下は根の断面の光学顕微鏡写真。中央は全体。右上は幼穂、右中央は葉、右下は茎の電子顕微鏡写真。学名はBrachypodium distachyonでブラキポディウムとも呼ばれる。コムギやオオムギと同じイネ科のイチゴツナギ亜科。ゲノムサイズが272メガ塩基対と高等植物の中では比較的小さく、遺伝子操作をして簡単に遺伝的性質を変化させることができ、また実験室で短期間に育成できることから麦類の研究やバイオマス研究に非常に有用だと考えられている。
写真:「バイオマス研究を担う草」

図2 完全長cDNAの情報をもとにミナトカモジグサの遺伝子構造を修正した例
今回得た完全長cDNAの配列情報とミナトカモジグサのゲノム配列と比べたところ、これまでに分かっているミナトカモジグサ遺伝子領域の構造について9,000カ所以上を修正することができた。