ポイント
- スパコンと世界最高性能の計算手法を組み合わせ、生物物理の基礎問題に決着
- 最新理論の誤りを指摘し、古典的理論を発展させた新規理論を提案
- iPS細胞など幹細胞の分化や免疫細胞の働きのより詳細な理解に役立つ
要旨
理化学研究所(理研、野依良治理事長)は、スーパーコンピュータによる大規模シミュレーションにより、細胞内分子間の情報伝達効率の上限を定義する基本理論をめぐる論争に終止符を打ちました。これは、理研生命システム研究センター(柳田敏雄センター長)生化学シミュレーション研究チームの海津一成基礎科学特別研究員、髙橋恒一チームリーダーと、オランダ原子分子国立研究所のピーター・レイン・テンウォルデ教授らの共同研究グループの成果です。
ヒトは多数の神経細胞を用いて「見」たり「考え」たりします。この時、神経細胞同士での情報の受け渡しの効率が重要です。同様に、細胞は分子を用いて外界を「感じ」、「考え」ます。この場合に、細胞内の分子の間でどれだけの情報を受け渡せるかの上限を定義するのが、1977年に米国ハーバード大学のハワード・バーグ教授らが提案した「バーグ=パーセル限界」と呼ばれる理論です。一方、米国プリンストン大学のウイリアム・ビアレック教授も2005年に同種の理論を発表しましたが、双方の理論の予測結果に矛盾があることが問題になっていました。しかし、最先端の計測機器を駆使してもこの理論の検証に十分な精度には達しませんでした。
共同研究グループは、髙橋チームリーダーらが開発した計算手法「改良グリーン関数反応動力学法:eGFRD[1]」を用いて、理研のスパコンRICC[2]上で、世界で初めてバーグ=パーセル限界の厳密な検証を行いました。その結果、より新しく精緻な理論とされてきたビアレックらの理論に誤りがあることが分かりました。一方、古典的なバーグらの理論が実際には厳密な検証にも耐えうることが証明され、この論争に終止符を打ちました。さらに共同研究グループは、シミュレーション実験の結果から、バーグ=パーセル限界と矛盾せず、かつビアレックらの理論のように精緻な新たな理論を導き出しました。
本研究成果は、iPS細胞(人工多能性幹細胞)などの幹細胞が、体内の成長因子をどの程度正確に検知し特定の種類の細胞への分化を決断するのか、免疫細胞がどのように異物を捕捉するのか-などをより詳細に理解する上で大きな役割を果たすと期待できます。また、細胞が行っている少数の分子を効率的に用いた情報処理を仮に現代の電子計算機で置き換えたとすると、消費電力は膨大なものになるため、その動作原理は将来超低消費電力の情報処理システム実現への一歩となる可能性があります。
本研究成果は、米国生物物理学会誌『Biophysical Journal』(2月19日号)に掲載されるに先立ち、オンライン(2月18日付け:日本時間2月18日)に掲載されます。
背景
ヒトは、どのように外界を「感じ」、物事を「考え」ているでしょうか?例えば、私たちは、眼球内の網膜にある神経細胞をセンサーとして物を見ています。また、物事を考える時には、脳の中にある神経細胞の間で情報の受け渡しが繰り返されています。これと同様に、細胞は分子を用いて外界を「感じ」、「考え」ます。細胞の「目」であり、また「神経細胞」にあたるのが、細胞が持つタンパク質分子です。細胞は、細胞膜上にある「受容体」と呼ばれる分子を用いて外界の環境を「感じ」ています。また、細胞内の分子の間で何段階にもわたって情報の受け渡しを繰り返すことで「考え」、どの遺伝子の発現をオンにしてどの遺伝子の発現をオフにするか、また幹細胞がどんな種類の細胞に分化するのかなどの意思決定を行っています。
分子の間でどのように情報が受け渡されるのか?その疑問に対し、1977年に米国ハーバード大学のハワード・バーグ教授らが、細胞内の分子の間でどれだけの情報を受け渡せるかの上限を定義する「バーグ=パーセル限界」の理論を提案しました。細胞の中や表面では分子は互いに結合と乖離(かいり)を繰り返し、伝言ゲームのように分子から分子へと情報を伝えます。例えば、細胞が周囲の栄養分子や細胞分化のきっかけとなるシグナル分子などの濃度を知りたい場合を考えます。これらの分子は「リガンド」と総称されます。細胞膜上に多数ある受容体は、リガンドと結合・乖離を繰り返しています。細胞は「受容体の占有率」、つまり、受容体分子のうち何割くらいがリガンドと結合しているかを指標にしてリガンドの濃度を見積ります。しかし、限られた時間内で状況を知ろうとすれば、実際のリガンドの濃度と受容体の占有率との関係には食い違い(誤差)が出ます。この誤差が大きければ、細胞は周囲の環境に対して誤った行動しかとれなくなります。与えられた時間内で細胞や分子がこの誤差をどこまで小さくできるのかの上限を表すのが、「バーグ=パーセル限界」です。
長年、バーグ=パーセル限界は「直感的に定義されたものであり、数理的には厳密な裏付けを持たない」と考えられてきました。2005年に米国プリンストン大学のウイリアム・ビアレック教授らは、現代的な統計物理学を駆使したより精緻な理論を提唱しましたが、双方の理論が予測する結果に矛盾があることが問題となっていました。どちらの理論が正しいのかを直接的に検証するためには、実験で分子と分子の結合と乖離を精密に計測する必要があります。しかし、この検証には時間スケールで1マイクロ秒(100万分の1秒)以下、空間スケールで数ナノメートル(1ナノメートルは10億分の1メートル)以下という精密さで分子1つひとつの動きを追う必要があり、レーザー顕微鏡などの最先端機器を用いても不可能でした。そこで共同研究グループは、非常に精密なコンピュータ・シミュレーションを用いてどちらの理論が正しいのか検証しました。
研究手法と成果
共同研究グループは、髙橋チームリーダーらが開発した世界最高性能の計算手法「eGFRD」を用い、理研のスパコンRICC上で大規模なシミュレーション実験を行いバーグ=パーセル限界の理論を直接的に検証しました。
具体的には、センサー分子(受容体など)とその周囲の分子(リガンドなど)1つひとつの振る舞いを1マイクロ秒以下、かつ数ナノメートル以下の高精度でRICC上に再現しました。次にセンサー分子とその周囲の分子との結合と乖離の状態を観測して得られた大規模な時系列データを解析し、分子濃度の見積りのあいまいさ(情報伝達の精度)を算出しました(図1)。このような計算をさまざまな分子の濃度や占有率に対して繰り返し行うことで、その結果をバーグ=パーセル限界の理論とビアレックらの理論と比較しました。
ビアレックらの理論は受容体分子1つひとつの結合・乖離状態を考慮した「1分子粒度」のモデルであるのに対し、バーグらの理論は個々の分子の振る舞いを考慮しない直感的なモデルです。それにも関わらず、検証結果は現代的なビアレックらの理論よりも古典的なバーグ=パーセル限界の理論の正しさを支持するものでした(図2)。
さらに共同研究グループは、シミュレーション実験による観測結果をもとにバーグ=パーセル限界の古典的な理論と矛盾せず、かつビアレックらのように1分子粒度での分子の結合・乖離まで考慮した、より精緻な理論を新たに導き出しました(図3)。
今後の期待
iPS細胞(人工多能性幹細胞)などの幹細胞は、周囲の成長因子により自らが筋肉や腎臓など、どんな種類の細胞に分化するのかを決めますが、この時、細胞は成長因子の濃度が非常に薄くても検知できることが知られています。バーグ=パーセル限界の理論はこのような細胞が持つバイオセンサーとしての感度の見積りに用いられてきました。本研究により細胞の分化や免疫を含むあらゆる細胞機能の理解の基盤がより強固になったと言えます。
細胞は1ピコリットル(1兆分の1リットル)と大変小さく、その中身には1種類の分子が数えられるほどの少数しか存在しないこともまれではありません(少数性問題:参考:少数性新学術領域)。また、細胞は1ピコワット(1兆分の1ワット)という非常に少ない消費エネルギーで動いています。従って、細胞内の少数分子の間での情報の受け渡しの多くはバーグ=パーセル限界の付近で非常に効率的に行われていると考えられます。この効率の上限を詳しく研究することは細胞システムをより深く理解する上でも重要な基礎であるとともに、将来的には超消費電力の情報処理システムの開発に結びつく可能性があります。
共同研究グループは、今回用いたシミュレーション手法のeGFRDよりさらに高度な手法の開発に取り組んでいます。これらの計算手法は理研生命システム研究センターが開発中の次世代細胞シミュレーター「E-Cell 4[3]」上に搭載し、オープンソースソフトウエアとして世界に向けて公開する予定です。また、理研が運用するスーパーコンピュータ「京」での利用も目指しています。
原論文情報
- Kazunari Kaizu, Wiet de Ronde, Joris Paijmans, Koichi Takahashi, Filipe Tostevin, and Pieter Rein ten Wolde,
“The Berg-Purcell limit revisited”,
Biophysical Journal, 2014, 10.1016/j.bpj.2013.12.030
発表者
理化学研究所
生命システム研究センター 生命モデリングコア 生化学シミュレーション研究チーム
チームリーダー 高橋 恒一(たかはし こういち)
お問い合わせ先
生命システム研究センター
広報担当 川野 武弘(かわの たけひろ)
Tel: 06-6155-0113 / Fax: 06-6155-0112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.改良グリーン関数反応動力学法:eGFRD
eGFRD はthe enhanced Greens Function Reaction Dynamicsの略。細胞内の分子1つひとつの運動までを再現した細胞シミュレーションを可能にするために開発した粒子反応拡散系のシミュレーション手法。細胞内の分子は多数存在する多体系であり、基礎方程式を直接解くことは不可能だが、1体あるいは2体問題であれば解(グリーン関数)が存在する。この性質を利用し、eGFRD法は多体問題を複数の1体あるいは2体問題に分解し、独立して解くことができるため従来よりも飛躍的に高性能の計算が可能になる。
注)2010年1月29日プレスリリース: 分子1つ1つの運動まで再現する細胞シミュレーション法を開発 - 2.RICC
RICCはRIKEN Integrated Cluster of Clustersの略。理研情報基盤センターが運用する科学技術計算用のスーパーコンピュータ。理論性能は約100テラフロップス(1テラフロップスは1秒間に1012回の浮動小数点演算を行う能力)であり、スーパーコンピュータ「京」の約100分の1。 - 3.E-Cell 4
E-Cell Systemとは、1996年に慶應義塾大学で発足したE-Cellプロジェクトにより開発された国産の細胞シミュレーター。複数の計算法や時間スケールなどが混在しても効率的にシミュレーションできるなどの特徴を持つ。E-Cell System Version 4 (E-Cell 4) は、理研が開発中の次世代版のシステムであり、本研究で利用したeGFRD法をはじめとして、分子1つひとつの細かさ(1分子粒度)で細胞内生化学反応経路の機能発現をシミュレーションできる。
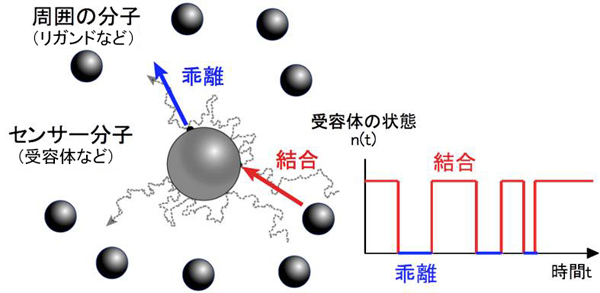
図1 シミュレーションで用いたモデル
センサー分子(受容体など)とその周囲の多数の分子1つひとつの動きと結合・乖離反応を計算機上で正確に再現し、受容体の状態の時系列を記録した。シミュレーションでは約1千億回の結合・乖離反応が記録され、データの総量は100ギガバイトに及んだ。この時系列を数値的に解析することでバーグ=パーセル限界の検証を行った。
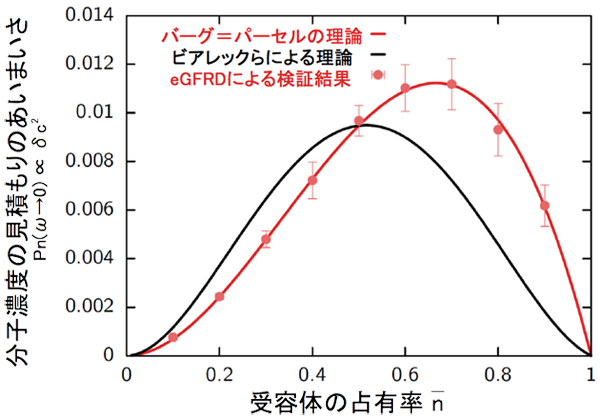
図2 バーグ=パーセル限界とビアレックらの理論の検証結果
受容体の占有率(横軸)を変化させたとき、その受容体による分子濃度の見積りのあいまいさ(縦軸)がどのように推移するかは、バーグ=パーセル限界の理論とビアレックらの理論で予測が異なる。これをシミュレーションによる検証で比較した結果、古典的なバーグ=パーセル限界の理論がeGFRD法によるシミュレーションとよく一致していることが分かる。
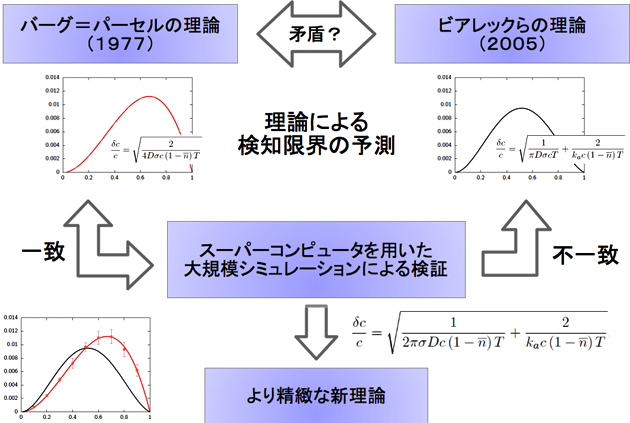
図3 本研究の概要
実証の困難さから議論となってきたバーグ=パーセル限界の理論をスーパーコンピュータを用いて検証し、分子1つひとつの動きまで考慮したより精緻な新理論を提案した。
