2014年9月22日
独立行政法人理化学研究所
株式会社ダナフォーム
ヒトの大部分の一塩基多型(SNP)を検出するツールをカタログ化
-SNPなどを利用した個別化医療の実現へ大きな一歩-
ポイント
- 約6,000万箇所のヒトSNPを検出するプライマー/プローブ配列を計算
- 大規模PCクラスタを使った並列処理によりわずか13日で必要な計算を終了
- SNPに基づく個別化医療分野での遺伝子解析技術の利便性が拡充
要旨
理化学研究所(理研、野依良治理事長)と理研ベンチャー[1]の株式会社ダナフォーム(三谷康正代表取締役社長)は、共同開発したDNA増幅法「PEM(PCR Eprobe Melting)法[2]」を使い、既知の約6,000万箇所の一塩基多型(SNP; Single Nucleotide Polymorphism)[3]を検出するための人工核酸[4](プライマーとプローブ)の最適な配列を全て計算し、このうち、約4,000万箇所をカタログ化しました。これにより、研究者がプライマー/プローブ配列の検討することなく、すぐに研究に取り入れられるようになります。これは、理研情報基盤センター(姫野龍太郎センター長)計算工学応用開発ユニットの須永泰弘センター研究員、理研予防医療・診断技術開発プログラム(林崎良英プログラムディレクター)と、ダナフォームの木村恭将チームリーダー(理研ライフサイエンス技術基盤研究センター客員研究員)との共同研究グループによる成果です。
SNPなどの遺伝子変異[5]に基づく個別化医療は、遺伝情報を活用した医療サービスの中で最も期待されている分野です。そのためにはSNPを高い精度で検出することが必要です。共同研究グループは、従来法では難しいSNPが密集した領域の遺伝子変異の有無も高い精度で簡便に検出できるPEM法を開発しました。
PEM法にはSNPを検出するためのプライマーとプローブのセットが必要です。そこで、共同研究グループは、PEM法に最適化した設計ソフトウェア「Edesign(イーデザイン)」を使って各SNPを検出するためのプライマーおよびプローブの最適な配列を計算し、セットを選出しました。そして、配列固有の理由でプライマーやプローブが設計できないSNPを除いた約4,000万箇所をカタログ化しました。
今回のカタログ化により研究が効率化するため、SNPに基づく個別化医療に向け研究の加速が期待できます。本研究成果は、9月22日、ダナフォームのウェブサイトで公開します。
背景
一塩基多型(SNP)などの遺伝子変異に基づく個別化医療は、遺伝情報を活用した医療サービスの中で最も期待されている分野です。現在では、個人のゲノム解析データの蓄積とともに、ヒトのゲノム上に疾病などとの関連が分かっているもの、未知のものを含め約6,000万箇所のSNPがあることが知られており、ヒトのSNPはほぼ出尽くしたと考えられています。今後は、これら既知のSNP情報をもとに、疾病関連SNPを同定する研究や診断に簡単に利用できるように基盤を整備することが重要です。
医学利用の観点からは、SNPを高い精度で検出することが必要です。これを可能にしたのが、理研予防医療・診断技術開発プログラムと理研ベンチャーの株式会社ダナフォームが開発したPEM(PCR Eprobe Melting)法(図1)です。PEM法は、従来法では難しいSNPが密集した領域の遺伝子変異の有無も高い精度で簡便に検出可能です。PEM法に必要な短い人工核酸(プライマーとプローブ)の設計は、比較的容易ですが、検出しようとするSNPごとにプライマーとプローブの配列を検討する必要があります。
そこで、共同研究グループは既知のほぼ全てのSNPを検出するために最適なプライマーとプローブのセットをあらかじめ計算して、それをカタログ化することに取り組みました。
研究手法と成果
共同研究グループは、PEM法に最適化した設計ソフトウェア「Edesign(イーデザイン)」を使って各SNPを検出するためのプライマーおよびプローブの最適な配列を計算し、セットを選出しました。この計算は、約6,000万箇所という大量の配列の計算になるため、一般的なデスクトップ型PCでは計算に8年程度かかると見積もられました。今回、理研の大規模PCクラスタでデータ転送などの処理を最適化して並列処理を行うことで、13日で計算を終了しました。その結果、他のSNPが非常に近くにあるなど、配列固有の理由でプライマーやプローブが設計できないSNPを除いた約4,000万箇所についてプライマーとプローブのセットの配列情報が得られました。得られた結果は1.2テラバイトのデータ量となりました。
研究で得たプライマーとプローブ約4,000万セットの配列情報は、ダナフォームのウェブサイトにて無償で公開しました(図2)。また、共同研究グループでは、PEM法のプライマーとプローブの設計を行った設計ソフトウェアのEdesignも合わせて公開しました。
今後の期待
PEM法のような遺伝子増幅・検出技術は個別化医療に資する遺伝子解析技術の中でも中核的な技術です。今回、既知のヒトSNPの多くの部分をカバーするプローブとプライマーのセットの配列カタログが整備されたことで、PEM法の利便性が大幅に向上し、SNPに基づく個別化医療の実現に大きな前進をもたらすと期待できます。
発表者
理化学研究所
情報基盤センター 計算工学応用開発ユニット
センター研究員 須永 泰弘(すなが やすひろ)
株式会社ダナフォーム
事業部インフォマティクスチーム
チームリーダー 木村 恭将(きむら やすまさ)
独立行政法人理化学研究所
予防医療・診断技術開発プログラム
連携促進コーディネーター 西川 実希(にしかわ みき)
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.理研ベンチャー
理研の研究成果を中核技術として起業し、一定の要件を満たすことで理研から認定を受けた企業。理研ベンチャーでは、最先端で活躍する理研の研究者が自然科学における研究テーマを追求する過程で考案した「新しい知見や技術」を日常の暮らしや産業技術に役立てることを目指し、研究成果の迅速な実用化と普及に取り組んでいる。 - 2.PEM(PCR Eprobe Melting)法
PCR法は、DNAを増幅する技術。PEM法では、この反応にEprobeと呼ばれる蛍光プローブを共存させ、対象(テンプレート)の配列とプローブ配列の一致度によるテンプレートとプローブの乖離(かいり)温度の差から変異の有無を検出する(融解曲線法)。 - 3.一塩基多型(SNP; Single Nucleotide Polymorphism)
ヒトゲノムは、約30億塩基対からなるとされているが、一人ひとりを比較するとその塩基配列には違いがある。このうち、集団内で1%以上の頻度で認められる塩基配列の違いを多型と呼ぶ。遺伝子多型で最も数が多いのは一塩基の違いであるSNPである。遺伝子多型による塩基配列の違いは、遺伝子産物であるタンパク質の量的または質的変化を引き起こし、病気へのかかりやすさや医薬品への応答性、副作用の強さなどに影響を及ぼす。 - 4.人工核酸
PEM法で用いる人工核酸は、合成した短い核酸に蛍光色素を結合させたもので、SNPの検出のために用いる。 - 5.SNPなどの遺伝子変異
今回の計算では、SNPのほか、複数塩基の変異、小規模な配列の挿入や欠失(in/del)、リピートといった遺伝子変異も含まれている。
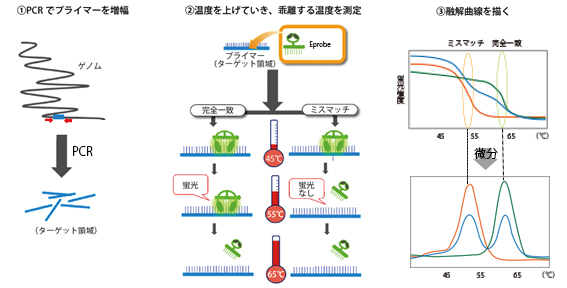
図1 PEM法の概要
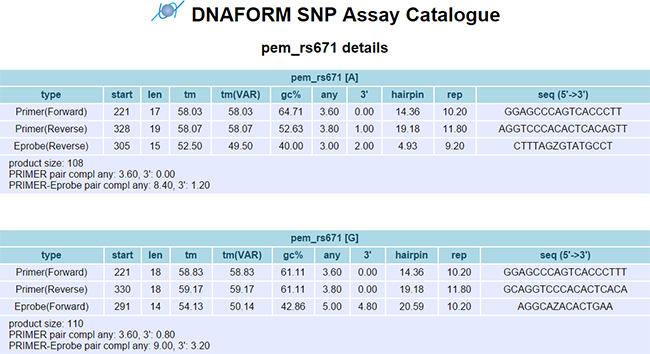
図2 公開したデータのイメージ
