2019年9月13日
理化学研究所
奈良先端科学技術大学院大学
病害寄生雑草ストライガの全ゲノム解読に成功
-アフリカを襲う農業被害の撲滅に光-
理化学研究所(理研)環境資源科学研究センター植物免疫研究グループの白須賢グループディレクター、奈良先端科学技術大学院大学先端科学技術研究科バイオサイエンス領域植物共生学研究室の吉田聡子特任准教授らの国際共同研究グループは、アフリカを襲う病害寄生雑草であるストライガ(Striga)の全ゲノム解読に成功しました。
本研究成果は、ストライガの進化および寄生メカニズムの理解、ストライガ撲滅に向けた除去剤開発などに貢献すると期待できます。
ストライガは主要な穀物に寄生し、収穫量を大幅に減らす有害植物であり、特にアフリカで深刻な被害をもたらしています。この問題の根本的な解決に向けてストライガの寄生メカニズムを理解するには、全ゲノム情報が必要でした。
今回、国際共同研究グループは、1950年代に米国に侵入したストライガの系統からゲノムDNAを抽出し、全ゲノムシークエンス解析[1]、トランスクリプトーム[2]解析などを行った結果、タンパク質をコードする遺伝子34,577個を同定しました。また、適応進化の過程において、ストライガは全ゲノム2倍化[3]を2回起こすことで寄生に必要な遺伝子を獲得したこと、ストリゴラトン[4]受容体ファミリーが著しく増えたことでさまざまな宿主を獲得したことが分かりました。さらに、ストライガゲノムに宿主から「遺伝子の水平伝播[5]」が起きた証拠も見つけました。
本研究は、米国の科学雑誌『Current Biology』の掲載に先立ち、オンライン版(9月12日付け:日本時間9月13日)に掲載されます。

図 ケニアに生息するストライガ(Striga asiatica)
背景
ストライガ(Striga)は、双子葉植物であるハマウツボ科に分類される「寄生植物」です。ストライガは、主要な穀物に寄生して水分や養分を横取りし、その収穫量を大幅に減らします。特にアフリカでの農業被害は深刻であり、“魔女の雑草”とも呼ばれています。特に、ソルガム[6]、キビ、トウモロコシ、陸稲、サトウキビなどの根に寄生し、その感染領域は、主にアフリカの半乾燥地域を中心に日本の面積を超える約40万km2に及び、年間被害額は約1000億円にものぼると推定されています注1)。また、ストライガは1950年代に米国に侵入し、大きな被害を出したこともあります。
ストライガは、独自の寄生システムを適応進化させていることから、撲滅が非常に困難です。例えば、ストライガは1個体で種子を10万個以上も作るうえ、それらは0.2mmほどと非常に小さいため、風によって遠くまで飛散します。さらに、飛散した種子は、宿主となる植物が現れるまで数十年間も休眠することができます。宿主が現れると、宿主の根から放出される「ストリゴラクトン」という植物ホルモンを感じて発芽します。発芽すると、ストライガの根は宿主の根に向かって伸びていき、根の先端に「吸器」という器官を発達させます。吸器は宿主の根に侵入し、宿主の維管束系[7]を連結することで、水と栄養素を奪うようになります。その際、宿主からの遺伝物質もストライガに移行することが知られており、これを「遺伝子の水平伝播」と呼びます。
ストライガによる農業被害を撲滅する手段として、寄生メカニズムに基づいた除草剤の開発が挙げられます。しかし、寄生の分子メカニズムを理解するには、遺伝子の情報とその機能解析が必要であり、それに必要な全ゲノム情報の解読が求められていました。
注1)Rodenburg J,Demont M, Zwart SJ, Bastiaans L (2016) Parasitic weed incidence and related economic losses in rice in Africa. Agric Ecosyst Environ 235:306–317.
研究手法と成果
国際共同研究グループは、ストライガ(Striga asiatica)の系統からゲノムDNAを抽出し、次世代シークエンサー[8]を用いて、全ゲノムシークエンス解析とアセンブリー[9]を行いました。さらに、トランスクリプトーム解析を行い、ゲノムアノテーション[10]を完成しました。その結果、タンパク質をコードする遺伝子34,577個を同定しました。
詳しい解析により、適応進化の過程でストライガゲノムは、全ゲノム2倍化を2回起こしていることが分かりました(図1)。これにより適応の自由度が増し、寄生に必要な遺伝子を獲得できたと考えられます。さらに、①寄生器官である吸器の獲得、②宿主による機能(水や栄養)により不必要になった遺伝子の削減が起きていることが分かりました。また、宿主と寄生の関係性と特異性を規定する遺伝子の獲得に関わる遺伝子群も同定されました。
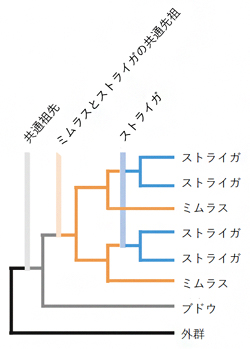
図1 ストライガの系統進化の間に発生した全ゲノム2倍化現象を示す系統樹
共通祖先は多くの双子葉植物によって共有されている全ゲノム3倍化であり、寄生植物のストライガと非寄生植物のミムラス(ハエドクソウ科ミゾホオズキ属)は全ゲノム2倍化(ゲノム重複)を共有しており(橙色の線)、さらにストライガは独立した全ゲノム2倍化を経験している(青の線)。
ストライガの種子は、宿主由来のストリゴラクトンに応答して発芽し、宿主根に侵入するための特殊な侵入構造である吸器を発達させます。ただし、ストリゴラクトンの種類は宿主によって異なるため、それらを感知するにはそれぞれに対応した受容体が必要です。今回のゲノム解析から、ストリゴラクトン受容体ファミリーの数は、非寄生植物のシロイヌナズナでは1個しかないのに対し、ストライガでは21個にまで増えていたことが分かりました(図2)。これにより、ストライガはさまざまな宿主を得ることができたと考えられます。また、非寄生植物において側根形成に関与する遺伝子が、ストライガにおける吸器発生中に誘導されることも判明しました。これは、吸器の進化において、部分的にこの側根形成のシステムが使われるようになったことを示しています。
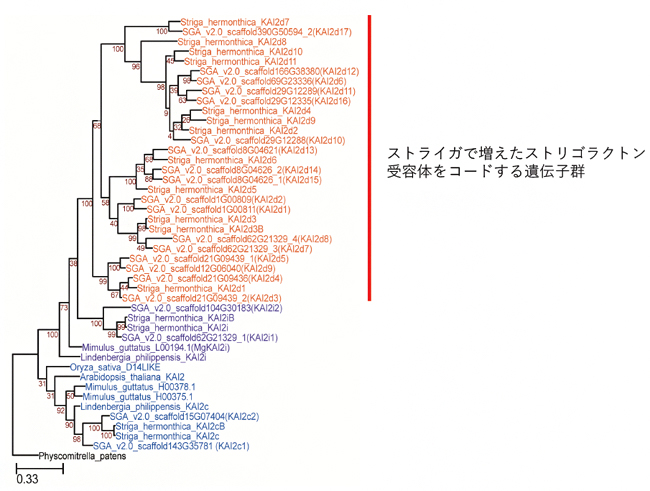
図2 ストライガと非寄生生物におけるストリゴラクトン受容体群の系統樹
青字:ほとんどの植物に保存されているストリゴラクトン受容体(保存型)、紫字:ストライガと、ストライガに近縁の非寄生植物ミムラス(Mimulus Lindenbergia)に共通するストリゴラクトン受容体(中間型)、赤字:ストライガ(Striga hermonthicaおよびStriga asiatica(SGA))に保存されているストリゴラクトン受容体(特異型)を示している。ストライガでは、ストリゴラクトン受容体は21個あったのに対し(全29個のうち8個は同種)、非寄生植物のシロイヌナズナ(Arabidopsis thaliana)では1個だけである。
さらに、ストライガのゲノム上に宿主由来のレトロトランスポゾン[11]と宿主の遺伝子が発見されました(図3)。これは、宿主からストライガに遺伝子情報が水平移動した証拠です。このことから、逆に寄生植物が農場でどのように進化しているのかが議論できます。
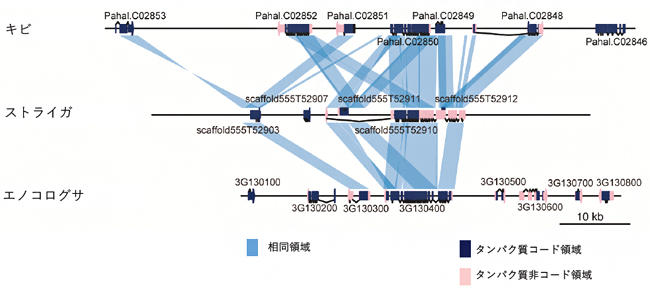
図3 ストライガが宿主から獲得した遺伝子群のゲノム領域の比較
ストライガと宿主植物のキビ、エノコログサのゲノム領域の比較。濃い青はタンパク質コード配列、ピンクはタンパク質非翻訳領域を示す。高い相同性(共通祖先)を示す領域は、スカイブルーの線で結ばれている。ストライガゲノムには、宿主由来の遺伝子が存在することが分かる。
今後の期待
本研究でストライガの生命設計図である全ゲノム配列が決定したことは、ストライガによる寄生のメカニズムの解明に向けた大きな前進です。また、寄生植物が農場でどのように進化しているのかについてもモニターできるようになりました。寄生植物の発芽誘導剤や阻害剤の探索など、新しい寄生植物防除の方法の開発につながり、ストライガによる農業被害の撲滅に貢献すると期待できます。
今回の研究成果は、国際連合が2016年に定めた17項目の「持続可能な開発目標 (SDGs) [12]」のうち「2.飢餓をゼロに」と「15.陸の豊かさも守ろう」に大きく貢献するものです。
補足説明
- 1.全ゲノムシークエンス解析
次世代シーケンサーを使って、生物の全ゲノム情報を解読し、配列の違いや変化を同定すること。 - 2.トランスクリプトーム
ある状況下における、転写産物の全体像。ここではmRNAを対象にして解析をしている。 - 3.全ゲノム2倍化
生物のゲノム全体が倍化する現象。全遺伝子数が倍加することで、適応の自由度が増し、生物の多様性が生み出されると考えられている。 - 4.ストリゴラクトン
植物ホルモンの一種で、寄生植物の発芽誘導だけではなく、枝分かれや葉の老化など植物のさまざまな発生段階に作用する。これまでの研究から、KAI2dタンパク質群がストリゴラクトンの受容体であることが分かっている。 - 5.遺伝子の水平伝播
親子関係の無い個体間、または他生物間で起こる遺伝子の取り込み。 - 6.ソルガム
熱帯アフリカ原産のイネ科の一年草。生産面積ではコムギ、イネ、トウモロコシ、オオムギに次いで世界第5位である。乾燥に強く、熱帯では主食として用いられる。 - 7.維管束系
導管や師管などからなり、水や養分の通り道や、植物体の機械的支持に働く複合組織。 - 8.次世代シークエンサー
細かく断片化された大量のDNA配列を同時並行的に解析し、塩基配列を高速に決定することができる解析装置。 - 9.アセンブリー
次世代シークエンサーによって解読されたDNA断片配列をつなげて対象ゲノム配列を復元するコンピュータ処理のこと。 - 10.ゲノムアノテーション
ゲノム配列から、ある特定の領域が遺伝子か、遺伝子でない(非コード領域)かなどの注釈を付けること。 - 11.レトロトランスポゾン
DNA→RNAへの転写と、RNA→DNAへの逆転写によって増殖する性質を持つ遺伝因子。ゲノム上のレトロトランスポゾンが転写されてRNAになり、さらに逆転写酵素によりDNAに戻ることでゲノム中を移動しコピー数を増やす。 - 12.持続可能な開発目標(SDGs)
2015年9月の国連サミットで採択された「持続可能な開発のための2030アジェンダ」にて記載された2016年から2030年までの国際目標。持続可能な世界を実現するための17のゴール、169のターゲットから構成され、発展途上国のみならず,先進国自身が取り組むユニバーサル(普遍的)なものであり、日本としても積極的に取り組んでいる(外務省ホームページから一部改変して転載)
国際共同研究グループ
理化学研究所
環境資源科学研究センター 植物免疫研究グループ
グループディレクター 白須 賢(しらす けん)
バイオリソース研究センター 植物-微生物共生研究開発チーム
チームリーダー 市橋 泰範(いちはし やすのり)
奈良先端科学技術大学院大学 先端科学技術研究科 バイオサイエンス領域
特任准教授 吉田 聡子(よしだ さとこ)
教授 出村 拓(でむら たく)
名古屋大学大学院 生命農学研究科
教授 榊原 均(さかきばら ひとし)
京都大学大学院 農学研究科
教授 山口 信次郎(やまぐち しんいちろう)
東京工業大学 地球生命研究所
助教 佐々木 結子(ささき ゆうこ)
宇都宮大学農学部
准教授 野村 崇人(のむら たかひと)
北海道大学 工学研究院 応用化学部門 生物工学分野
助教 堀 千明(ほり ちあき)
東京農工大学 グローバルイノベーション研究院
教授 笠原 博幸(かさはら ひろゆき)
明治大学 農学部 農芸化学科
専任講師 瀬戸 義哉(せと よしや)
ソウル大学(韓国)
教授 ドイル・チョイ(Doil Choi)
ペンシルベニア州立大学(米国)
教授 クラウド・デパンフィリス(Claudede Pamphilis)
ヘルシンキ大学(フィンランド)
教授 アラン・シュルマン(Alan Schulman)
カリフォルニア大学リバーサイド校(米国)
教授 デイビッド・ネルソン(David Nelson)
バージニア大学(米国)
教授 マイケル・ティムコ(Michael Timko)
トロント大学(カナダ)
助教 シェリー・ルンバ(Shelley Lumba)
国際家畜研究所(ケニヤ)
研究員 ムセンビ・ムトゥク(Musembi Mutuku)
ジョイントバイオエナジー研究所(米国)
ディレクター ジェニー・モーティマー(Jenny C.Mortimer)
研究支援
本研究は、文部科学省新学術領域研究(研究領域提案型)「植物の成長可塑性を支える環境認識と記憶の自立分散型統御システム(研究領域代表者:木下俊則)」の計画研究「寄生植物による維管束情報ハイジャック機構の解明(研究代表者:白須賢)」などによる支援を受けて行われました。
原論文情報
- Satoko Yoshida, Seungill Kim, Eric K. Wafula, Jaakko Tanskanen, Yong-Min Kim, Loren Honaas, Zhenzhen Yang, Thomas Spallek, Caitlin E. Conn, Yasunori Ichihashi, Kyeongchae Cheong, Songkui Cui, Joshua P. Der, Heidrun Gundlach, Yuannian Jiao, Chiaki Hori, Juliane K. Ishida, Hiroyuki Kasahara, Takatoshi Kiba, Myung-Shin Kim, Namjin Koo, Anuphon Laohavisit, Yong-Hwan Lee, Shelley Lumba, Peter McCourt, Jenny C. Mortimer, J. Musembi Mutuku, Takahito Nomura, Yuko Sasaki-Sekimoto, Yoshiya Seto, Yu Wang, Takanori Wakatake, Hitoshi Sakakibara, Taku Demura, Shinjiro Yamaguchi, Koichi Yoneyama, Ri-ichiroh Manabe, David C. Nelson, Alan H. Schulman, Michael P. Timko, Claude W. dePamphilis, Doil Choi, Ken Shirasu, "Genome sequence of Striga asiatica provides insight into the evolution of plant parasitism", Current Biology, 10.1016/j.cub.2019.07.086
発表者
理化学研究所
環境資源科学研究センター 植物免疫研究グループ
グループディレクター 白須 賢(しらす けん)
奈良先端科学技術大学院大学 先端科学技術研究科 バイオサイエンス領域 植物共生学研究室
特任准教授 吉田 聡子(よしだ さとこ)
 白須 賢
白須 賢
 吉田 聡子
吉田 聡子
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
奈良先端科学技術大学院大学 企画総務課 広報渉外係
Tel: 0743-72-5026 / Fax: 0743-72-5011
E-mail:s-kikaku [at] ad.naist.jp
