要旨
理化学研究所(理研)生命システム研究センター細胞動態計測研究グループの藤田恵介特別研究員、柳田敏雄グループディレクターらの研究チームは、転写分子がDNAに沿って動き、遺伝情報を読んで(転写して)いるところを1分子イメージング法で直接見ることに成功しました。さらに、複数の転写分子がDNAに沿って動くとき、高速道路の渋滞と同じような渋滞が起こり、その渋滞がひどくなったり緩和したりする“ゆらぎ”がDNAの転写頻度のゆらぎとなることを明らかにしました。このことは、転写分子の渋滞が細胞個性[1]を作り出していることを示唆しています。
遺伝的に同じでかつ環境が同じでも、細胞には個性が現れます。細胞個性は受精卵からさまざまな細胞に変化(分化)していく過程に必須であることが近年明らかになってきました。さらに、転写頻度のゆらぎは細胞個性を生むことで注目されていますが、なぜ転写頻度のゆらぎが起こるのかについてはまだよく分かっていません。
研究チームは、転写分子であるRNAポリメラーゼ(RNAP)がDNA上を動きながら遺伝情報を読み取り、それをmRNAに写し取る反応(転写)を細胞外で再現し、全反射蛍光顕微鏡[2]で観察しました。その結果、RNAPとDNAだけで再現された転写においても、細胞内と同じく転写頻度のゆらぎが現れたことから、このゆらぎはRNAPとDNAの分子が原因であると結論付けました。また、RNAPの1分子イメージング、原子間力顕微鏡[3]画像撮影、コンピューターを用いたRNAPの運動シミュレーションを行いました。以上の結果を総合して、「転写頻度のゆらぎはDNA上で起こるRNAPの“渋滞とその緩和”によって引き起こされること」が明らかになりました。
研究チームは、生命がこのような転写頻度のゆらぎをもつ背景には、細胞がより単純だった進化の初期から、転写頻度のゆらぎ、つまりそれによって生まれる細胞個性が環境変化への適応に有利だったことがあると考えています。
今後、本発見をもとに細胞内で起こるRNAPの渋滞とその緩和を調節することができれば、例えば、iPS細胞からさまざまな細胞を作るときに必要な細胞状態の予測と制御に役立つ可能性があると考えられます。
本成果は、国際科学雑誌『Nature Communications』(12月7日付け)に掲載されます。
背景
“生命の設計図(遺伝情報)”はDNAに塩基配列(A、T、G、Cの4文字の並び)として書かれていますが、細胞が実際に生命活動を行うには、その塩基配列の一部を読み出して機能的分子に変換(遺伝子発現)する必要があります。転写はその変換過程の最初の段階で、RNAポリメラーゼ(RNAP)と呼ばれるタンパク質がDNA上を動きながら遺伝情報を読み取ると同時に、そのコピーとなるメッセンジャーRNA(mRNA)を合成していく反応です。細胞内のmRNAの量は細胞状態と関係するため、mRNAの量の違いは、細胞状態の違いを生む可能性があります。
転写頻度のゆらぎ(mRNAの合成が一定頻度で起こらないこと)は、集団内の個々の細胞にあるmRNAの量に大きなばらつきを与えます。転写頻度のゆらぎは、同じDNAを持ち、同じ環境にいる細胞集団に個性を与え、受精卵からさまざまな細胞に分かれていく、発生や分化のような過程に必須であることが近年明らかになってきました注1)。また、転写頻度のゆらぎは大腸菌からヒト細胞までのさまざまな細胞で観察されており、生命に普遍的な現象であるといわれています注2)。
しかし、細胞の中では、分子の挙動を詳細に解析することができないため、転写頻度のゆらぎがどのような分子機構によって引き起こされるのかは、これまでよく分かっていませんでした。
注1)Eldar, A. & Elowitz, M.B. Functional roles for noise in genetic circuits. Nature 467, 167-173 (2010).
注2)Sanchez, A. & Golding, I. Genetic determinants and cellular constraints in noisy gene expression. Science 342, 1188-1193 (2013).
研究手法と成果
今回、研究チームは、細胞から転写に必要な分子(RNAPとDNA)を取り出し、全反射照明蛍光顕微鏡下で転写を再現しmRNAの合成頻度を計測しました。
具体的には、鋳型DNA(転写の際に塩基配列が読み取られるもととなる鎖)をガラス表面上に固定し、溶液中には蛍光色素(Cy3)で標識された短いDNA断片(DNAプローブ)を加えておきます。さらに、RNAPとmRNAの材料になる分子(リボヌクレオチド)を加えると、RNAPは鋳型DNA上で転写を始め、mRNAが合成されていきます。DNAプローブはmRNAと相補的に結合するため、mRNAが1分子合成されるごとにDNAプローブがそのmRNAに1分子結合します。
全反射照明蛍光顕微鏡での観察では、DNAプローブの蛍光はmRNAに結合したときのみ検出でき、mRNAを1分子ずつ高感度に検出できます。鋳型DNAは別の波長(色)の蛍光色素(Cy5)で標識されており、二つの蛍光色素(Cy3とCy5)の重なりによって鋳型DNA上でmRNAが合成されたことを確認できます(図1上)。
このような実験系を用いて、単位時間当たりに合成されたmRNAの分子数をカウントしました。その結果、mRNA合成頻度が時間経過とともに大きくゆらぐことを示しており、さまざまな細胞で報告されている転写頻度のゆらぎと同様の現象が起こることを確認できました(図1下)。
次に、研究チームは、RNAPがmRNA合成中に頻繁に停止することに着目し、「RNAPの停止によってDNA上でRNAPの“渋滞”が起こり、その結果mRNA合成頻度が大きくゆらぐ」という仮説を立てました。
まず、研究チームは実際にRNAPが頻繁に停止していることをRNAPの運動の1分子イメージング(図2)によって確認しました。この実験系では、鋳型DNAの両端をガラス表面上に固定しています。その鋳型DNA上でRNAPは転写を開始します。RNAPはQdot655で標識されており全反射蛍光顕微鏡によって動きを観察できます。さらに、mRNA合成の1分子イメージング(図1)と同様にDNAプローブを加えることで、RNAPの動きと同時にmRNAの合成を検出しました。図2下のデータは、RNAPの運動とともにmRNAが合成され、転写の途中でRNAPが長時間停止する様子が観察されています。
さらに、この頻繁に起こるRNAPの停止がRNAPの渋滞を引き起こしていることは、原子間力顕微鏡画像によっても確認されました(図3)。
さらに、RNAPの運動をコンピューター上でシミュレーションすることで、仮説が正しいことを定量的に証明しようと試みました。図4は、研究チームが用いたシミュレーションモデルを模式的に説明しています。このモデルでは、緑色の丸はRNAP、一つの箱はDNAの1塩基対を表しています。RNAPは左端から箱を進んでいきますが、一定の確率で停止状態(赤色)になります。RNAPは、実際の実験と同じく、一定の確率で左端に現れます(転写開始)。そのため、DNAの途中でRNAPが停止状態になることは、後ろから続くRNAPの妨げになり、分子渋滞が起こります。停止状態のRNAPは後ろからくるRNAPによって押し出されることが報告されており注3)、このモデルにおいても後ろから押されている停止状態のRNAPは一定の確率で、また動き出すと仮定しています。以上のシミュレーションモデルの中で、RNAPはDNA上で渋滞と緩和を繰り返します。
研究チームはこのモデルを用いて、コンピューター上で転写頻度のゆらぎを再現することに成功し、転写頻度のゆらぎはDNA上で起こるRNAPの渋滞と緩和によって引き起こされるという仮説が正しいことを定量的にも証明しました(図5)。
注3)Epshtein, V. & Nudler, E. Cooperation between RNA polymerase molecules in transcription elongation. Science 300, 801-805 (2003).
今後の期待
これまで、転写頻度のゆらぎはさまざまな制御分子が関わって生まれると考えられていました。その意味で、今回研究チームが明らかにした、RNAPの渋滞と緩和で転写頻度のゆらぎが引き起こされるというモデルは常識を覆すものです。では、なぜこのような単純な分子機構で細胞個性が引き起こされるのでしょうか。RNAPは転写という生命に必須の現象を担うタンパク質で、現在のすべての生物に存在し、進化的に同じ起源を持っているといわれています注4)。また転写頻度のゆらぎ、その結果起こる細胞個性は生物に普遍的な現象で、微生物にとっても細胞個性が生存に有利であることが分かっています注5)。
以上のことから、研究チームは、転写頻度のゆらぎが生存に有利だったため、原始的な細胞が今回発見した単純な分子機構を進化的に獲得し、それが現代の生物に引き継がれているのではないかと考えています。また今後、本成果をもとに細胞内で起こるRNAPの渋滞とその緩和を調節することができれば、細胞個性、さらには分化の予測と制御に役立つ可能性があると考えられます。
注4)Werner, F. & Grohmann, D. Evolution of multisubunit RNA polymerases in the three domains of life. Nat Rev Microbiol 9, 85-98 (2011).
注5)Wakamoto, Y. et al. Dynamic persistence of antibiotic-stressed mycobacteria. Science 339, 91-95 (2013).
原論文情報
- Keisuke Fujita, Mitsuhiro Iwaki, Toshio Yanagida, "Transcriptional bursting is intrinsically caused by interplay between RNA polymerases on DNA", Nature Communications, doi: 10.1038/NCOMMS13788
発表者
理化学研究所
生命システム研究センター 細胞動態計測コア 細胞動態計測研究グループ
特別研究員 藤田 恵介(ふじた けいすけ)
グループディレクター 柳田 敏雄(やなぎだ としお)
 藤田 恵介
藤田 恵介
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.細胞個性
同じDNAを持ち、同じ環境にいる細胞集団でも、個々の細胞の振る舞いに違いが現れること。この振る舞いの違いは、さまざまな場面で生物にとって有益な結果をもたらすことが近年分かってきた。例えば、細菌を薬で殺菌しようとしても、常に集団の一部が生き残る。また、受精卵やiPS細胞が他の細胞に分化するときにも、細胞の振る舞いの違いが、分化する細胞の違いに反映されることが分かっている。 - 2.全反射照明蛍光顕微鏡
蛍光分子を1分子の精度で検出(1分子イメージング)するための顕微鏡。ガラスと水溶液の界面で励起光を全反射させることで、水溶液中の、界面から100nm程度の高さの領域だけに励起光を閉じ込め、その領域に存在する蛍光分子だけを励起、検出することができる。 - 3.原子間力顕微鏡
試料と顕微鏡探針先端の原子の間に働く力を検出して、試料表面の形態を観察する顕微鏡で、原子レベルの分解能が実現されている。
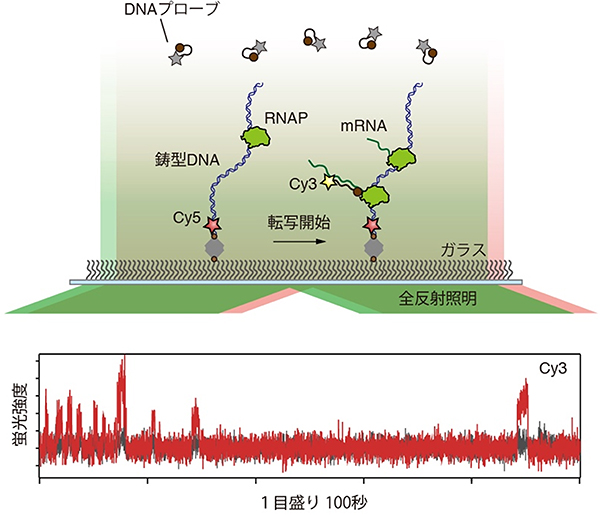
図1 細胞外で再現したmRNA合成の1分子イメージング
上:ガラス表面場で再現した転写は、全反射照明蛍光顕微鏡を用いて観察される。Cy3で標識されたDNAプローブはmRNAと相補的に結合したときにのみ蛍光を検出できる。鋳型DNAはCy5で標識され、その位置を検出できる。
下:鋳型DNA上で変化するCy3の蛍光強度。スパイク状の蛍光強度の変化は、1分子のmRNAが合成されたことを示す。mRNA合成頻度が時間経過とともに大きくゆらぐことを示している。
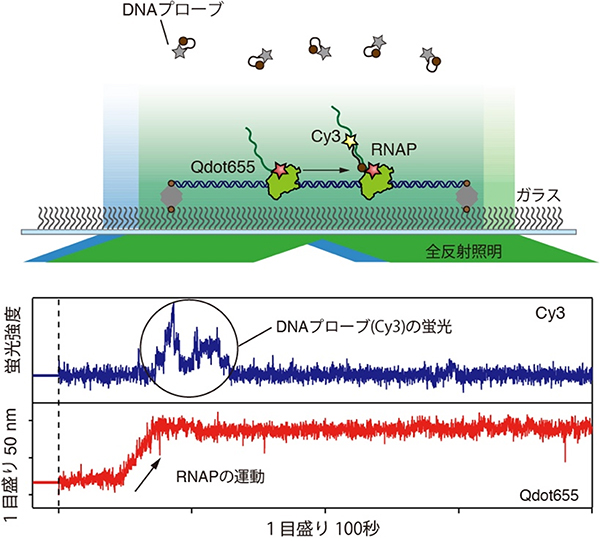
図2 RNAPの1分子イメージング
上:mRNAの合成をCy3で検出するとともに、Qdot665で標識されたRNAPの運動を観察した。
下:RNAPはmRNA合成中に長時間停止することを直接観察した。RNAPの運動後にDNAプローブの蛍光強度が増加していることから、RNAPの動きはmRNA合成中の運動であることが分かる(DNAプローブの結合には時間がかかるため、RNAPの運動とDNAプローブの蛍光強度の増加には時間差がある)。転写される鋳型DNAの長さは150 nmであり、RNAPは鋳型DNAの途中で停止していることが分かる。nmは10億分の1メートル。

図3 原子間力顕微鏡画像
DNA上でRNAPが分子渋滞を起こしていることを示す原子間力顕微鏡画像。RNAPと鋳型DNAを用いて試験管内で転写を再現し、その溶液を雲母上で乾燥させた後、撮像した。細いひも状に見えるのがDNAで、白く丸いものがRNAPである。nmは10億分の1メートル。
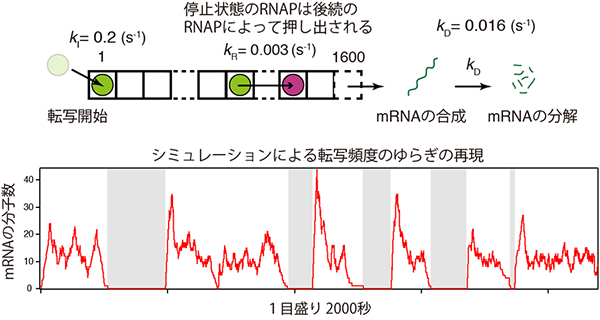
図4 細胞内で起こるmRNA合成のシミュレーションモデル
上:モデルの模式的な説明。一つの箱はDNAの1塩基対を表しており、1,600塩基対(大腸菌で転写される平均的なDNAの長さ)分の箱が並んでいる。RNAPは一定の頻度(kI)で左端から現れ(転写開始)、一つずつ箱を進んでいく。端に到達するとmRNAが合成され、合成されたmRNAは一定の頻度(kD)で分解されると仮定する。RNAPは一定の頻度で停止状態になる(赤色)。停止したRNAPは後続のRNAPに押されているときに一定の頻度(kR)で再び動き出す。s-1は頻度の単位で、1秒間に起こる反応の回数を示す。
下:実際のシミュレーション結果。RNAPの渋滞とその緩和によって、細胞内のmRNAの数は時間とともに大きくゆらぐことが示された。
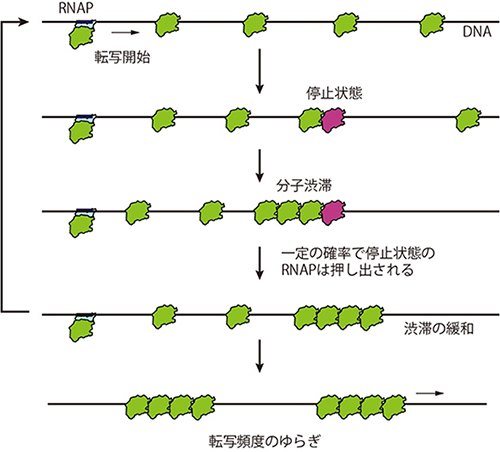
図5 RNAPの渋滞と緩和が生む転写頻度のゆらぎ
転写中のRNAPは一定の確率で停止状態に入る。転写開始は一定の確率で起こるので、後続のRNAPは停止状態のRNAPを先頭に分子渋滞を起こす。しかし、後続のRNAPは、停止状態のRNAPを一定の確率で押し出し、停止状態を解くことができる(RNAPの渋滞と緩和)。結果としてRNAPは集団を作ってmRNAを合成し、単位時間当たりに合成されるmRNA分子の数は大きくゆらぐ(転写頻度のゆらぎ)。
