理化学研究所(理研)計算科学研究センター粒子系生物物理研究チームの杉田有治チームリーダー(開拓研究本部杉田理論分子科学研究室主任研究員、生命機能科学研究センター分子機能シミュレーション研究チームチームリーダー)、開拓研究本部杉田理論分子科学研究室のドカイニッシュ・M・ヒシャム特別研究員らの共同研究グループは、スーパーコンピュータ「富岳」[1]を用いて、新型コロナウイルス(SARS-CoV-2)表面に存在する「スパイクタンパク質[2]」のシミュレーションを行い、ウイルス感染に必要なスパイクタンパク質の構造変化を引き起こす分子機構を明らかにしました。
本研究成果は、新型コロナウイルス感染症(COVID-19)の治療薬やワクチンの開発に貢献するだけでなく、新しい変異型に関する感染予防の知見の基礎を与えるものと期待できます。
SARS-CoV-2のスパイクタンパク質の受容体結合ドメイン(RBD)[2]には、不活性な「ダウン型構造[3]」と活性な「アップ型構造[3]」が存在し、ヒト細胞に侵入する際にはアップ型構造をとることが分かっています。
今回、共同研究グループはスーパーコンピュータ「富岳」を用いて、スパイクタンパク質のダウン型からアップ型への構造変化の分子動力学シミュレーション[4]を行いました。このシミュレーションは通常の手法では困難なことから、独自に開発していた高効率の構造探索アルゴリズム「レプリカ交換分子動力学法[5]」の一つである「gREST法[6]」により、原子レベルでの構造変化を解明することに成功しました。さらに、クライオ電子顕微鏡[7]で明らかになっている安定な立体構造だけでなく、複数の準安定な立体構造の存在を予測しました。
本研究は、科学雑誌『eLife』オンライン版(3月24日付)に掲載されました。
背景
新型コロナウイルス(SARS-CoV-2)が引き起こした新型コロナウイルス感染症(COVID-19)のパンデミックは2019年12月に発生し、今もなお私たちの生活に大きな影響を与えています。SARS-CoV-2自身とそこに含まれるタンパク質やRNAの構造や機能に関する基礎研究が世界中で盛んに行われ、有効なワクチンや医薬品の開発が進められています。しかし、SARS-CoV-2に含まれるタンパク質、特にウイルス表面に存在するスパイクタンパク質の変異型の発生がこの感染症の終息を阻み、パンデミックの長期化につながっています。
SARS-CoV-2のスパイクタンパク質は3本のポリペプチド鎖[8]から構成されており、それぞれのポリペプチド鎖に含まれる「受容体結合ドメイン(RBD)」と呼ばれる部分が不活性な「ダウン型」から活性な「アップ型」に構造変化することで、SARS-CoV-2がヒト細胞に侵入できるようになることが多くの実験により明らかになっています。
また、X線結晶構造解析[9]やクライオ電子顕微鏡を用いた単粒子解析[10]により、複数のRBDの構造がアップ型であることも多数報告されています。しかし、ダウン型からアップ型(三つのRBDが全てアップ型)、あるいは一つのRBDのアップ型(残りの二つはダウン型)から二つのRBDがアップ型(残りの一つはダウン型を取る構造などへの構造変化を直接観察した研究はほとんどありませんでした。また、最も安定な立体構造と思われるダウン型にどのような分子運動が含まれているかも明らかではありませんでした。
2021年に杉田有治チームリーダーらは、独自に開発した分子動力学ソフトウェアGENESIS[11]を用いて、SARS-CoV-2のスパイクタンパク質のダウン型とアップ型の立体構造をスタート構造とした1マイクロ秒(100万分の1秒)間の分子動力学シミュレーションをスーパーコンピュータ「富岳」で実行しました。その結果、スパイクタンパク質表面に多く存在する「糖鎖[12]」がダウン型とアップ型のそれぞれの立体構造を安定化することを解明しました注1)。今回、共同研究グループはスパイクタンパク質に含まれるRBDのダウン型からアップ型への構造変化をシミュレーションすることにしました。
- 注1)2021年2月18日プレスリリース「新型コロナウイルス感染の分子機構を解明」
研究手法と成果
ダウン型からアップ型への構造変化を直接計算することは、通常の分子動力学シミュレーションの手法では不十分であることから、共同研究グループは独自に開発した高効率の構造探索アルゴリズム「レプリカ交換分子動力学法」の一つである「gREST法」を使用しました(図1)。レプリカ交換分子動力学法では、異なる温度を持つ複数の分子動力学シミュレーション(レプリカと呼ぶ)を並列に実行します。計算の途中でその温度を交換して、低温と高温の分子動力学シミュレーションが混じり合った状態を作ることで、高温での多くの構造の探索と低温での安定構造の発見が両立します。gREST法はREST/REST2法を拡張したもので、シミュレーション実行前に、系の一部を「溶質」として定義し、その部分の温度を変えたレプリカを複数用意して、レプリカ交換分子動力学法と同じように温度交換を行います。
gREST法は、タンパク質と阻害剤の結合・解離のシミュレーションにも有効であり、杉田有治チームリーダーらが2019年に行った計算では、「阻害剤分子とタンパク質結合部位のアミノ酸残基」を「溶質」として定義しました注2)。今回はスパイクタンパク質のRBDの動きに着目しているため、三つのRBD表面に含まれる電荷を持つアミノ酸残基(アルギニン、リジン、グルタミン酸、アスパラギン酸)を「溶質」として定義し、それらの相互作用を制御することでRBDの構造変化を比較的短い時間で予測することに成功しました。
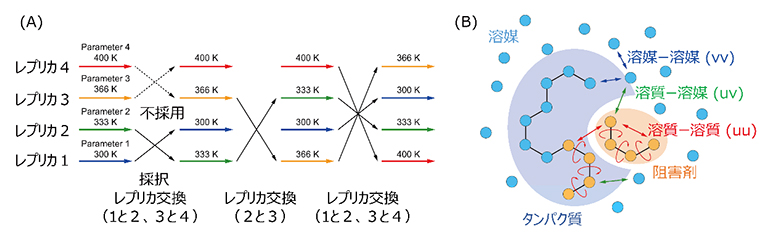
図1 gREST法の模式図
- (A)gREST法やレプリカ交換分子動力学法の計算のイメージ図。異なる色の水平の矢印は異なる温度(あるいは溶質温度)での分子動力学シミュレーションを意味する。メトロポリス判定で「交換」可能かどうか自動的に決定し、温度(あるいは溶質温度)空間での一次元ランダムウォークを実現する。
- (B)gREST法ではシミュレーションモデルの一部を「溶質」(黄色)として定義して、その温度を変えたレプリカを複数用意し、その温度を交換することによって実行的に計算時間を加速する。
- 注2)2019年9月6日プレスリリース「酵素-阻害剤結合の初期会合体を予測」
今回行ったgREST法に基づく分子動力学シミュレーションは、ダウン型を初期モデルとした計算、アップ型を初期モデルとした計算、ダウン型を初期モデルとして故意にスパイクタンパク質表面の糖鎖を含めない計算の3種類です。各計算に使ったモデル系には、スパイクタンパク質と水、イオンが含まれており、全体で約65万原子が含まれます。さらにgREST法では、溶質温度の異なるモデル系のコピー(レプリカ)が16個必要でした。計算全体として約1040万(=65万X16)の原子を含む分子動力学シミュレーションを実行するため、スーパーコンピュータ「富岳」(以下「富岳」)において2048ノード[13]を同時に(並列に)使う必要がありました。「富岳」以外の国内外のスーパーコンピュータで、このような超大規模な計算を継続的に実行することは困難でした。
さらに、「富岳」での計算終了後、得られたビッグデータ[14]を解析することも容易ではありませんでした。さまざまな解析技術を開発し組み合わせることで、複雑なスパイクタンパク質の構造変化の解明に成功しました。クライオ電子顕微鏡によって明らかにされたスパイクタンパク質の立体構造と比較した結果、通常のダウン型(三つのRDB全てがダウン型)だけでなく、一つのRBDがアップ型を取る「1アップ型」、一つのRBDが通常のアップ型よりもさらに開いた構造を取る「1オープン型」、二つのRBDがアップ型に近い構造を取る「2アップ(ライク)型」といった非対称な構造をシミュレーションで予測できたことが分かりました(図2)。
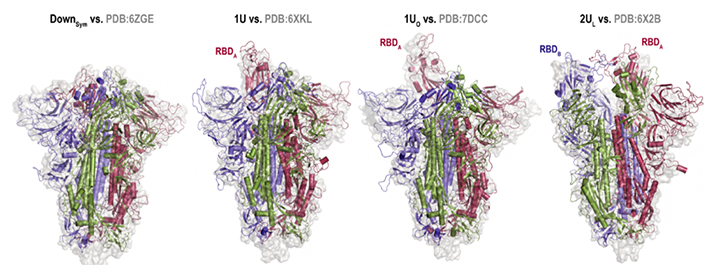
図2 分子動力学シミュレーションとクライオ電子顕微鏡で得られた構造の比較
左から、「ダウン型」「1アップ型」「1オープン型」「2アップ(ライク)型」についての分子動力学シミュレーション(リボン図)とクライオ電子顕微鏡(サーフェイス図)で得られた構造の比較(eLife 2022;11:e75720より転載)
さらに、これらの三つの安定構造への変化の途上に存在するさまざまな準安定な中間体の構造も明らかになりました。これらの中間体の立体構造は、安定構造には見られないポケット(くぼみ)が存在していました(図3)。このような過渡的な立体構造に見られるポケットは、スパイクタンパク質をターゲットとした薬剤開発の際のスクリーニングに利用できる可能性があります。
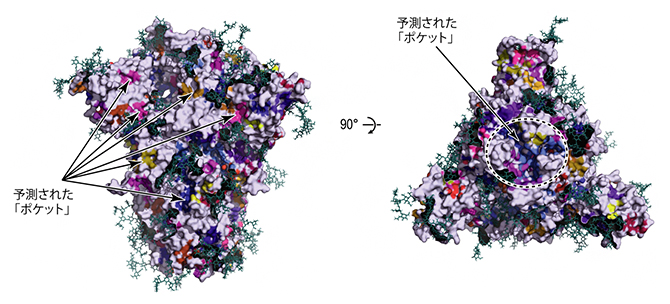
図3 分子動力学シミュレーションで得られた立体構造に含まれる薬剤が結合し得るポケット
シミュレーションで得られた立体構造には、安定構造には見られないポケット(くぼみ)が存在していた。このポケットは薬剤が結合する可能性のある部位として、バーチャルスクリーニングなどインシリコ創薬の手法に活用できる(eLife 2022;11:e75720より転載)
また、既に解明されていたダウン型とアップ型における糖鎖との相互作用だけでなく、過渡的な立体構造における糖鎖との相互作用も明らかになりました。ワクチン開発によって、多くの抗体分子がスパイクタンパク質表面に結合し、ダウン型からアップ型への構造変化を阻害することで、ヒト細胞への感染を予防することが示されています。クライオ電子顕微鏡で解析された安定構造だけでなく、過渡的な構造や中間構造についても糖鎖を含めた相互作用を解析することで、抗体分子との結合のしやすさについても検討し、その詳細な分子機構が明らかになりました(図4)。
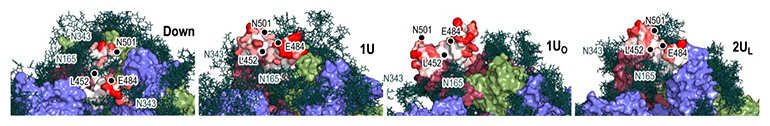
図4 スパイクタンパク質・糖鎖・抗体の相互作用
左から、スパイクタンパク質の「ダウン型」「1アップ型」「1オープン型」「2アップ(ライク)型」の各構造における糖鎖と抗体分子の相互作用。抗体分子は、クライオ電子顕微鏡によって得られた複合体の中で、シミュレーション構造と最も近いものを用いて可視化している。糖鎖を緑色のスティックモデルで、抗体分子を白~赤のサーフェイスモデルで表現した(eLife 2022;11:e75720より転載)
今後の期待
本研究では、分子動力学シミュレーションにより、SARS-CoV-2スパイクタンパク質の構造変化を生み出す分子機構を示すことができました。また、これまでは非常に安定な構造であると思われていたダウン型構造においても、対称性を維持した構造(クライオ電子顕微鏡で解かれたものと同じ)だけではなく、非対称な立体構造も含まれていることが分かりました。つまり、スパイクタンパク質が持つ本来の立体構造が非常に"柔らかい"ことが分かりました。
本研究で対象としたのは、変異が導入される前のアミノ酸配列を持つスパイクタンパク質ですが、今後はオミクロン株など異なるアミノ酸配列を持つスパイクタンパク質の柔らかさも同じ手法で予測できます。この立体構造の柔らかさは、ダウン型からアップ型への構造変化のしやすさとも関係していると考えられるため、SARS-CoV-2の感染性変化の予測にもつながると期待できます。
補足説明
- 1.スーパーコンピュータ「富岳」
「京」の後継機。社会的・科学的課題の解決で日本の成長に貢献し、世界をリードする成果を生み出すことを目的とし、電力性能、計算性能、ユーザーの利便性・使い勝手の良さ、画期的な成果創出、ビッグデータやAI(人工知能)の加速機能の総合力において世界最高レベルのスーパーコンピュータ。15万8976個の中央演算装置(CPU)を搭載し、1秒間に約44京2010兆回の計算が可能。2020年6月から2021年11月にかけて、世界のスパコンランキング「TOP500」「HPCG」「HPL-AI」「Graph500」で4期連続の世界一位を獲得した。 - 2.スパイクタンパク質、受容体結合ドメイン(RBD)
新型コロナウイルスなどの表面に見られる「トゲ」の部分をスパイクタンパク質と呼ぶ。受容体結合ドメイン(RBD)はスパイクタンパク質の一部で、ヒト細胞表面の受容体と結合することで感染が引き起こされる。 - 3.ダウン型構造、アップ型構造
スパイクタンパク質の構造はX線結晶構造解析やクライオ電子顕微鏡解析によって決定された。感染性の低いダウン型構造と感染性の高いアップ型構造が存在することが知られている。スパイクタンパク質は同一の三つのポリペプチド鎖からなる大きなタンパク質であり、それぞれのポリペプチド鎖に受容体結合ドメイン(RBD)を一つずつ持つ。アップ型については、さまざまな立体構造がクライオ電子顕微鏡によって決定されている。 - 4.分子動力学シミュレーション
コンピュータを用いた分子シミュレーション法の一つ。原子間相互作用をフックの法則やクーロンの法則などから計算し、分子系の運動をニュートン方程式 F = maに基づいて数値的に解くことで、分子の動きを理論予測し解析する方法。 - 5.レプリカ交換分子動力学法
構造探索効率を上げるための方法の一つ。系の複数コピー(レプリカ)に対して温度の異なる分子動力学法計算を実施して、レプリカ間で温度を適宜交換することで構造探索の効率を上げる。この方法はいわゆる「拡張アンサンブル法」の一つと考えられる。拡張アンサンブル法は、エネルギーあるいは構造空間の一次元ランダムウォークを実現することによって、少数の状態にとどまることなく広い構造空間の探索が可能になる。さらに、再重法などと組み合わせることで熱力学平均量を計算することができる。1999年に岡本祐幸特任教授(名古屋大学)と杉田有治チームリーダーによって開発された。 - 6.gREST法
系の一部を「溶質分子」として定義し、その温度だけが異なるレプリカ間で交換することで、少ないレプリカ数で構造探索の効率を上げる計算方法をREST(Replica-Exchange with Solute Tempering)法という。gREST法は、構造探索効率を上げるために溶質分子の定義を拡張した計算方法で、generalized RESTの略。 - 7.クライオ電子顕微鏡
タンパク質を含む溶液を極低温(液体窒素温度)にまで急速に冷却し、試料を観察する透過型電子顕微鏡。近年、試料調製法の改良や、電子直接検出器の開発、解析ソフトの進歩により、近原子分解能の性能が得られるようになった。2017年、タンパク質立体構造解析への応用に貢献したジャック・デュボシェ、ヨアヒム・フランク、リチャード・ヘンダーソンの3氏にノーベル化学賞が授与されている。 - 8.ポリペプチド鎖
アミノ酸がペプチド結合を介して長く連なった生体高分子化合物。天然には20種類のアミノ酸が存在し、それぞれ異なる化学的性質を持っている。例えば、セリン、スレオニン、アスパラギンは親水性、バリン、イソロイシンは疎水性、アスパラギン酸、グルタミン酸は負電荷、リシン、アルギニンは正電荷を持っている。このようなアミノ酸が連なることで、特定の立体構造を形成する。特に細胞内で機能を発現するポリペプチドはタンパク質と呼ばれる。 - 9.X線結晶構造解析
タンパク質の結晶を作製し、その結晶にX線を照射して得られる回折データを解析することで、タンパク質の内部の原子の立体的な配置を調べる方法。この方法によって、タンパク質の立体構造や内部構造を知ることができる。 - 10.単粒子解析
クライオ電子顕微鏡によって観察された溶液中にランダムに配向したタンパク質の多数の投影像から立体像を再構築する手法。 - 11.分子動力学ソフトウェアGENESIS
理研計算科学研究センターを中心に開発されている分子動力学ソフトウェア(GENESIS)。細胞環境を含む大規模な生体分子系のシミュレーションやレプリカ交換法などの構造探索手法を利用できるという特徴を持つ。 - 12.糖鎖
グルコース、ガラクトースなどの単糖がグリコシド結合を介して長く連なった化合物。多くのタンパク質の表面は、小胞体やゴルジ体内で酵素の働きにより糖鎖が付加される。糖鎖の修飾を受けたタンパク質は、糖タンパク質と呼ばれ、糖鎖はタンパク質の安定性やウイルスの認識などに重要な役割を果たす。 - 13.ノード
スーパーコンピュータにおけるオペレーティングシステムが動作できる最小の計算資源の単位。「富岳」の場合は、1つのCPU(中央演算装置)および32GiB(ギビバイト)のメモリから構成される。 - 14.ビッグデータ
さまざまな計測のみならず、シミュレーションにおいても大量データが生成され、その解析によって、対象とする系の特徴を見いだすことができる。分子動力学シミュレーションでは、系に含まれる原子数と計算時間の長さに比例してデータ量が増大する。さらに、レプリカ交換分子動力学法やgREST法ではレプリカ数にも比例する。従って得られるデータは膨大であり、このビッグデータを効率良く解析することが研究において非常に重要である。
共同研究グループ
理化学研究所
計算科学研究センター 粒子系生物物理研究チーム
チームリーダー 杉田 有治(すぎた ゆうじ)
(開拓研究本部 杉田理論分子科学研究室 主任研究員、生命機能科学研究センター 分子機能シミュレーション研究チーム チームリーダー)
技師 小林 千草(こばやし ちぐさ)
(運用技術部門 ソフトウェア開発技術ユニット 技師)
研究員 ジョン・ジェウン(Jaewoon Jung)
(開拓研究本部 杉田理論分子科学研究室 専任技師)
開拓研究本部 杉田理論分子科学研究室
特別研究員 ドカイニッシュ・M・ヒシャム(Hisham M. Dokainish)
専任研究員 森 貴治(もり たかはる)
医薬基盤・健康・栄養研究所
研究員 李 秀栄(り すよん)
研究支援
本研究は、「富岳」の優先的な試行的利用課題「「富岳」を用いた新型コロナウイルス表面のタンパク質動的構造予測(課題代表者:杉田有治)」および新型コロナウイルス感染症対応HPCI臨時公募課題「新型コロナウイルス表面のタンパク質動的構造予測(課題代表者:杉田有治)(課題番号:hp200153)」を通じて、スーパーコンピュータ「富岳」と「Oakforest-PACS」の計算資源の提供を受け、実施しました。
原論文情報
- Hisham M. Dokainish, Suyong Re, Takaharu Mori, Chigusa Kobayashi, Jaewoon Jung, Yuji Sugita, "The inherent flexibility of receptor binding domains in SARS-CoV-2 spike protein", eLife, 10.7554/eLife.75720
発表者
理化学研究所
環境資源科学研究センター 粒子系生物物理研究チーム
チームリーダー 杉田 有治(すぎた ゆうじ)
(開拓研究本部 杉田理論分子科学研究室 主任研究員、生命機能科学研究センター 分子機能シミュレーション研究チーム チームリーダー)
開拓研究本部 杉田理論分子科学研究室
特別研究員 ドカイニッシュ・M・ヒシャム(Hisham M. Dokainish)
報道担当
理化学研究所 計算科学研究センター 広報グループ
Email: r-ccs-koho [at] ml.riken.jp
理化学研究所 広報室 報道担当
広報室 お問い合わせフォーム
