2016年1月8日
理化学研究所
次世代型逆遺伝学による睡眠遺伝子Nr3aの発見
-交配不要で解析も簡便かつ低コストな新しい逆遺伝学を確立-
要旨
理化学研究所(理研)生命システム研究センター合成生物学研究グループの上田泰己グループディレクター、砂川玄志郎 元研究員(現 理研多細胞システム形成研究センター網膜再生医療研究開発プロジェクト研究員)、鵜飼(蓼沼)磨貴テクニカルスタッフⅠ、ディミトリ・ペリン元研究員(現 客員研究員)、高速ゲノム変異マウス作製支援ユニットの隅山健太ユニットリーダーらの研究チームは、特定の遺伝子をノックアウトした個体を、交配を必要とせず効率よく作製する「トリプルCRISPR法」と、呼吸パターンを用いることで非侵襲かつ高効率に睡眠表現型解析[1]を行う「SSS(Snappy Sleep Stager)」を開発し、次世代型の逆遺伝学を実現するプラットフォームを確立しました。そして、この手法により新たな睡眠遺伝子「Nr3a」を、マウスを使った実験で発見しました。
特定の遺伝子を改変したりノックアウトし、どのような表現型が得られるかを観察する遺伝学を「逆遺伝学(リバースジェネティックス)[2]」と呼びます。逆遺伝学は従来の遺伝学と比べて遺伝子から生命現象への対応が分かりやすく、遺伝子と生命現象を結びつける強力な手段といえます。逆遺伝学で個体ごとに異なる行動や睡眠などの複雑な生命現象を観察するには、数多くの遺伝子改変動物を効率よく作製する技術と、個体ごとの表現型を効率よく解析する技術が不可欠です。
研究チームが開発した「トリプルCRISPR法」と「SSS」を用いると、特定の遺伝子をノックアウトしたマウスを効率よく短期間で作製し、個体ごとに睡眠の表現型を非侵襲かつ高効率に解析できます。実際にこの手法を用いて、さまざまな精神疾患との関連が知られているNMDA受容体[3]の7つのサブユニットの遺伝子を、それぞれノックアウトしたマウスを作製し、睡眠表現型解析を行ったところ、Nr3a遺伝子をノックアウトしたマウスの睡眠時間が、野生型のマウスと比較して有意に短いことが分かりました。Nr3a遺伝子は今まで睡眠との関係は知られておらず、新たな睡眠遺伝子といえます。
本成果は、米国の科学雑誌『Cell Reports』(1月26日号)に掲載されるのに先立ち、オンライン版(1月7日付け:日本時間1月8日)に掲載されます。
背景
哺乳類の睡眠時間と覚醒時間の割合が一定に保たれていることは知られていますが、その理由はよく分かっていません。一方で、睡眠障害(不眠や過眠など)は現代人を悩ませている代表的な精神疾患の1つです。睡眠障害はさまざまな精神疾患や神経変性疾患(うつ病、パーキンソン病、アルツハイマー病など)の合併症としても見受けられます。睡眠障害に対する診断法や治療法の開発には、睡眠と覚醒のバランスがどのように保たれているのかを理解することが重要です。
近年、特定の遺伝子と生命現象を結びつける強力な研究手法として「逆遺伝学(リバースジェネティックス)」が注目されています。逆遺伝学では、特定の遺伝子を改変したりノックアウトし、どのような表現型が得られるかを観察します。一方、従来の遺伝学では、特定の生命現象を示す動物のゲノムを詳細に調べることによって関係している遺伝子を同定していくため、逆遺伝学は従来の遺伝学と比べて、遺伝子から生命現象への対応が分かりやすいという特徴があります。遺伝子改変動物の作製技術の進歩に伴い、逆遺伝学を用いた研究が数多く行われ、より多くの遺伝子の機能が明らかにされてきています。
しかし、睡眠現象のような個体ごとに異なる複雑な生命現象に対して逆遺伝学を用いて効率的に研究を行うには、2つの課題がありました。1つ目の課題は、数多くの遺伝子改変動物を高速に作製することです。これまでの技術では、遺伝子改変動物を大量に作製するには複数回の交配を介するため、少なくとも年単位の時間が必要でした。2つ目の課題は、作製した遺伝子改変動物に対して個体ごとに睡眠を高効率かつ定量的に解析することです。従来、小動物の睡眠を解析するには、脳波を取得するための電極を頭蓋骨に装着する必要がありました。これは手術を伴うため、動物に対して侵襲性が高く、安定した解析を行うには高い技術を持つ多くの人材が必要となり、費用も高くなります。
そこで研究チームは、高速な遺伝子改変動物の作製と高速な睡眠表現形解析を実現し、個体ごとの睡眠制御メカニズムを逆遺伝学によって明らかにするプラットフォームの確立を目指して、新たな技術の開発に取り組みました。
研究手法と成果
(1)トリプルCRISPR法の開発
近年、CRISPR/Cas法[4]の開発によりゲノムを効率よく編集できるようになり、受精卵に同手法を用いることで特定の遺伝子をノックアウトした動物(遺伝子ノックアウト動物)が従来の方法よりも高効率で作製できるようになりました。しかし、遺伝子のノックアウト率は約50%程度でした。したがって、遺伝子ノックアウト動物を大量に作製するには数回の交配が必要となり、少なくとも1年以上の時間が必要でした。また、複数の遺伝子を同時にノックアウトしようとすると、効率がさらに落ちるという課題もありました。
CRISPR/Cas法では1つの遺伝子(対象遺伝子)に対して1ヶ所をガイドRNAで切断していました。研究チームは、ガイドRNAのデザインを改良し、3ヶ所を同時に切断する「トリプルCRISPR法」を開発しました。この手法をマウスに適用したところ、1世代目で極めて高い確率(ほぼ100%)で大量の遺伝子ノックアウトマウスを作製することに成功しました(図1)。開発した手法では、交配を重ねる必要がないため、遺伝子改変マウスの作製期間はわずか3ヶ月程度です。
(2)SSS(Snappy Sleep Stager)の開発
動物の睡眠を解析するには、脳波と呼ばれる脳の電気活動によって生じる微弱な電位変化を検出する必要があります。小動物の場合、正確な脳波を取得するには頭蓋骨に複数の電極を装着する必要があります。これは手術を伴うため、動物にとって侵襲性が高く、手法そのものの難易度や費用も高いことから、効率よく睡眠表現型解析を行うことは困難でした。そこで研究チームは、マウスの呼吸を触れることなく計測し、呼吸パターンから睡眠を解析する「SSS(Snappy Sleep Stager)」を開発しました(図2)。これにより、非侵襲な解析が可能となり、高度な技術も必要とせずコストは従来の10%以下という、これまでにない効率かつ簡便な睡眠表現型解析を実現しました。
(3)NMDA受容体を用いた睡眠遺伝子の選別
睡眠と覚醒のバランスを制御しているメカニズムはいまだに分かっていません。臨床的には多くの精神疾患や神経変性疾患で睡眠障害との関連が知られています。そこで、多くの精神疾患と関与が指摘されているNMDA受容体を構成する7つのサブユニットの遺伝子をそれぞれノックアウトしたマウスをトリプルCRISPR法で作成し、SSSによる睡眠解析を行うことで、睡眠と関連する遺伝子を選別しました。その結果、7つのサブユニットの遺伝子のうちNr3a遺伝子をノックアウトしたマウスの睡眠時間が、野生型のマウスと比べて有意に短いことがわかりました(図3)。Nr3a遺伝子は今まで睡眠との関係は知られておらず、新たな睡眠遺伝子といえます。
研究チームが開発した手法では、従来の逆遺伝学を用いた睡眠表現型解析における、遺伝子ノックアウト動物を得るための長い時間と、解析の難解さや高コストといった課題をクリアしました。この点で、次世代型の逆遺伝学を実現するプラットフォームを確立したといえます。
今後の期待
研究チームが確立した次世代型逆遺伝学のプラットフォームを応用することで、哺乳類の睡眠制御メカニズムのより深い理解に繋がると期待できます。具体的には、多くの遺伝子をノックアウトした動物を短期間に作製し個体ごとに睡眠への影響を調べることができるようになったため、逆遺伝学を用いた睡眠の研究を、より高い効率で行うことができます。
また、トリプルCRISPR法を応用することで、より複雑な遺伝子改変動物を高速に作成できる可能性があります。さらに、SSSはマウスだけでなく、ヒトを含めた他の動物の睡眠を非侵襲に測定する手法としての応用が期待できます。
原論文情報
- Genshiro A. Sunagawa, Kenta Sumiyama, Maki Ukai-Tadenuma, Dimitri Perrin, Hiroshi Fujishima, Hideki Ukai, Osamu Nishimura, Shoi Shi, Rei-ichiro Ohno, Ryohei Narumi, Yoshihiro Shimizu, Daisuke Tone, Koji L. Ode, Shigehiro Kuraku and Hiroki R. Ueda, "Mammalian reverse genetics without crossing reveals Nr3a as a short-sleeper gene", Cell Reports, doi: 10.1016/j.celrep.2015.12.052
発表者
理化学研究所
生命システム研究センター 細胞デザインコア 合成生物学研究グループ
グループディレクター 上田 泰己 (うえだ ひろき)
元研究員 砂川 玄志郎 (すながわ げんしろう)
(現 理研多細胞システム形成研究センター網膜再生医療研究開発プロジェクト研究員)
テクニカルスタッフⅠ 鵜飼(蓼沼) 磨貴(うかい(たでぬま) まき)
元研究員 ディミトリ・ペリン(Dimitri Perrin)(現 客員研究員)
生命システム研究センター 細胞デザインコア 合成生物学研究グループ 高速ゲノム変異マウス作製支援ユニット
ユニットリーダー 隅山 健太 (すみやま けんた)
 上田 泰己
上田 泰己
 砂川 玄志郎
砂川 玄志郎
 鵜飼(蓼沼) 磨貴
鵜飼(蓼沼) 磨貴
 ディミトリ・ペリン
ディミトリ・ペリン
 隅山 健太
隅山 健太
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.睡眠表現型解析
動物の睡眠・覚醒パターンを解析すること。脳波を用いた解析が標準的であるが、小動物では動物への侵襲性、実験手技の難易度の高さ、コストの高さの問題があり、大規模な解析は困難であった。 - 2.逆遺伝学(リバースジェネティックス)
特定の遺伝子を改変したりノックアウトし、生命現象がどのように変化するか観察することで遺伝子機能を解析する手法。特定の生命現象を示す動物のゲノムを詳細に調べることによって関係している遺伝子を同定していく従来の遺伝学をフォワードジェネティクス(生命現象から遺伝子)と呼ぶのに対し、遺伝子から生命現象を関連付けるため、解析法が逆向きであり、リバース(=逆方向の)ジェネティクスと呼ばれるようになった。 - 3.NMDA受容体
グルタミン酸受容体の1つ。シナプスの可塑性や記憶に関連する受容体として知られる。多くの精神依存性のある薬物(覚せい剤など)の作用部位としても知られ、NMDA受容体の状態を乱すことで鎮静状態や興奮状態を誘導できる。 - 4.CRISPR/Cas法
CRISPR/Cas系を用いたゲノム編集技術の1つ。CRISPR/Cas系は古細菌が持つ獲得免疫機構で、外来性のDNAを自らのゲノムに組み込み、2度目の感染のときにこの外来性のDNAの断片から2つのガイドRNAを発現させ、侵入してきた標的DNAを、Cas9ヌクレアーゼを用いて切断する系である。1つのガイドRNAとCas9の2つの因子だけでゲノムDNAの切断ができることが分かっており、手法の簡便さからゲノム編集の主たる手法となりつつある。
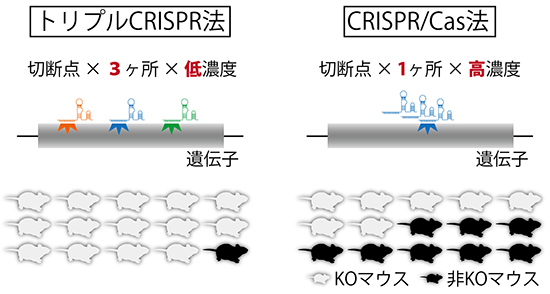
図1 トリプルCRISPR法とCRISPR/Cas法(従来法)の比較
従来のCRISPR/Cas法では1つの対象遺伝子の1ヶ所をガイドRNAで切断していたが、トリプルCRISPR法では3ヶ所を同時に切断する。これにより効率のよい遺伝子編集を実現した。切断効率が上がったことで、ガイドRNAの濃度を上げることなくノックアウト率を上げることに成功している。従来法では50%程度だった遺伝子のノックアウト率は、ほぼ100%となった。
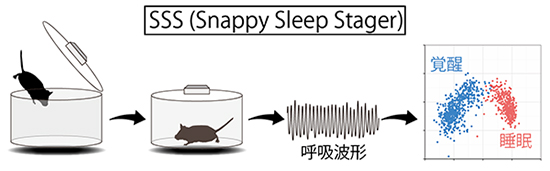
図2 SSS(Snappy Sleep Stager)によるマウスの睡眠解析
SSSではマウスをチャンバー内に収容するだけで、マウスに侵襲を加えることなく呼吸パターンを検出する。検出した呼吸パターンに基づいて全自動で睡眠・覚醒の解析を行う。
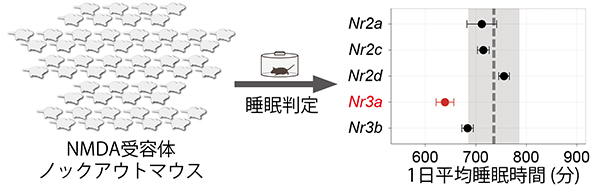
図3 Nr3a遺伝子のノックアウトマウスにおける睡眠時間の減少
トリプルCRISPR法によってNMDA受容体の7つのサブユニットの遺伝子を、それぞれノックアウトしたマウスを作製した。そのマウスにSSSを用いて睡眠解析を行ったところ、Nr3a遺伝子をノックアウトしたマウスの睡眠時間が、野生型のマウスと比べて有意に短いことが分かった。グラフ中の破線は野生型マウスの睡眠時間を示す。
