理化学研究所(理研)生命機能科学研究センター 先端バイオイメージング研究チームの渡邉 朋信 チームリーダー(広島大学 原爆放射線医科学研究所 教授)、細胞極性統御研究チームの岡田 康志 チームリーダー(東京大学大学院 医学系研究科 教授・同大学院 理学系研究科 兼務)、広島大学 両生類研究センターの岡本 和子 助教らの共同研究グループは、マウスES細胞[1]で働く転写因子[2](Nanog、Oct4)の挙動を1分子精度で定量解析し、ES細胞の分化多能性を維持するための新しいメカニズムを発見しました。
本研究成果は、幹細胞研究分野に新たな知見をもたらすほか、iPS細胞[3]の作製効率の向上や品質の安定化などに貢献するものと期待できます。
ES細胞は、体のどの細胞にも分化できる性質(分化多能性)を持っています。NanogとOct4は、ES細胞が分化多能性を維持するために必須の転写因子であり、自分自身の発現をそれぞれ促進させるとともに、互いの発現も促進させます。これまでNanogやOct4の細胞内での分子動態とクロマチン[4]構造の変化、分化多能性との関連性は明らかにされていませんでした。
今回、共同研究グループはタンパク質1分子の運動を観察できる特殊な顕微鏡を用いて、マウスES細胞が分化する瞬間のNanogとOct4の動きを1分子精度で観察し、DNA上での滞留時間や頻度など物理的な挙動に関するさまざまな特徴を定量しました。解析の結果、Nanogは分化が始まるとDNA上に長くとどまるようになるなどの新たな相関を発見し、NanogとOct4が協働してES細胞の分化が進み過ぎないように制御するという新しい「負のフィードバック機構」の提案に至りました。
本研究は、科学雑誌『The EMBO Journal』オンライン版(8月23日付:日本時間8月23日)に掲載されました。
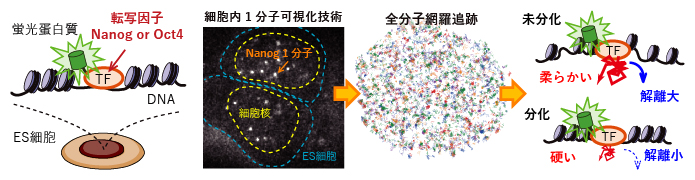
今回の研究の流れを示す概略図
背景
受精卵が分裂して私たちの体を作る細胞に分化し始めるのは、胚盤胞と呼ばれる初期胚の幹細胞(内部細胞塊)が出発点となります。ES細胞(胚性幹細胞)は内部細胞塊から樹立された細胞で、体を構成する全ての細胞に分化できる分化多能性を持っており、この分化多能性を維持するために、NanogやOct4をはじめとする転写因子が細胞核の中で働いています。Oct4はiPS細胞(人工多能性幹細胞)の樹立に用いられる因子の一つであり、またNanogの発現はiPS細胞の品質を反映することが知られています。
従って、これらの転写因子が分化多能性を維持する仕組みの理解は、iPS細胞の生産と品質管理、あるいはES細胞やiPS細胞の分化制御など、再生医療に大きく貢献すると期待されています。そのため、多くの研究者がそれぞれの研究分野でさまざまな先端技術を駆使して、この問題に取り組んできました。これまでの生物学実験の結果と計算機実験による解析から、NanogとOct4は協働してES細胞の分化多能性状態を安定化させており、それらの転写活性の制御には、DNAが折り畳まれて格納されているクロマチン構造の局所的な変化が関与することが示唆されています(図1)。
しかし、これらの仮説の最終的な証拠を提示するには、細胞内で機能している転写因子の直接観察が不可欠です。そこで本研究では1分子計測技術を用いて、NanogやOct4の細胞内での実際の挙動を直接観察し、NanogやOct4の分子動態とクロマチン構造の変化、分化多能性との関連性を探りました。
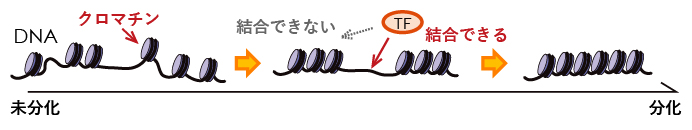
図1 クロマチン構造と細胞分化の関係を示す概念図
細胞核の中でDNA(黒線)は、クロマチンと呼ばれる構造に折り畳まれ格納されている。転写因子(TF)は、折り畳まれていない部分にしか相互作用できない。ES細胞では、分化の進行に伴いクロマチンが凝集されていくため、転写因子は限られた箇所にしか結合できなくなる。その結果、それぞれ特異的な機能を持つ体細胞へと分化すると考えられている。
研究手法と成果
NanogとOct4の1分子計測を実現するには、細胞内のNanogとOct4を蛍光タンパク質[5]で標識する必要があります。まず、共同研究グループはゲノム編集技術[6]を用いて、NanogもしくはOct4と蛍光タンパク質との融合タンパク質を発現するマウスES細胞を作製しました。通常の蛍光顕微鏡では、蛍光タンパク質1分子を観察することはできませんが、薄層斜光照明法(HILO法)[7]を用いることで注1)、転写因子に融合した蛍光タンパク質を、生きたES細胞の核内で1分子ずつ観察できるようになりました(図2上)。
また、遊離状態の転写因子は細胞核内をカメラでは撮像できないほど速く拡散運動していますが、転写因子が標的遺伝子座[8]と相互作用すると運動が停止するため、輝点として捉えることができます。そこで、NanogやOct4が相互作用する遺伝子座に注目して動画を撮影した結果、同じ遺伝子座にNanogやOct4が繰り返して相互作用する様子を確認できました(図2下)。
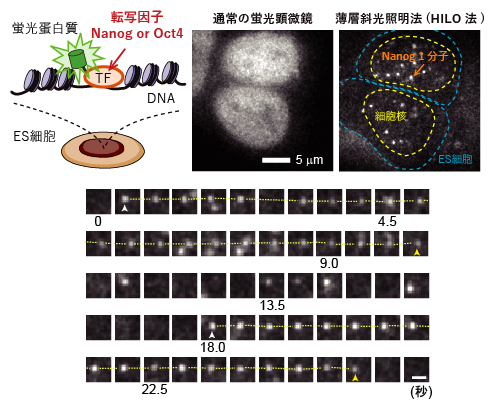
図2 核内の転写因子(NanogとOct4)の1分子計測
- (上段)実験の模式図(左)と、通常の蛍光顕微鏡および薄層斜光照明法(HILO法)による観察の比較(中・右)。ゲノム編集により、一つの転写因子(TF)に一つの蛍光タンパク質を融合させた。このES細胞を通常の蛍光顕微鏡で観察すると、核全体が光っているように見えるが、HILO法では蛍光タンパク質を融合した転写因子(この図ではNanog)の一つ一つが白い輝点として確認でき、1分子観察が実現できた。
- (下段)ある遺伝子座付近を連続的に観察した例。NanogがDNAへの結合と解離を繰り返す様子が分かる。よく見ると輝点は完全に停止しておらず、ゆらゆらと動いている。スケールバーは1マイクロメートル(µm、1µmは1,000分の1mm)。
白血病抑制因子[9]などを含む培養液を用いることで、「未分化状態」を維持させたままマウスES細胞を培養することが可能です。白血病抑制因子を培養液から除去すると、ES細胞の「分化が誘導」されます。逆に、さらに2種類の試薬を加えて培養すると、分化多能性のより高い「基底状態」にもできます。分化多能性の程度に応じた三つの培養条件(基底状態、未分化状態、分化誘導後)において、NanogおよびOct4を蛍光タンパク質によりそれぞれ標識した2種類の細胞について1分子観察を行いました。その結果、500~2,000個の輝点を含む4,000枚の画像からなる動画を計500本以上(再現性確認用や予備実験用を含む)収集しました。この膨大なデータの解析には、輝点の運動を網羅的に計測する1分子追跡法(SMT)[10]と、新たに独自開発した全自動輝点識別アルゴリズムを用いました。
これまで、ES細胞が分化すると、NanogとOct4の転写が不活性化されてNanogとOct4の「発現量」は低下し、分化が進むとNanogとOct4の標的遺伝子座上での「相互作用時間」は短くなると予想されていました。
しかし、基底状態、未分化状態、分化誘導後におけるNanogの標的遺伝子座からの解離速度(相互作用時間の逆数)を定量したところ、予想とは異なり、基底状態よりも未分化状態の方が相互作用時間は長くなっていました(図3左の緑丸)。さらに、同じ分化状態でも、Nanogの発現量が低い細胞の方が相互作用時間は長くなっていました(図3左の棒グラフ)。細胞がNanogの発現を減らして分化方向に進もうとしても、Nanogはそれに反抗して、未分化状態に戻そうとするかのように、標的遺伝子座との相互作用時間を増やしている描像が思い浮かばれます。一方、Oct4はNanogとは異なる挙動を示したことから、Nanogとは異なる機能を担っていると考えられます(図3右)。
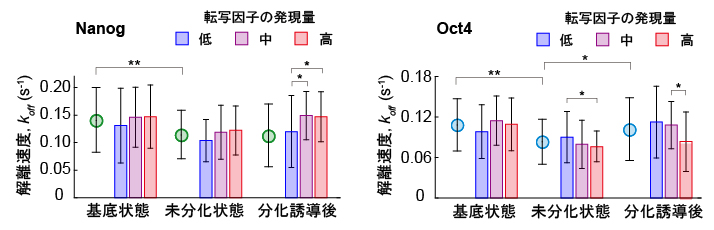
図3 NanogおよびOct4の標的遺伝子座との相互作用時間および発現量の関係
- (左)解離速度koff(相互作用時間の逆数)とNanogの発現量との相関。緑丸は平均値を示す。解離速度が小さいほど、「相互作用時間」が長いことを示す。分化の進み方は、基底状態→未分化状態→分化誘導後の順番である。Nanogは分化が進んだ細胞の方が、相互作用時間が長く、また同じ分化度の細胞では、その「発現量」が低い細胞ほど相互作用時間が長い。
- (右)解離速度koffとOct4の発現量との相関。Oct4は未分化状態では基底状態より相互時間が長くなるが、分化誘導後は相互作用が短くなる。また、基底状態を除く同じ分化度の細胞では、その発現量が低い細胞ほど相互作用時間が短い。
標的遺伝子座に結合した転写因子は、縄にくくられたボールのように、DNAとともにゆらゆらと動きます。つまり、転写因子の動きは標的遺伝子座周辺のDNAの様子を表します。そこで、この"ゆらゆら"を定量するため、輝点の軌跡(図4左上段)から運動の大きさを表す指標を計算し、そこから輝点の「動く範囲」を見積もりました。ES細胞の分化に伴ってクロマチンが凝集すると、揺れる範囲は小さくなるので、転写因子の動く範囲は小さくなると予想されます(図4左下段)。計算の結果、Nanogの動く範囲は予想通り、基底状態から未分化状態で小さくなりましたが、Oct4の動く範囲は分化多能性の程度による変化が見られませんでした(図4右)。つまり、Oct4が相互作用する遺伝子座の周辺のDNAは一貫して折り畳まれていないと考えられます。
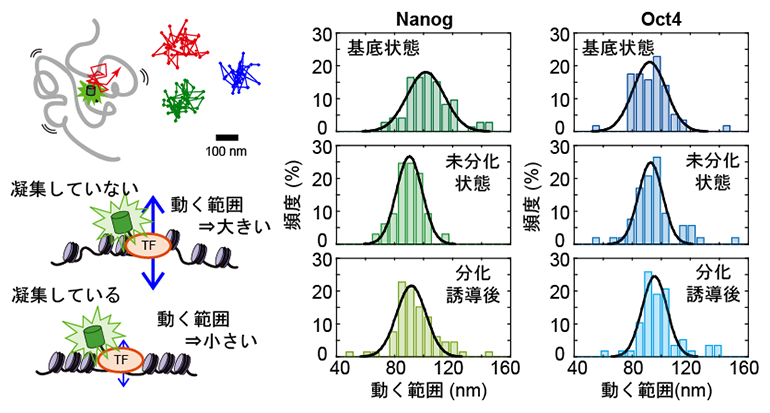
図4 転写因子の動く範囲とクロマチン構造の関係
- (左)DNAに結合した転写因子の運動の概念図。転写因子は、ある範囲でランダムな運動をしている(上)。輝点の「動く範囲」はクロマチンの凝集と関係し、クロマチンが凝集していなければ動く範囲は大きくなり、凝集度合いに伴い動く範囲は小さくなる(下)。
- (右)NanogとOct4の動く範囲のヒストグラム。Nanogは基底状態から未分化状態で動く範囲が狭くなり、分化誘導後でさらに変化がなく、免疫染色によるクロマチン凝集度合いと一致していた。一方でOct4ではどの状態であっても一定であった。
また、DNAは分化に伴ってさまざまな化学的修飾を受けるため、物理特性が変化します。その物理特性は、転写因子の「運動の速さ」に影響を与えます。1分子観察の結果、運動の速さはNanog、Oct4ともに、条件により差異はありませんでした。ただし、それぞれの発現量と運動の速さには正の相関があり、発現量が高いほどDNAは柔らかいことが分かりました。
NanogやOct4は、同じ標的遺伝子座と相互作用を繰り返します。この相互作用頻度を定量するため、輝点が現れる密度を色の違いで可視化すると、基底状態から未分化状態への変化で高密度な箇所が出現しました(図5)。この現象に着目した1分子計測の解析により、相互作用の平均発生確率(結合頻度)と、標的遺伝子座付近のDNA鎖の流動性に由来する位置移動性(繰り返し結合の範囲)が求められます。
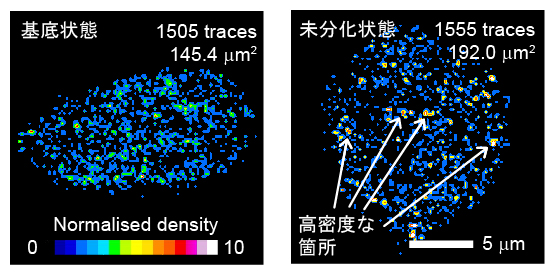
図5 Nanogと標的遺伝子座の結合頻度解析
核内に輝点が現れる密度を擬似カラーで表現した図。暖色系は密度が高いことを示す。基底状態の培養条件では、高密度な輝点は少ないが(左)、未分化状態に移すと高密度な箇所が出現した(右)。
こうして、それぞれの分化条件におけるNanogとOct4の挙動を表す6個のパラメータとして、「発現量」「相互作用時間」「動く範囲」「運動の速さ」「結合頻度」「繰り返し結合する範囲」が求められ、これらのパラメータ間の相関を調べることで、NanogとOct4の違いやそれぞれの働きが見えてきました。そして共同研究グループは、NanogとOct4の作業仮説を提案しました(図6)。
今回、ES細胞の分化が誘導されると、クロマチン凝縮によりDNAが硬くなり、DNAの流動性は減りますが、それと相関して、Nanogと遺伝子座の相互作用時間が長くなることが示されました。すなわち、Nanogの発現量は分化に伴い減少するものの、DNA上に長くとどまることで転写因子としての機能が補償されていると考えられます。このメカニズムは分化多能性の状態を維持し、分化が進み過ぎないように制御するための新しい「負のフィードバック機構」である可能性があります。
パラメータ間の相関を解析したところ、Oct4ではクロマチン凝縮によりDNAが硬くなりその流動性が減ると、発現量が増加する関係が確認されました。また、Oct4が相互作用する遺伝子座周辺のDNAは分化の状態に依らず折り畳まれず、柔らかいことが示されました。このことは、Oct4がクロマチン凝集をほどく働きをするリモデリング因子[11]を遺伝子座に誘導する役割を担うという「パイオニア因子仮説[11]」に矛盾しません。
またES細胞では、Nanogの発現にヘテロジェナイティ[12](不均一性)が確認されています。新しい負のフィードバック機構は、Nanog発現の揺らぎが分化多能性を維持させているモデルであり、これがNanog発現の不均一性の原因である可能性があります。
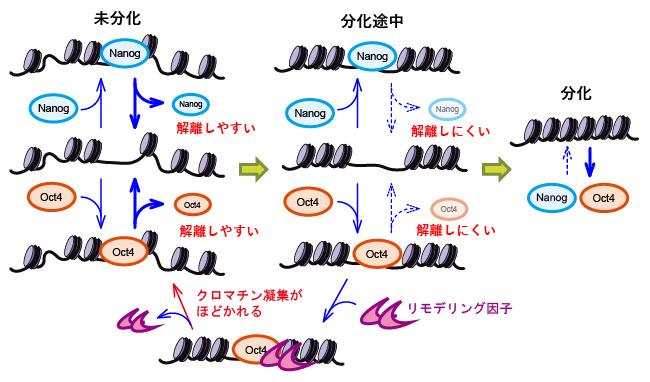
図6 本研究結果から提案されるNanogとOct4の作業仮説「負のフィードバック機構」
共同研究グループは、Nanog、Oct4ともに、クロマチンが凝集すると遺伝子座から解離しにくくなる基本メカニズムの仮説を立てた。分化が進むにつれてクロマチンが凝集するが、凝集に伴いNanog、Oct4が遺伝子座に長く相互作用し、未分化維持に必要な遺伝子の発現を促進させる。これにより、分化速度が低下するように機能する負のフィードバックが形成されている。さらに、Oct4がクロマチン凝集をほどく働きをするリモデリング因子を遺伝子座の近くに誘導することで、クロマチン凝集がほどかれて未分化状態が維持される。つまり、NanogとOct4は協働して、ES細胞の分化多能性の状態を維持し、分化が進み過ぎないように制御している。
- 注1)Tokunaga M, Imamoto N, Sakata-Sogawa K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nat Methods. 5, 159-161 (2008).
今後の期待
共同研究グループは、NanogとOct4は自身の発現量が下がると遺伝子座との相互作用時間を延長する実験結果から、新しい作業仮説を提案しました。この仮説は、転写因子の発現量のみからその転写活性を推定した従来の描像とは逆であり、転写因子の機能メカニズムに新しい知見をもたらすことは間違いありません。
幹細胞を用いる再生医療において、ES細胞やiPS細胞を安定に供給するために、ES細胞やiPS細胞の分化多能性を維持したまま培養することは必須です。本研究成果を基盤に、新しいES/iPS細胞の安定樹立法、安定培養法が開発されることが期待できます。
また、今回の研究で用いた1分子輝点の自動認識アルゴリズムは、細胞核内における1分子計測の解析効率を格段に向上させるため、転写因子の1分子計測が容易となります。この方法が普及すれば、さまざまな転写因子の細胞核内における挙動が明らかになっていくかもしれません。
今後、クロマチンの状態とNanogやOct4の標的遺伝子座との相互作用との因果関係を調査し、Oct4がクロマチン凝縮をほどくのか、あるいはOct4が柔軟なクロマチン領域と優先的に相互作用するだけなのかを決定する必要があります。
補足説明
- 1.ES細胞
胚性幹細胞。ヒトやマウスなどの動物の胚盤胞から単離された細胞。より詳しくは、胚発生の初期段階である胚盤胞期の内部細胞塊より作られる細胞を指す。自己複製能力を持つと同時に、外胚葉、中胚葉、内胚葉のどの胚葉系にも分化できる分化多能性を持つことを特徴とする。ESはembryonic stemの略。 - 2.転写因子
ゲノムDNA上の特定の遺伝子配列(標的遺伝子座)に特異的に結合し、その配列のRNAポリメラーゼによる転写を促進あるいは抑制するタンパク質群の総称であり、転写制御因子ともいう。 - 3.iPS細胞
人工多能性幹細胞。皮膚や血液などから採取した細胞に少数の遺伝子などを導入して作製された多能性幹細胞。ES細胞と同様に分化多能性を持つ。2006年に京都大学山中伸弥教授らにより発明され、2012年に山中教授はその功績によりノーベル医学・生理学賞を受賞した。iPSはinduced pluripotent stemの略。 - 4.クロマチン
細胞核内に存在する染色体の構成要素。DNA、タンパク質、RNAから成る複合体であり、DNAを収納・整理し、細胞の機能を制御している。細胞は、クロマチンの状態を変化させることで、遺伝子の発現を制御する。クロマチンは、主にユークロマチン、ヘテロクロマチンと呼ばれる二つの形態を持つ。ユークロマチンはDNAが緩く巻かれた状態、ヘテロクロマチンはDNAが密に巻かれた状態である。ユークロマチンでは、遺伝子の転写因子がDNAにアクセスしやすいため遺伝子が活発に発現し、一方、ヘテロクロマチンでは、遺伝子の転写因子がDNAにアクセスしにくいため遺伝子の発現が制限されるといわれている。 - 5.蛍光タンパク質
光を吸収し蛍光を発することができるタンパク質群の総称。1960年代に下村脩博士によりオワンクラゲから発見された。この功績は、2008年ノーベル化学賞の受賞対象となった。遺伝子操作により観察したい対象のタンパク質を標識でき、蛍光顕微鏡を用いれば、観察したいタンパク質のみが選択的に画像化できる。 - 6.ゲノム編集技術
生物が持つゲノムDNA上の任意の塩基配列(DNA配列)を編集(削除、挿入、置換)する技術。従来の遺伝子組換えと比べて、安全かつ簡単にDNAを編集できる技術として、研究ツールだけではなく医療・農業・水産業でも広く応用が進んでいる。 - 7.薄層斜光照明法(HILO法)
1分子計測を実現するための顕微鏡法の一つであり、試料表面に斜めからの照明を行うことで、高い解像度とコントラストを実現する手法。通常の蛍光顕微鏡では、背景光が強過ぎて、微弱な蛍光分子一つの信号を検出できない。対物レンズの端にレーザー光を入射させることで、円筒状のレーザー光をつぶして板状に成型しつつ、細胞の側面から照射できる。細胞をシート状の光で「切る」ように照射することで、焦点面以外への励起を防ぎ、背景光が劇的に減少する。 - 8.遺伝子座
染色体上に存在する遺伝子の位置を指す用語で、特定の遺伝子が位置している場所を示す。遺伝子座は、通常、染色体の特定の領域に配置されている。マウスの細胞核には、19対の染色体が格納されており、各染色体に数千から数百万の遺伝子座が存在する。 - 9.白血病抑制因子
細胞から分泌される低分子タンパク質の一つ。細胞の分化を抑制する効果を示す。最初の発見が、骨髄性白血病細胞に対する増殖・分化抑制であったため、「白血病」の名が頭に付くが、白血病特異的ではなくさまざまな細胞に影響を与える。マウスES細胞の分化を阻害するので、ES細胞を継代培養する際に使われる。 - 10.1分子追跡法(SMT)
観察対象となるタンパク質を蛍光分子により標識し、その蛍光分子一つの運動を追跡する手法の総称。SMTはSingle-molecule trackingの略。 - 11.リモデリング因子、パイオニア因子仮説
リモデリング因子とは、クロマチンの構造や形状を変化させるタンパク質を指す。パイオニア因子はリモデリング因子の一つで、転写因子が相互作用できないように凝集したクロマチンを解く機能を持つ。パイオニア因子仮説はパイオニア因子の存在を肯定する一方、近年になり、パイオニア因子は実在せず概念的な存在に過ぎないという主張も見受けられる。 - 12.ヘテロジェナイティ
不均一性。同一空間において、表現型が異なる細胞が混在している様子。例えば、ES細胞では同じ「未分化」という状態であっても、Nanogの発現が高いES細胞と低いES細胞が混在している。
共同研究グループ
理化学研究所
生命機能科学研究センター
先端バイオイメージング研究チーム
チームリーダー 渡邉 朋信(ワタナベ・トモノブ)
(広島大学 原爆放射線医科学研究所 教授)
研究員 佐々木 健介(ササキ・ケンスケ)
技師 塩井 剛(シオイ・ゴウ)
細胞極性統御研究チーム
チームリーダー 岡田 康志(オカダ・ヤスシ)
(東京大学大学院 医学系研究科 教授・同大学院 理学系研究科 兼務)
発生動態研究チーム
チームリーダー 大浪 修一(オオナミ・シュウイチ)
上級研究員 新海 創也(シンカイ・ソウヤ)
バイオリソース研究センター
動物変異動態解析技術開発チーム(研究当時)
チームリーダー 阿部 訓也(アベ・クニヤ)
(現 バイオリソース研究センター 客員主管研究員)
広島大学
原爆放射線医科学研究所
助教 藤田 英明(フジタ・ヒデアキ)
学部生(研究当時)藤本 健太(フジモト・ケンタ)
両生類研究センター
助教 岡本 和子(オカモト・カズコ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、日本医療研究開発機構(AMED)再生医療実現拠点ネットワークプログラム「難治性心筋症疾患特異的iPS細胞を用いた集学的創薬スクリーニングシステムの開発と実践(研究代表者:宮川繁)」、科学技術振興機構(JST)戦略的創造研究推進事業CREST「オールオプティカルメカノバイオロジーの創出に向けた技術開発と発生生物学への応用(研究代表者:倉永英里奈)」による助成を受けて行われました。
原論文情報
- Kazuko Okamoto, Hideaki Fujita, Yasushi Okada, Soya Shinkai, Shuichi Onami, Kuniya Abe, Kenta Fujimoto, Kensuke Sasaki, Go Shioi, Tomonobu M Watanabe, "Single-molecule tracking of Nanog and Oct4 in living mouse embryonic stem cells uncovers a feedback mechanism of pluripotency maintenance", The EMBO Journal, 10.15252/embj.2022112305
発表者
理化学研究所
生命機能科学研究センター 先端バイオイメージング研究チーム
チームリーダー 渡邉 朋信(ワタナベ・トモノブ)
(広島大学 原爆放射線医科学研究所 教授)
細胞極性統御研究チーム
チームリーダー 岡田 康志(オカダ・ヤスシ)
(東京大学大学院 医学系研究科 教授・同大学院 理学系研究科 兼務)
広島大学 両生類研究センター
助教 岡本 和子(オカモト・カズコ)
 岡本 和子
岡本 和子
 渡邉 朋信
渡邉 朋信
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
広島大学 広報室
Tel: 082-424-4383
Email: koho [at] office.hiroshima-u.ac.jp
東京大学 医学部・医学系研究科 総務チーム
Tel: 03-5841-3304
Email: ishomu [at] m.u-tokyo.ac.jp
※上記の[at]は@に置き換えてください。
