理化学研究所(理研)生命機能科学研究センター バイオコンピューティング研究チームのエリオット・ジャコパン 大学院生リサーチ・アソシエイト、海津 一成 上級研究員、高橋 恒一 チームリーダーらの研究チームは、細胞内の代謝経路[1]の挙動(振る舞い)をシミュレーションし、仮想現実(VR)ヘッドセットで可視化するシステム生物学[2]研究向けVRソフトウェア「ECellDive[3]」を開発しました。
本研究成果は、仮想空間(メタバース)での新しい協働的な生命科学研究を提案するものであり、人間には全体像の把握が困難な大規模で複雑な細胞内の分子ネットワークを解明し、生命現象の理解を目指すシステム生物学の発展に貢献することが期待されます。
細胞内では、複数の分子が相互に作用し合い、大規模で複雑なネットワークを形成しています。この生命システムを包括的に理解するためには多様な知識と技術が求められますが、専門分野の異なる複数の研究者が共同で一つのモデルを作り上げることは容易ではありません。
今回、研究チームは、VRデバイスを活用して仮想空間上で代謝ネットワークをシミュレーションし、操作するためのソフトウェアを開発しました。本ソフトウェアは、複数の研究者が仮想空間上で協働しながらモデルを解析し、構築することを可能にします。また本開発で得られた知見を元に、データの追跡性[4]や完全性[4]など、科学研究に求められるメタバースの要件を提案しました。
本研究は、科学雑誌『npj Systems Biology and Applications』オンライン版(1月27日付)に掲載されました。

VRヘッドセットで細胞内代謝ネットワークにダイブ
背景
細胞は生命を形作る最も基本的な単位の一つです。細胞の中には、DNAやRNA、タンパク質などのさまざまな分子が存在し、それぞれが個別に相互作用して複雑な分子ネットワークを形成することで多様な細胞機能を実現しています。「システム生物学」は、細胞機能などの生命現象を分子や遺伝子といった個々の要素ではなく、それらが構成する相互作用ネットワークに着目し、実験生物学と計算科学を統合して、この大規模な生命システムを全体として理解しようという試みです。例えば、オミクス[5]実験は細胞中の分子全体を網羅的に測定し解析することで細胞システムの振る舞いや分子間の相互作用を明らかにします。
しかし、こうした測定に基づいて推定された分子ネットワークは複雑かつ大規模であり、人間の頭ではその振る舞いを正確に予測することは困難です。そこでシステム生物学では、計算機上に分子ネットワークのモデルを構築し、シミュレーションによって振る舞いを予測します。これにより新たな相互作用を発見したり、分子ネットワークを計算機上で設計したりすることもできます。
さらに最近では、遺伝子ネットワークや代謝ネットワーク、シグナル伝達ネットワークのようにこれまで個別に扱われてきたシステムをも統合し、細胞をまるごと一つのシステムとして理解する「全細胞モデリング[6]」という試みが進められています。細胞をこのように包括的に理解するためには、さまざまな対象を研究する研究者や技術者、実験科学者と理論科学者らが協働して一つの複雑で巨大なモデルを作り上げていく必要があります。しかし、異なる領域の研究者はそれぞれが有する基礎知識もさまざまであり、大規模な多次元データの共有も困難です。そこで研究チームは、近年一般にも急速に普及しつつあるVRデバイスを活用し、研究者らがデータベース上の知識やデータ、シミュレーションによる予測の助けを得ながら協働でモデルを作り上げるための仮想空間を開発しました。
研究手法と成果
研究チームは、分子ネットワークの代表例である代謝を対象として、そのモデリング・プラットフォームとなるVRソフトウェア「ECellDive」を開発しました。代謝は細胞内で行われる一連の化学反応で、生命活動に必要な物質やエネルギーをさまざまな酵素分子によって生み出します。代謝は生命にとって不可欠な活動であり、ヒトであれば健康や疾患(代謝疾患)と関連します。また酵母による発酵など、多様な微生物による代謝が有用物質の生産に活用されています。近年、1,000以上の反応から成る微生物の代謝ネットワークに遺伝子改変を加え、さらに培地中の物質条件を変化させることで、目的物質をより効率的に高い生産性をもって作らせるよう設計する「代謝工学」が特に注目されています。その設計過程では代謝ネットワークを構成する分子や遺伝子の情報を含む大量の多次元データ、そしてネットワーク改変によって代謝の振る舞いがどのように変化したかを確認しながら、効率の良い設計を見いだしていく必要があります。研究チームが開発した仮想空間では、VRヘッドセットを用いることで、3次元空間中に代謝ネットワークが可視化され、ユーザーはその中で動き回り、反応経路に改変を加えながらネットワークの各反応速度がどのように変化するのかを直感的に理解することができます。
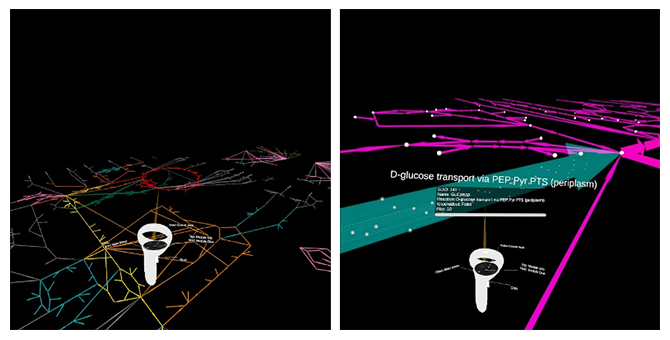
図1 VRアプリケーション上に可視化された代謝ネットワークの例
仮想空間に大腸菌の代謝ネットワークが可視化されている様子(左)。各線は代謝反応を表している。実験やシミュレーションデータに基づいて線の色などを設定することで代謝の振る舞いの変化を可視化できる。ユーザーはVRヘッドセットの操作によって仮想空間内を自由に移動できる。また、対応するオブジェクトを操作することで、データベースから取得したネットワーク中の代謝物質や反応に関する情報を表示させることができる(右)。
システム生物学など研究活動のためのVRソフトウェアや仮想空間では、異なる分野の専門家でも容易に科学的知識を入手できるアクセシビリティ(アクセスのしやすさ)と、それらの知識がどのデータベースに基づいて構築されているかを追跡できるデータのトレーサビリティ(追跡性)、そして高い精度と再現性を備えた正確なシミュレーションが求められます。それに加えて研究チームは、プラットフォームが研究者コミュニティに対して開かれている(誰もが自由にアクセスし、拡張できる)ことも重要だと考えました。
そこで研究チームは、「データの管理」と「データの可視化とインタラクション」を切り離し、既存の生物学データベースと通信し生物学的知識へのアクセスを一元化するサーバーシステム「Kosmogora」と、生物学的知識をナビゲートするVRアプリケーション「ECellDive」の二つに分けて開発を行いました。ECellDiveはKosmogoraを介して既存の代謝データベースから分子や反応に関して得た情報を表示し、分野外の研究者の理解を助けます。また、Kosmogoraは仮想空間において研究者らの知識をサポートするだけでなく、高精度なシミュレーションを行い、研究者が試行錯誤の中で設計したモデルの記録やデータを取り込み、管理します。これにより、データの可視化に加えて、計算コストの高いデータの解析や高精度なシミュレーションをインタラクティブに実行することを可能にします。
現在のバージョンでは、利用することのできるデータベースや機能は限られていますが、こうした機能はモジュール化されているため、将来的にはKosmogoraを拡張することで各研究者や分野に合わせて自由にカスタマイズすることができます。扱うデータについても、代謝ネットワークの表示レイアウトにはグラフネットワーク可視化のためのオープンソースソフトウェアであるCytoscapeのCYJS形式、モデルには生命モデルの国際標準の一つであるシステムバイオロジー・マークアップ言語(SBML)を用いており、システム生物学の既存の研究・開発環境を中心に他のソフトウェアやデータベースとの連携が考慮されています。
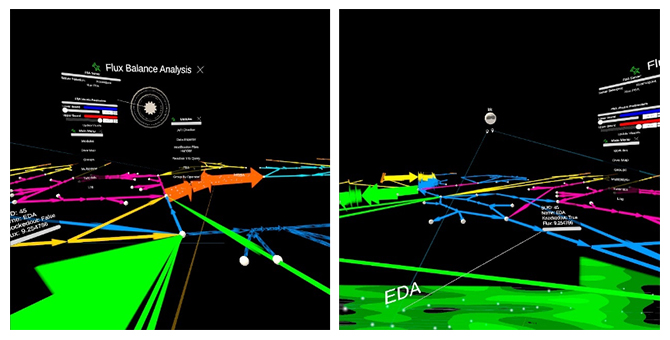
図2 仮想空間内で複数のユーザーが協働している様子
複数のユーザーが仮想空間内で一つの代謝ネットワークを共有できる。代謝流速解析によって代謝の振る舞いを予測し、目前のネットワークに手を加えながらさまざまな設計を検討できる(左)。修正や解析の結果は同じ空間にいる別のユーザー(右図中央)にもリアルタイムで共有され、別のユーザーも同時に設計に手を加えることができる(右)。
VRアプリケーションの利点の一つは、仮想空間内で複数のユーザーがリアルタイムに共通の仮想オブジェクトを操作できることです。ECellDiveの場合では、複数の研究者が共通の代謝ネットワークを設計できるようにしました。ユーザーは他のユーザーとともに、ネットワーク中の遺伝子の欠損(反応の削除)や細胞外の物質濃度条件(反応速度の制限)などの改変を加えることができます。これらの改変が代謝システムの振る舞いに与える結果は、代謝速度や細胞の増殖速度の変化として示されます。こうした改変により得られた設計図はKosmogoraによってその場にいない研究者と共有することもできます。また研究活動には欠かせないデータの追跡性と完全性を実現するため、こうした設計図が元のどのネットワークからどのような改変を加えて得られたか、といった履歴も同時に記録しています。
今後の期待
本研究では、多様な構成要素から成り本質的に大規模かつ複雑な生命ネットワークを取り扱うシステム生物学における研究活動の新たなアプローチとして、VRデバイスを用いた仮想空間(メタバース)の活用が有効であることを示しました。
「メタバース」の定義としてはこれまでさまざまなものが提案されていますが、おおむね「複数のユーザーが参加し、行動できる通信ネットワーク上に作られた仮想空間」を意味します。その中でも特にメタバースを科学的研究活動に活用するためには、データの追跡性、完全性、シミュレーションの精度といった高い基準を満たすデータ管理や、オープン性といった要件を実現する必要があります。
研究チームは本研究を通してこれらを満たすVRアプリケーションの一例を示しました。今後より多くのデータベースやシミュレーション機能を拡張していくことで、研究者を知識の面からサポートし、複雑なネットワークの振る舞いの予測に基づいて、異なる専門分野の研究者が協働で設計を行うことのできる新しい「研究の場」を実現することが期待できます。
また、このアプリケーションは代謝ネットワークを対象としていますが、遺伝子発現制御ネットワークやシグナル伝達ネットワークなど性質の異なる細胞内のさまざまなシステムにも応用することが可能です。さらにこれらのシステムをすべて統合した「トランスオミクス[5]」や「全細胞モデリング」などの、ゲノム・遺伝子・分子・代謝物質の間の関係性を明らかにする、より包括的で大規模な研究が主流になるにつれ、一人の研究者の持ち得る知識や予測能力には限界があるため、本研究のような研究活動をサポートする技術の重要性は今後より高まると考えられます。
本研究チームは現在、細胞のまるごとシミュレーションを目標に汎用細胞シミュレーション環境「E-Cellシステム」を開発しています。このソフトウェアでは詳細な代謝ネットワークの解析や、確率的な遺伝子発現、3次元的な細胞内でのシグナル伝達など、生命現象に合わせて高度な計算技法が利用できます。今回の開発で得られた成果もE-Cellシステムへと統合することで、システム生物学全般にわたって誰もが高性能なシミュレーションを直感的に操作できるような場を提供することを目指しています。
補足説明
- 1.代謝経路
複数の代謝酵素が関与する連鎖的な化学反応のこと。多くの代謝経路は途中で分岐したり、回路を形成したりするなど、複雑なネットワーク構造をとる。 - 2.システム生物学
動的で複雑な細胞が引き起こす現象を定量的および網羅的に測定し、そのデータを数理的に解析することで、そのメカニズムを明らかにすることを目指す研究分野。システムバイオロジーとも呼ばれる。 - 3.ECellDive
Meta Quest2(Meta社)上で動作する。使用イメージは以下の動画の通り。
- 4.追跡性、完全性
ここでは、研究データの履歴を追跡可能にし、一連のデータの正確性が完全に担保されていることを指す。追跡性や完全性は、データ改ざんや偽装を防ぎ、適切な科学研究の遂行に重要である。 - 5.オミクス、トランスオミクス
オミクスとは、生体中に存在するDNA、RNA、タンパク質、代謝産物などの分子種全体やそれらを網羅的に研究する方法論を指し、それぞれの対象によりゲノム(DNA)、トランスクリプトーム(RNA)、プロテオーム(タンパク質)、メタボローム(代謝産物)などの名称がある。トランスオミクスは、これら複数のオミクスデータの解析によってオミクス階層間の相互作用ネットワークを再構築する方法論である。 - 6.全細胞モデリング
遺伝子発現、代謝、ゲノム複製などを含め、一つの細胞を全ゲノム規模でまるごと計算機上にモデル化すること。
研究チーム
理化学研究所
生命機能科学研究センター バイオコンピューティング研究チーム
大学院生リサーチ・アソシエイト エリオット・ジャコパン(Eliott Jacopin)
(大阪大学大学院 生命機能研究科 博士後期課程3年)
研究パートタイマーⅠ 坂本 裕紀(サカモト・ユウキ)
上級研究員 海津 一成(カイヅ・カズナリ)
チームリーダー 高橋 恒一(タカハシ・コウイチ)
生命医科学研究センター 統合細胞システム研究チーム
研究パートタイマーⅠ 西田 孝三(ニシダ・コウゾウ)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究、大学院生リサーチ・アソシエイトプログラム)で実施し、科学技術振興機構(JST)戦略的創造研究推進事業さきがけ「ゲノム配列から自動で全細胞モデリングする技術の開発(研究代表者:海津一成)」、日本学術振興会(JSPS)科学研究費助成事業若手研究(B)「階層を超えて分子構造と細胞機能をつなぐ新規1分子粒度計算技法の開発とその応用(研究代表者:海津一成)」による助成を受けて行われました。
原論文情報
- Eliott Jacopin, Yuki Sakamoto, Kozo Nishida, Kazunari Kaizu, Koichi Takahashi, "An architecture for collaboration in systems biology at the age of the Metaverse", npj Systems Biology and Applications, 10.1038/s41540-024-00334-8
発表者
理化学研究所
生命機能科学研究センター バイオコンピューティング研究チーム
大学院生リサーチ・アソシエイト エリオット・ジャコパン(Eliott Jacopin)
上級研究員 海津 一成(カイヅ・カズナリ)
チームリーダー 高橋 恒一(タカハシ・コウイチ)
 エリオット・ジャコパン
エリオット・ジャコパン
 海津 一成
海津 一成
 高橋 恒一
高橋 恒一
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
