2012年9月6日
独立行政法人理化学研究所
国際プロジェクト「ENCODE」がヒトゲノム機能の80%を解明
―日本から唯一参加の理研OSCは、CAGE法で機能解析に大きく貢献―
ポイント
- 32研究機関442名が参画、最大級の国際プロジェクトからの大規模ゲノム研究成果
- 理研OSCは独自技術のCAGE法による遺伝子の転写開始点解析で貢献
- タンパク質をコードしていないDNA領域でも生命維持に重要な機能保持
要旨
理化学研究所(野依良治理事長)が参加する国際プロジェクト「ENCODE(エンコード)※1」は、5年間をかけて、DNAエレメントデータ※2と呼ばれる遺伝子由来の膨大なデータを収集して解析し、ヒトゲノムの80%の領域に機能があることを明らかにしました。その中で理研オミックス基盤研究領域(OSC:林崎良英領域長)は、独自の遺伝子解析技術CAGE法※3を用いて、DNAからRNAが合成されるときに重要な役割をもつ領域である「遺伝子転写開始点」の解析に貢献しました。これは、理研OSCゲノム機能研究チームのピエロ カルニンチ(Piero Carninci)チームリーダーらによる研究成果です。
本プロジェクトでは、ヒトゲノムの1%の領域が解析対象であったパイロットプロジェクト(2007年6月14日プレスリリース)から大きく発展し、全領域を対象に遺伝子機能の解析を試みました。また、今までゲノム情報として重要視されていたタンパク質合成以外の役割を果たす多様な分子の同定とその機能の解明にも焦点が当てられました。
理研OSCが独自に開発したCAGE法は、ゲノム全体の遺伝子転写開始点の位置とその発現を定量的に調べることが可能です。この技術を用いて約62,000の「遺伝子転写開始点」を同定し、それらのデータは、ヒストン修飾※4や転写因子結合部位とRNA発現の関係の、これまでにない詳細な解析に寄与しました。今回ENCODEが確立した詳細な解析手法は、今後の解析法の標準として貢献することが期待されます。
理研OSCはこれまでに林崎領域長のリードにより国際FANTOMコンソーシアム※5を立ち上げ、CAGE法を開発・活用してゲノム機能の網羅的な解明に大きく貢献してきました。今回のENCODEによるデータは、これまでのFANTOMデータと相補的に働き、疾患におけるゲノムの制御機能などの理解に貢献することが期待できます。理研OSCは今後も医療への貢献を目指し、CAGE法を使って様々な種類の細胞についての解析を進めていきます。国際プロジェクトのそれぞれの成果は、英国の科学雑誌『Nature』はじめ著名な学術誌に合計30本掲載され、アメリカ国立衛生研究所(NIH)やNatureからもプレスリリースを行います。なお、本研究成果は、『Nature』2012年9月6日号に掲載されます。
背景
ヒトゲノム情報は生命体をつかさどる設計図であり、解読することでさまざまな生命の仕組みを解き明かすことが期待できます。しかし、30億塩基といわれるヒトゲノムの機能の多くが謎に包まれたままです。2003年にスタートした国際プロジェクトENCODEは、ヒトゲノムにコードされているすべての機能要素を解明し、複合的に解析することを目指しています。解析に関する転写領域、転写因子結合部位、クロマチン構造※6、ヒストン修飾といった要素をヒトゲノム上にマッピングするためには、機能と塩基配列を関係づけるさまざまなデータが要求されます。そこで、優れたゲノム解析方法を有する5か国(スペイン、アメリカ、イギリス、日本、シンガポール)にまたがる32の研究機関が本プロジェクトに参加して、DNAエレメントデータの収集とその解析に挑みました。その中で理研OSCは、19研究機関と協力して、主に転写に関わる機能解析を解明するためのデータ収集と解析を担当し、独自に開発したCAGE法を用いて転写開始点を網羅的に同定することを目指しました。
研究手法と成果
理研OSCは、Bリンパ芽球様細胞などを含んだ15種のヒト由来細胞のRNAを核由来と細胞質由来に分け、1種類ずつ解析しました。さらに核由来RNAからクロマチン、核質、核小体※7の情報を得るため、このうちの1種(K562細胞株)について、これら3つに分別した解析を行いました。以上の方法により分別された各細胞成分における抽出RNAは、その長さによって200塩基以上のロングと、それ以下のショートに分類しました。さらにロングは、タンパク質のアミノ酸配列をコードしているメッセンジャーRNA(mRNA)※8とそれ以外のRNAとに分けて、これらを対象に塩基配列や転写開始点などの特徴を調べました。転写開始点の同定には、理研OSCで独自に開発されたCAGE法を用いました。
CAGE法で同定された転写開始点(以下CAGEデータ)は、ENCODEの他の研究機関から得られたヒストン修飾や転写因子結合データ、プロモーターにおける転写活性との詳細な解析、そして末端エンハンサー領域※9などのデータと総合して解析し、ヒストン修飾や転写因子結合部位データと転写活性の関係を予測するモデルを構築しました。(図1)。
また、CAGEデータのうち18%は繰り返し配列と重なりました。この頻度は遺伝子内領域における転写開始点のマッピングに比べ明らかに高いこと、つまり繰り返し配列の転写活性がより高く、ある特徴的な領域に偏って存在しており、なんらかの機能を有していることが示唆されます。さらに、CAGEデータは、たとえわずかな量であっても、細胞特異的に転写効率を高めるエンハンサー領域を示すことできるため、これまで難しかったその特徴解析を可能にしました(図2)。
今後の期待
ENCODEにより、転写活性に影響するヒストン修飾と転写の関係、エンハンサー領域と転写活性との関係をはじめとするヒトゲノムの80%の生物的機能が明らかになりました。この結果は、ヒトゲノムの機能のさらなる解析を可能にする質と量を備えた貴重なデータベースです。今後、このデータベースは、さまざまな角度からヒト疾患をはじめとする生命現象解明に有効に活用されることが期待できます。理研OSCでは、今後もこのような大規模データベースの整備を進めていくことを計画しています。
原論文情報
- The ENCODE Project Consortium, “An integrated encyclopedia of DNA elenments in the human genome”. Nature, 2012. doi: 10.1038/nature11233
- S. Diebali., et. al, “Landscape of transcription in human cells” Nature, 2012. doi: 10.1038/nature11247
発表者
理化学研究所
オミックス基盤研究領域 LSA要素技術開発グループ ゲノム機能研究チーム
チームリーダー ピエロ カルニンチ
お問い合わせ先
横浜研究推進部 企画課
Tel: 045-503-9117 / Fax: 045-503-9113
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.ENCODE(エンコード)
The Encyclopedia of DNA Elements。アメリカ国立ヒトゲノム研究所(NHGRI)が2003年に立ち上げたヒトゲノム解析プロジェクト。ヒトゲノムのすべての機能要素の解析を目指している。世界5か国(スペイン、アメリカ、イギリス、日本、シンガポール)から32の研究機関が参加している。日本からは唯一理化学研究所オミックス基盤研究領域が参加している。
The ENCODE Project: ENCyclopedia Of DNA Elements(英語) - 2.DNA エレメントデータ
ENCODEプロジェクトでは、ヒトゲノム解析に用いた各機能的塩基配列をDNAエレメントデータと称している。この中に含まれるものは、ある生成物(タンパク質、ノンコーディングRNAなど)をコードする配列や再現性をもつ生物的機能性(タンパク質結合、特異的クロマチン構造など)配列がある。 - 3.CAGE法
Cap Analysis of Gene Expressionの略。理研オミックス基盤研究領域が開発した方法で、耐熱性逆転写酵素やcap捕捉法を組み合わせて転写物の5’末端の塩基配列を決定する実験技法。この塩基配列を読み取ってゲノム配列と照らし合わせて、どこからどこまでコピーが始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる。 - 4.ヒストン修飾
ヒストンはDNAとの親和性の高い塩基性タンパク質。主に4種のヒストン(2A,H2B,H3,H4)がそれぞれ2分子ずつ集まって8量体を形成し、この周りにDNAが巻き付いてクロマチン構造の最小単位‘ヌクレオソーム’を形成する。ヌクレオソームからは各ヒストンのアミノ酸末端領域が突出しており、アセチル化、メチル化などの修飾を受けて、クロマチン構造が弛緩、凝縮し、遺伝子発現が制御される。 - 5.国際FANTOMコンソーシアム
2000年に、理研ゲノム科学総合研究センター 遺伝子構造機能研究グループ(現・オミックス基盤研究領域)が中心となって結成した。哺乳動物(マウス)の遺伝子を網羅的に機能注釈することを主眼とする国際的研究コンソーシアム共同集団(Functional ANnoTation Of Mammalian genome)の略称。活動範囲は拡大され、2009年には遺伝子ネットワークの解明に成功。現在は様々な種類の細胞の遺伝子ネットワーク解析に取り組んでいる。現時点で、18カ国100機関以上が参加している。 - 6.クロマチン構造
真核生物のゲノムDNAは、ヒストンやそれ以外のタンパク質と結合し、高度に凝縮した状態で存在する。この構造をクロマチン構造と呼ぶ。局所的なクロマチン構造の変化により、転写因子などのタンパク質が染色体DNAに接近しやすさを制御する。 - 7.クロマチン、核質、核小体
クロマチン、核質、核小体は、細胞内核に存在する。クロマチンとは、真核細胞内に存在するDNAとタンパク質の複合体のことを表す。核質もしくは核原形質とは、核膜に包まれている原形質(液体)の総称であり、DNA複製等に必要なヌクレオチドをはじめとする多くの物質や、細胞核の中で直接作用する酵素などが溶解している。核小体(かくしょうたい)は、真核生物の細胞核の中に存在する、分子密度の高い領域で、rRNAの転写やリボソームの構築が行われる場所。核内に存在するRNAの分布は、この3種間で異なると考えられるため、それぞれの器官に分布するRNAを調査し、このRNAの由来であるゲノム領域を特定することで、ゲノム機能解明の手がかりとなる可能性が示唆される。 - 8.完全長メッセンジャーRNA(mRNA)
mRNAの5‘末端であるメチル化キャップ構造から反対側(3’)の末端であるポリアデニル化末端までの長さを持つmRNA。タンパク質のアミノ酸配列をコードしている。 - 9.エンハンサー領域
遺伝子の数10k塩基上流や下流に位置し、隣接遺伝子の転写効率を変化させるDNAの特定の配列のうち、転写効率を著しく高める部分をエンハンサー領域(配列)という。
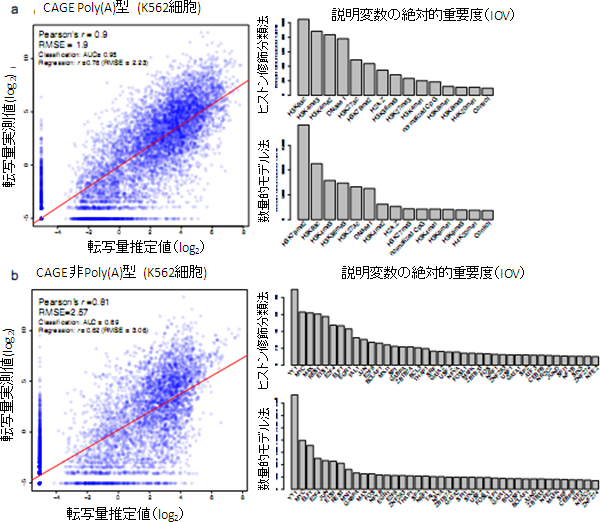
図1 ヒストン修飾と転写因子結合とそれぞれの転写活性の関係
- (a)
- 左グラフ:ヒストン修飾から予測された転写配列(横軸)とCAGEデータによる転写活性の実測値(縦軸)
ヒストン修飾から予測される転写活性と実際の転写量に相関があることからこの転写活性予測モデルの妥当性が示唆される。 - 右グラフ:ヒストン修飾のタイプのうち、より転写活性に関与していると予測されたもの(棒グラフの高さは説明変数の相対的重要度(IOV)を示す)。
上段はヒストン修飾の分類による重要度推定、下段は数量的モデルによる重要度推定を示す。このデータが転写量実測値との相関性解析に用いられる。
- 左グラフ:ヒストン修飾から予測された転写配列(横軸)とCAGEデータによる転写活性の実測値(縦軸)
- (b)
- 左グラフ:DNAへの転写因子結合パターンから予測された転写配列(横軸)とCAGEデータによる転写活性実測値(縦軸)
転写因子結合パターンから予測される転写活性と実際の転写量に相関があることからこの転写活性予測モデルの妥当性が示唆される。 - 右グラフ:転写因子結合パターンのうち、より転写活性に関与していると予測されたもの(棒グラフの高さは説明変数の相対的重要度(IOV)を示す)。
上段は転写因子の分類による重要度推定、下段は数量的モデルによる重要度推定を示す。このデータが転写量実測値との相関性解析に用いられる。
- 左グラフ:DNAへの転写因子結合パターンから予測された転写配列(横軸)とCAGEデータによる転写活性実測値(縦軸)
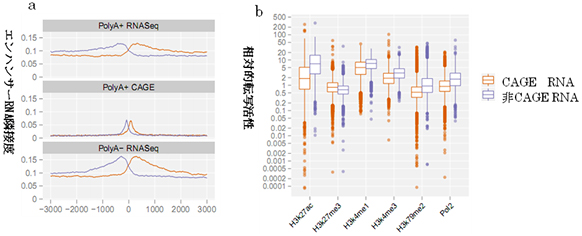
図2 エンハンサー領域における転写
- (a)
エンハンサー領域付近のRNAパターン
赤はプラス鎖(DNAの5’末端から3‘末端の方向に写し取られているもの)
青はマイナス鎖(DNAの3’末端から5‘末端の方向に写し取られているもの)
横軸:エンハンサー中心部からの相対的距離、縦軸:エンハンサーRNA隣接度。
3つの図は、上からそれぞれポリアデニル化RNA配列、CAGEデータ、ポリアデニル化していないRNA配列によるエンハンサーポジションの予測。
エンハンサーの中心(図ではポジション0)に近づくにつれ、発現が活性化されると考えられるため、エンハンサー付近のRNA配列の発現頻度によりエンハンサー領域が予測される。 - (b)
エンハンサー領域付近のクロマチン状態
赤はCAGEデータ以外のRNA、青はCAGEデータ(転写開始部位)
RNAポリメラーゼII結合部位(POL2)は、転写開始部位(CAGEデータ)と考えられるが、ヒストン修飾による転写活性化については、CAGEデータの方が必ずしもより頻度が高いわけではなく、さまざまなパターンが見られる。
