ポイント
- 細胞形態の変化パターンをデータベース化した「モルフォベース」を構築
- さまざまな薬剤の作用をモルフォベースで分類、形態変化と薬理作用を関連付け
- モルフォベースを活用し新規有用物質の標的分子や既知薬剤の副作用を予測
要旨
理化学研究所(野依良治理事長)は、さまざまな抗がん剤をがん細胞に添加することで起きる細胞形態の変化パターンをデータベース化した「モルフォベース」を構築しました。さらに、モルフォベースの特徴や情報を基に新規抗がん剤の作用を予測する手法「モルフォベースプロファイリング」を開発しました。これは、理研基幹研究所(玉尾皓平所長)長田抗生物質研究室の長田裕之主任研究員、化学情報・化合物創製チーム 二村友史特別研究員、化合物ライブラリー評価研究チーム 川谷誠専任研究員らの研究グループによる成果です。
優れた観察者は細胞の「かたち(形態)」を見ただけで、細胞内の変化について多くの情報を得ることができると言われています。実際、細胞の形態変化を指標に発見されたラクタシスチン※1は再発性多発性骨髄腫の治療薬開発の足がかりになりました。これは、形態観察という原始的な方法が未だ有益な創薬手法のひとつであることを物語っています。しかし、形態という定性的な現象を頼りにした活性評価法は煩雑で時間がかかり、また観察者の主観に全面的に依存してしまう危険性もはらんでいます。そのため、細胞の形態変化を定量化し客観的に判断、解析できる技術が求められていました。
研究グループは、薬剤を細胞に添加して出現する重要な形態変化の判断基準を作るため、60種類の既存抗がん剤を研究用がん細胞の一種であるsrcts-NRK細胞※2に用いて変化する形態を観察しました。その結果、添加した抗がん剤によって多種多様な形態変化を示すこと、また同じ標的分子をもつ薬剤は似た形態変化を誘導することが分かりました。同時にイメージングサイトメーター※3を使って客観的に細胞形態変化を評価し、薬剤作用と細胞形態に関する複数の情報を定量的に関係付けた形態変化データベース「モルフォベース」を構築しました。さらに、モルフォベースを活用することで、誰もが特徴的な形態変化から簡単に薬剤の作用メカニズムを予測できる手法「モルフォベースプロファイリング」を開発しました。
このような細胞形態変化データベースを基にした作用解析手法は、化合物を作用メカニズム別に分類し、新規有用物質の標的分子を迅速に同定、また副作用も予測できることから、抗がん剤創薬を一層加速するものと期待できます。
本研究成果は、米国の科学雑誌『Chemistry & Biology』オンライン版(12月20日付け:日本時間12月21日)に掲載されます。
背景
がんの原因分子だけを狙い撃ちにする分子標的薬を開発するには、抗がん剤の候補化合物がどのようながんに対して有効で、なぜ効果を示すのか、また副作用はないか、といった作用メカニズムを開発早期から知る必要があります。近年発展したポストゲノム生命科学研究では、遺伝子やタンパク質の網羅的な変動解析(オミックス※4)を基盤に薬剤の作用メカニズムを探る研究が盛んに行われています。2010年に研究グループも、データベースとの照合により薬剤の生体内標的分子を予測する手法「プロテオームプロファイリング法※5」を開発しました(2010年5月28日プレスリリース)。その一方で、特殊な実験技法を必要とせず、より簡便に薬剤の作用メカニズムを知る方法についても模索していました。
研究グループは、研究用がん細胞の一種であるsrcts-NRK細胞を用いた実験を行っていたときに特徴的な形態変化を誘導する薬剤を偶然見いだしました。また、この細胞の形態は添加した薬剤の作用に応じて規則的に変化していることにも気づきました。これをきっかけに、顕微鏡で観察される形態変化から簡単に薬理作用を予測することを目指して、作用メカニズムが明らかな薬剤を網羅的に評価し、形態変化と作用メカニズムとを対応づけた細胞形態変化データベースの構築を試みました。
研究手法と成果
研究グループはまず、作用メカニズムがよく分かっている60種類の抗がん剤を添加したときのsrcts-NRK細胞の形態を顕微鏡で観察し、形態変化パターンによる分類を行いました。その結果、アクチンや微小管などの細胞骨格に作用する抗がん剤は作用ごとに特徴的な形態変化を誘導しており、これを容易に判別することができました。また、細胞骨格とは関連のない高分子合成阻害剤や熱ショックタンパク質90(HSP90)、プロテアソームを阻害する抗がん剤なども、それぞれ独特な形態変化を誘導していたため、目視でその作用を推測することができました(図1)。
こうした顕微鏡での形態観察は非常に多くの情報を与えてくれる一方で、観察者の技量に左右されがちな欠点もあります。そこで研究グループは、誰もが同じように解析できるようにするため、細胞形態をイメージングサイトメーターで定量化することを目指しました。また、薬剤の種類数や作用予測の精度を上げるために、srcts-NRK細胞と同様に特徴的な形態変化を示すヒト由来培養細胞のHeLa細胞※6の形態変化データも加えました。
まずイメージングアルゴリズムの開発を行い、コンピューター上で微細かつ複雑な細胞形態を認識できるようにしました。次に、薬剤が誘導するさまざまな形態変化を特徴づけるため、細胞質や核、薬剤添加の影響で生じる顆粒や液胞などの構造体についてその大きさや数、扁平率など12種類のパラメータを設定しました(図2 ステップ1)。さらに、207種類の作用既知薬剤によって誘導されるsrcts-NRK細胞とHeLa細胞の形態変化をそれぞれ数値化し、得られた数値を計71次元の座標に変換した統計値で分析しました。すると、類似の作用を示す作用既知薬剤群は近傍に位置し14種類のクラスターを形成しました(図2 ステップ2)。すなわち、細胞や細胞小器官の形状、細胞内タンパク質の挙動など複数のパラメータを一挙に定量化することで薬剤作用と形態変化とを定量的に関係付けることができました。このように蓄積した形態変化の情報をデータベース化し、「モルフォベース」を構築しました。
次に新規の抗がん剤候補物質の作用を予測するため、その候補物質がモルフォベース内のどの作用既知薬剤と類似するかを計算できるプログラムを作りました。具体的には①候補物質と各作用既知薬剤の類似度のランキング②14種類のクラスターに分類された54種類の典型的な抗がん剤のデータを利用し、候補物質がどのクラスターに分類されるかのスコア化、の2通りの方法で類似性を計算し、形態変化パターンから薬剤作用を予測する手法「モルフォベースプロファイリング法」を開発しました(図2・ステップ3)。
実際に研究グループは、理研が保有する天然化合物バンクNPDepo※7の化合物ライブラリーから抗がん剤候補物質NPD6689を選び、今回開発したモルフォベースプロファイリング法を駆使して、その標的分子を明らかにすることを試みました。その結果、NPD6689は微小管作用薬のクラスターに分類され、微小管を標的としていることが予測できました(図3A)。推定した効果を試験管あるいは細胞レベルで実験的に検証したところ、NPD6689は確かに微小管に作用し、細胞骨格を破壊していることが分かりました。また副作用を引き起こすことが知られているミトコンドリア呼吸鎖阻害剤rotenone(ロテノン)や細胞周期阻害剤3-ATAについてモルフォベースへ照合しました。すると、主な作用メカニズムとは異なる微小管作用やDNA合成阻害活性を有することが予測されました(図3B)。つまり、モルフォベースはこれら薬剤の副作用についても正確に予測することができました。
今後の期待
研究グループは、より精度が高いプロファイリングシステムの構築を目指し、現在も新たな作用既知薬剤や遺伝子ノックダウンが誘導する形態変化を収集・登録してデータベースを拡充しています。モルフォベースプロファイリング法は、新規候補薬剤の作用メカニズムをよりシンプルな方法で高精度かつ迅速に予測するツールとして、今後抗がん剤創薬研究に広く活用されることが期待できます。また発想を転換すると、モルフォベースに照合されない形態変化を誘導する薬剤はこれまでに知られていない作用メカニズムをもったものといえます。それらをNPDepoや微生物代謝産物より探索し、”first-in-class”(新規標的分子に作用する画期的新薬)の発見を目指してさらなる研究を進めることが可能です。
原論文情報
- Futamura Y., Kawatani M., Kazami S., Tanaka K., Muroi M., Shimizu T., Tomita K., Watanabe N., Osada H.
"Morphobase, an encyclopedic cell morphology database, and its use for drug target identification."
Chemistry & Biology, 2012, doi: 10.1016/j.chembiol.2012.10.014
発表者
理化学研究所
基幹研究所 長田抗生物質研究室
主任研究員 長田 裕之(おさだ ひろゆき)
特別研究員 二村 友史(ふたむら ゆうし)
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.ラクタシスチン
神経芽腫Neuro2A細胞に分化を誘導する化合物。放線菌代謝産物より発見され、その標的分子はユビキチン化タンパク質分解酵素20Sプロテアソームである。プロテアソーム阻害剤全般は、がん細胞選択的に細胞死や細胞周期停止を誘導する。そのため、プロテアソームは有効ながん分子標的と認識され、ベルケード(velcade®)が再発性多発性骨髄腫の治療薬として開発された。 - 2.srcts-NRK細胞
ラット腎細胞由来正常繊維芽細胞NRKを親株とするがん遺伝子v-srcの温度感受性株で研究用に用いられている。許容温度(32℃)で培養すると、 v-Srcが活性化してがん細胞形質を示す。v-Srcはニワトリにがんを作るラウス肉腫ウイルスから見いだされた世界最初のがん遺伝子。 - 3.イメージングサイトメーター
細胞のさまざまな現象を高速にイメージングし、細胞1個ずつを定量解析する測定装置。一度に得られる大量の数値情報から、細胞集団の様子を統計的に理解することが可能。 - 4.オミックス
2003年にヒトゲノムが解読されて以来、遺伝子の網羅的な発現情報(トランスクリプトーム)や細胞内のタンパク質(プロテオーム)、細胞内の代謝物(メタボローム)などさまざまな種類の分子情報の網羅的収集・解析が急速に発展してきた。このような生物の体の中にある分子全体を網羅的に調べる学問をオミックスという。 - 5.プロテオームプロファイリング法
この方法では、処理した薬剤の作用に応じて細胞が固有のタンパク質変動(プロテオーム)を誘導する性質を利用する。研究チームは41種類の作用既知薬剤がHeLa細胞に対して誘導するプロテオームデータを取得し、データベース化している。新規有用物質の作用メカニズムは、その物質が誘導するプロテオームパターンをデータベースと照合することにより予測できる。 - 6.HeLa細胞
世界で初めて樹立されたヒトの細胞株。子宮頸がん由来の細胞で、世界中でさまざまな研究に利用されている。名前は、患者である黒人女性の名前(Henrietta Lacks)に由来する。 - 7.天然化合物バンクNPDepo
理研・ケミカルバイオロジー研究基盤施設は、天然物やその誘導体を網羅的に収集した化合物バンクNPDepo(http://npd.riken.jp/)を2007年に創設した。NPDepoでは独自に発見した化合物を保管するとともに、大学や企業から化合物を寄託してもらい、多様性ある化合物ライブラリーを構築している。同時に、化合物の化学・生物活性情報を収載したデータベースNPEdiaを整備し、インターネットで公開している。
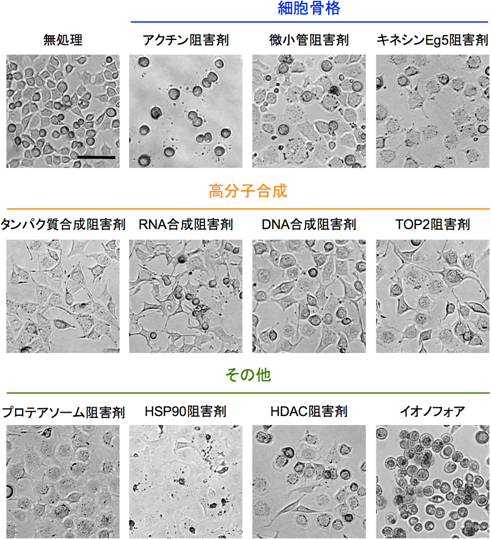
図1 srcts-NRK細胞は薬剤の作用に応じて多彩な形態変化を示す
srcts-NRK細胞に60種類の抗がん剤を添加し48時間後に細胞の様子を観察した。形態変化の類似性で分類したところ、作用メカニズム(標的分子)が同じ薬剤は類似の形態変化を示した。ここに示す11パターンの形態は細胞骨格や高分子合成などに作用する典型的な抗がん剤の表現型である。
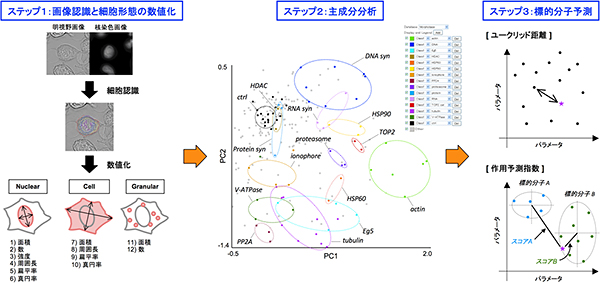
図2 モルフォベースプロファイリングによる標的分子予測の流れ
- ステップ1:作用既知薬剤を添加したsrcts-NRKとHeLa細胞についてイメージングサイトメーターで細胞画像を取得し、独自のアルゴリズムで細胞の核、細胞質、薬剤を添加した影響で生じる顆粒や液胞などの構造体を認識する。また認識した細胞成分について12種のパラメータを数値化する。
- ステップ2:各作用既知薬剤について約1,000個の細胞を数値化し、計71次元の統計値を主成分分析する。第1主成分(PC1)と第2主成分(PC2)を座標軸にし、207種類の作用既知薬剤の主成分得点をプロットした図を示した。標的分子が同じ薬剤はこの主成分得点プロット上で近傍に位置し、14種のクラスター(点線で図示)を作ることが分かる。
- ステップ3:抗がん剤候補物(★印)の作用は、①作用既知薬剤との距離を基にした類似度ランキング②14種の標的分子クラスターのうちどれに分類されるかをスコア化して類推する。
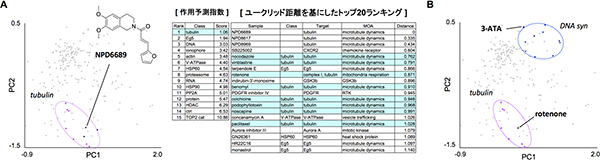
図3 モルフォベースプロファイリング法で化合物の標的分子や副作用を予測
- A: 新規の抗がん剤候補物NPD6689が誘導する形態変化を数値化し、モルフォベースプロファイリング法で解析した結果を示した。作用予測指数やトップ20ランキングからNPD6689の標的分子は微小管であることが類推された。
- B: ロテノンと3-ATAの形態変化データをモルフォベースプロファイリング法で解析した結果を示した。ロテノンは微小管作用を有すること、3-ATAはDNA合成を阻害することがわかり、副作用に関する標的分子、作用メカニズムについても正確に予測できた。
