要旨
理化学研究所(理研)環境資源科学研究センター合成ゲノミクス研究グループの松井南グループディレクターらの研究グループ※は、2014年6月に公開したソルガム(モロコシ)の完全長cDNA(相補的DNA)[1]データベース「MOROKOSHI」の全データを解析し直すとともに、共発現ネットワークを可視化したグラフを加え、トランスクリプトーム[2](全RNA)データベースとして新たに公開しました。
ソルガムは主要な穀物の1つであり、乾燥や多雨、高温、塩害といった環境ストレスに強い植物です。また、収量も多いため、バイオプラスチックやバイオエタノール原料として注目されており、2009年には全ゲノム配列が解読されています。ソルガムの生産性や環境耐性をより向上させるには、ゲノム配列の機能注釈(アノテーション)や完全長cDNAなどのリソースが整備されたデータベースが必要ですが、そのようなデータベースは今までありませんでした。
研究グループは、できるだけ多くの遺伝子に関する完全長cDNAのデータを得るために、葉や花などさまざまな器官、さまざまな育成段階(8段階)からメッセンジャーRNA(mRNA)を採取し、完全長cDNAライブラリを作成しました。次にそれを基に、約4万種の完全長cDNAクローンの塩基配列を決定し、重複しているものを除いた結果、約1万個の遺伝子を同定しました。また、遺伝子の塩基配列がmRNAへと転写される際の制御解析に重要となる転写開始点を新たに約2万カ所決定しました。研究グループは、これらの情報をまとめ、ソルガムの完全長cDNAデータベース「MOROKOSHI」として2014年6月に公開しました。
そして今回、全データを解析し直すとともに、共発現ネットワークを可視化したグラフを加え、トランスクリプトームデータベースとして新たに公開しました。次世代シーケンサ技術(超高速塩基配列解読装置)を活用したトランスクリプトーム解析からは、独自のデータと公開データの計26サンプルデータを使うことで、多様な条件・器官における各遺伝子の発現パターンを解析・公開しています。同じ代謝系にある遺伝子群は、発現パターンも類似していると期待できるため、既存の代謝系遺伝子を中心とした包括的な共発現解析[3]により、新規遺伝子の同定に寄与することを目的としています。
「MOROKOSHI」は、ソルガム研究はもちろん、その近縁のサトウキビやトウモロコシ研究にも利用できるほか、バイオマス研究を加速する基盤のデータベースとして利用が期待できます。また、完全長cDNAクローンは理研バイオリソースセンターから配布しています。
本研究は、日本植物生理学会の学術誌『Plant and Cell Physiology』(1月号)に掲載されました。
※研究グループ
理化学研究所 環境資源科学研究センター バイオマス工学研究部門 合成ゲノミクス研究グループ
グループディレクター 松井 南(まつい みなみ)
研究員 蒔田 由布子(まきた ゆうこ)
研究員 嶋田 勢津子(しまだ せつこ)
背景
ソルガム(Sorghum bicolor)は、サトウキビやトウモロコシと近縁なアフリカ原産の穀物です。高粱(コウリャン)やモロコシとも呼ばれ、栽培面積は穀物の中で、小麦、イネ、トウモロコシ、大麦に次いで第5位です。乾燥や多雨、高温、塩害といった環境ストレスに強く、ほかの農作物の栽培ができない土地でも栽培できます。また収量も多いため、バイオプラスチックやバイオ燃料の原料として注目されています。
2009年にソルガムの全ゲノム配列が解読されました。生産性や環境耐性をより向上させるにはゲノム配列の機能注釈(アノテーション)や、完全長cDNAなどのリソースを整備したデータベースが必要ですが、そのようなデータベースは今までありませんでした。
完全長cDNAは、メッセンジャーRNA(mRNA)の完全なコピーであり、タンパク質の合成に必要な遺伝情報を備えています。特に、遺伝子の発現調節に重要な手がかりとなる転写開始点情報と、同じ遺伝子領域から複数種の成熟mRNAをつくるスプライシングバリアント[4]情報を得ることができます。これらの情報は有用遺伝子の探索や遺伝子機能を解明する上で必要不可欠な基礎情報で、近縁種であるサトウキビやトウモロコシの研究を進める上でも重要な情報となります。
また、大規模に遺伝子の塩基配列が同定された後には、それらの機能推定も必要となります。
そこで研究グループは、ソルガムの完全長cDNAを大規模に解析するとともに、全ての遺伝子において共発現解析を行なうことで、発現パターンが類似する遺伝子から、機能未知遺伝子の機能の推定ができるようなデータベースの構築を目指しました。
研究手法と成果
研究グループは、ゲノムの全塩基配列が2009年に解読されたソルガムの品種「BTx623」を利用しました。できるだけ多くの遺伝子に関する完全長cDNAのデータを得るために、この品種の根を除く茎や葉、花、種子などさまざまな器官、さまざま育成段階(8段階)のmRNAを採取し、完全長cDNAライブラリを作成しました。そこから約4万の完全長cDNAクローンの塩基配列を決定しました。重複しているものを除くと、約1万個の遺伝子が含まれていました。ソルガムの全遺伝子は約3万個あるといわれているので、その1/3に相当します。また、遺伝子の塩基配列がmRNAに転写される際の正確な転写開始点を新たに約2万カ所決定しました。このデータは、転写調節に関する新しい要因を探索する上で重要な手がかりとなります。研究グループは、これらの情報をまとめ、ソルガムの完全長cDNAデータベース「MOROKOSHI」として2014年6月に公開しました(図1)
そして今回、全データを解析し直すとともに、共発現ネットワークを可視化したグラフを加え、トランスクリプトームデータベースとして新たに公開しました。共発現ネットワークの解析には、次世代シーケンサ技術(超高速塩基配列解読装置)を利用したトランスクリプトームデータを利用しました。研究グループでは、デンプンを蓄積していく過程にある種子の発育段階2点と、比較対象として茎のトランスクリプトームデータを取得しました。さらに他の研究機関から論文発表されている全てのトランスクリプトームデータを合わせた計26のサンプルデータを統合することで、より高い精度での共発現解析情報を提示できるようにしました。同じ代謝系にある遺伝子群は、発現パターンも類似していると期待できるため、既存の代謝系遺伝子を中心とした包括的な共発現解析により、新規遺伝の同定が見込まれます。
新たに公開した「MOROKOSHI」では、発現パターンの類似する上位20遺伝子について、発現量の折れ線グラフやネットワーク図も表示しています(図2)。また、それぞれの遺伝子が属する経路情報もまとめています。
今後の期待
ソルガム完全長cDNAの大規模収集は、世界のソルガム研究や近縁種のサトウキビやトウモロコシ研究に貢献できる成果といえます。また完全長cDNAのデータと共発現データを提供するデータベース「MOROKOSHI」を活用することで、効率よく有用遺伝子の探索を進めることができるため、バイオマス生産能のより高い品種の開発につながると期待できます。
完全長cDNAクローンは理研バイオリソースセンターから配布しています。
原論文情報
- Yuko Makita, Setsuko Shimada, Mika Kawashima, Tomoko Kondou-Kuriyama, Tetsuro Toyoda, Minami Matsui, "MOROKOSHI: Transcriptome Database in Sorghum bicolor.", Plant and Cell Physiology, doi: 10.1093/pcp/pcu187
発表者
理化学研究所
環境資源科学研究センター バイオマス工学研究部門 合成ゲノミクス研究グループ
グループディレクター 松井 南(まつい みなみ)
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.完全長cDNA(相補的DNA)
cDNAとは、ゲノムDNAのうちタンパク質をコードする塩基配列のみの遺伝情報物質であるmRNA(メッセンジャーRNA)を鋳型にして作られたDNAのこと。cDNAはmRNAから作られるが、その際mRNAの全ての領域をカバーできずに不完全なものになることが多い。完全長cDNAは、mRNAの全領域をカバーするように作られているため、その配列情報からは遺伝子の完全な構造(塩基配列)が分かり、そのクローンを翻訳してタンパク質を合成したり、遺伝子機能を細かく調べたりすることができる。 - 2.トランスクリプトーム
細胞内の全DNAの塩基配列情報をさす「ゲノム」に対し、細胞内の全転写産物(全RNA)をトランスクリプトームと呼ぶ。 - 3.共発現解析
生物は多様な状況下に適応するため複数遺伝子が同時に働く。それら機能的なパートナーである遺伝子群は、さまざまな条件下で遺伝子発現のON/OFFのパターンが類似してくる。この仕組みを利用し、同じ発現パターンを示す遺伝子群から機能的パートナーを類推する方法。 - 4.スプライシングバリアント
遺伝子は、アミノ酸に翻訳されるための情報を持つ配列「エキソン」とそのような情報を含まない配列「イントロン」が交互に並んでできている。イントロンを除去しエキソンのみを切り出す過程をスプライシングと呼ぶ。その際、複数エキソンの組み合わせにより、同じ場所から異なる配列(成熟mRNA)をつくることができる。この多様な成熟mRNAをスプライスバリアントと呼ぶ。

図1 ソルガムのトランスクリプトームデータベース「MOROKOSHI」
MOROKOSHI Sorghum transcriptome database(英語)
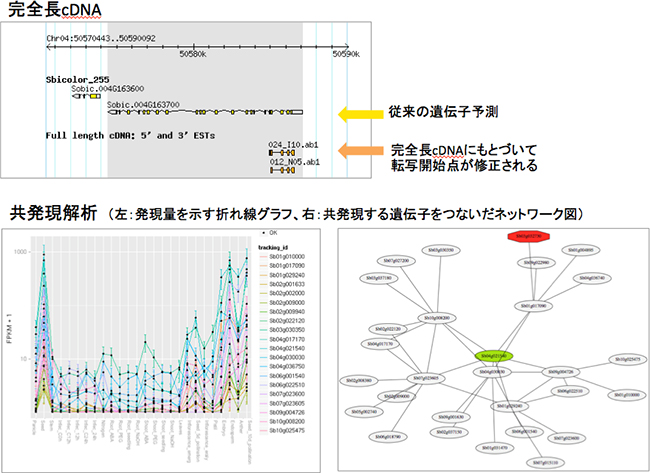
図2 MOROKOSHIデータベースの内容例
- 上段:完全長cDNAのマッピング結果。
- 下段:共発現解析の結果例(左:類似発現パターンを示す遺伝子群の折れ線グラフ。右:共発現遺伝子をつないだ遺伝子ネットワーク。緑:該当遺伝子、赤:転写因子)。
