要旨
理化学研究所(理研)ライフサイエンス技術基盤研究センタートランスクリプトーム研究チームのピエロ・カルニンチ チームリーダーと橋本浩介専任研究員らの研究チームは、がん組織および周辺組織におけるB型肝炎ウイルス(HBV)の遺伝子発現パターンを明らかにし、これまで知られていなかった転写開始点を発見しました。
HBVは肝臓がんの主要な原因であり、世界中で年間約68万6,000人がこのウイルスに起因する疾患で死亡していると推定されています注1)。治療にはウイルスの増殖を抑制する複数の抗ウイルス薬が使用されていますが、ウイルスを体内から完全に排除することはできず、慢性的な肝臓疾患の原因となっています。HBVのゲノムはコンパクトで、遺伝子が四つしかないにも関わらず、がん組織中におけるウイルスの詳細な遺伝子発現パターンはこれまで知られていませんでした。
今回、研究チームはCAGE法[1]を用いて、肝臓がんの一種である肝細胞がん[2]の組織、がん化していない周辺組織、血液、および4種類の実験モデルシステムにおけるウイルスの遺伝子発現パターンを解析し、1塩基単位の詳細な転写マップ[3]を作成しました。これにより、ウイルスの増殖に重要な「X遺伝子」上にこれまで知られていなかった転写開始点を発見し、異なるサイズのタンパク質が作られていることが分かりました。また、実験モデルシステムと実際の肝臓がん組織におけるウイルスの活性の類似性や違いも明らかにしました。
本研究で得られたHBVの網羅的な転写マップは、今後、抗ウイルス薬の開発・評価においてウイルスの転写活性を研究するためのリソースとして活用できるため、新しいHBV治療薬の開発への貢献が期待できます。
本研究は、米国の科学雑誌『Journal of Virology』に掲載されるのに先立ち、オンライン版(9月28日付け)に掲載されました。
注1)2016年7月のWHO発表(英語)より
背景
B型肝炎ウイルス(HBV)は肝臓がんを引き起こす主要な原因であり、世界中で年間約68万6,000人がこのウイルスに起因する疾患で死亡していると推定されています。治療にはラミブジンやエンテカビルなどウイルスの増殖を抑制する複数の抗ウイルス薬が使用されていますが、ウイルスを体内から完全に排除することはできず、肝臓がんの他に肝炎や肝硬変など慢性肝疾患の原因ともなっています。また、長期にわたる抗ウイルス薬の投与によりウイルスが耐性を持ってしまうことも問題となっていることから、より有効な抗ウイルス薬が求められています。
HBVのゲノムは環状2本鎖DNAであり、全長が3,200塩基対(bp)程度と非常にコンパクトです(図1B)。しかしその遺伝子構造は複雑で、タンパク質をコードする4つの領域が重なり合っています。複数の転写開始点から6種類のmRNAが転写され7種類のタンパク質が作られることが分かっていますが、mRNAの配列も互いに重なり合っているため、通常のRNA-seq法[4]では、どのmRNAがどれだけ発現しているかを区別するのは困難でした。そのため、感染する細胞の違いや細胞状態の変化により、ウイルスゲノムの転写がどのように制御されているのか不明な点が多く残されています。
研究チームはこれまで、理研の独自技術であるCAGE法を用いて、さまざまな細胞でのRNAの発現を網羅的・定量的に解析してきました。その成果としてゲノム中に存在する内在性レトロウイルスに由来するLTR配列[5]が、肝細胞がんにおいてノンコーディングRNA(ncRNA)[6]として発現していることを明らかにしています注2)。
本研究では前回と同じサンプル・技術を用いて、外来性のウイルスであるHBVの網羅的な発現解析を試みました。
注2)2015年10月29日プレスリリース「肝細胞がんで活性化するレトロウイルス由来のRNA」
研究手法と成果
研究チームは、HBVに感染している肝細胞がんの患者から切除されたがん組織、およびがん化していない周辺組織について、発現するRNAをCAGE法によって解析しました。組織に存在するヒトのRNAとHBVのRNA配列を区別するために、まずCAGE法で得られたRNA配列をヒトゲノム配列と比較し、一致しなかったものをHBVのゲノム配列と照合しました。その結果、解析した16人のサンプルのうち15人のサンプルにおいて、がん組織および周辺組織の両方でウイルスの発現がみられました(図1A)。また、発現量はサンプルごとに大きく異なっていました。これは、治療に用いられた薬の違いなどによるウイルスの活性度の違いを反映していると考えられます。
次に、得られたCAGEデータからウイルスゲノム上に17箇所の転写開始点を同定しました(図1B)。これにより、既に知られている転写開始点を正確に再現すると同時に、過去に報告されていない複数の転写開始点を同定しました。
今回見つかった新しい転写開始点の一つは、「X遺伝子」上に存在していました(図2のPeak #11)。X遺伝子はウイルスの増殖に必須であり、感染した細胞のさまざまな遺伝子を活性化することが知られていますが、その詳細な機能は明らかになっていません。X遺伝子が作るタンパク質は、これまで知られている転写開始点(図2のPeak #10)から転写されるmRNAが持つ、1番目の開始コドン[7]ATG(アデニン・チミン・グアニン)で始まる一種類のみと考えられてきました。しかし、研究チームが発見した転写開始点は1番目の開始コドンの後ろにあるため、2番目の開始コドンから翻訳が始まる小型のタンパク質も作られている可能性が考えられました。
HBVは変異しやすいウイルスであり、さまざまな変種が存在します。今回の患者サンプルで見つかった2番目の開始コドンが他のウイルス株にも存在するかを調べるため、データベースに登録されている6,949個のウイルスの配列において、どれほど保存されているかを調べました。その結果、1番目と2番目の開始コドンはどちらも99.6%という高い保存度であることが明らかになりました(図2下)。このことは、2番目の開始コドンが1番目と同様にウイルスにとって重要であることを示しています。
次に、同様のRNA解析をHBVの研究に利用されている4つの実験モデルシステム(2種類のヒト肝臓がん由来細胞株、マウスの肝臓に強制的にHBVを感染させたもの、ヒト肝細胞の初代培養細胞にHBVを感染させたもの)について行いました。CAGE法を用いて転写開始点を特定し、ヒト患者がん組織における結果と比較したところ、いずれの実験モデルシステムでも転写開始点はほぼ一致していました。これは、実験モデルシステムがヒト肝臓がん組織におけるウイルス発現をよく再現していることを示します。また、先述したX遺伝子における2番目の転写開始点も同じ位置に検出され、実験モデルシステムにおいても小型のXタンパク質が存在することが示されました(図3)。その一方で、がん組織と実験モデルシステムとの間では、Basal Core Promoterと呼ばれるプロモーター[8]の位置が40bpほど異なっており、多少の違いが存在することも明らかになりました。
さらに、血中に存在するウイルス由来のRNAについても解析を行いました。HBVは一般的に肝臓の細胞だけで転写されると考えられていますが、ウイルス感染者の血中にはしばしばウイルス由来のRNAが検出されます。肝臓がん未発症でHBV陽性の患者の血液8検体について、CAGE法によるRNA解析を行った結果、調査した8検体すべてにおいてウイルスのRNAが検出されました。
しかし、その大部分は一つの転写開始点(図1Bの16)から始まるpregenomic RNA(ウイルスが複製するときの鋳型となるRNA)であり、17箇所の転写開始点が見つかった肝臓組織とは対称的でした。このことから、血液中で検出されたpregenomic RNAは、複製中のウイルスゲノムを取り込んだ不完全なウイルス粒子が、肝臓から血液中に放出されたものと推定されます。
今後の期待
今回得られたHBVの網羅的な転写マップは、抗ウイルス薬の開発・評価においてウイルスの転写活性を研究するためのリソースとして活用できるため、新しいHBV治療薬の開発への貢献が期待できます。
原論文情報
- Kübra Altinel, Kosuke Hashimoto, Yu Wei, Christine Neuveut, Ishita Gupta, Ana Maria Suzuki, Alexandre Dos Santos, Pierrick Moreau, Tian Xia, Soichi Kojima, Sachi Kato, Yasuhiro Takikawa, Isao Hidaka, Masahito Shimizu, Tomokazu Matsuura, Akihito Tsubota, Hitoshi Ikeda, Sumiko Nagoshi, Harukazu Suzuki, Marie-Louise Michel, Didier Samuel, Marie Annick Buendia, Jamila Faivre, & Piero Carninci, "Single-nucleotide-resolution mapping of HBV promoters in infected human livers and hepatocellular carcinoma", Journal of Virology, doi: 10.1128/JVI.01625-16
発表者
理化学研究所
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 LSA要素技術研究グループ トランスクリプトーム研究チーム
チームリーダー ピエロ・カルニンチ (Piero Carninci)
専任研究員 橋本 浩介(はしもと こうすけ)
 ピエロ・カルニンチ チームリーダー
ピエロ・カルニンチ チームリーダー
 橋本浩介 研究員
橋本浩介 研究員
お問い合わせ先
理化学研究所 ライフサイエンス技術基盤研究センター
広報・サイエンスコミュニケーション担当 山岸 敦(やまぎし あつし)
Tel: 078-304-7138 / Fax: 078-304-7112
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.CAGE法
理研が独自に開発した手法で、耐熱性逆転写酵素やmRNAのCap構造を捕捉する技術を組み合わせて転写産物の5'末端の塩基配列を決定する実験手法。この塩基配列を読み取ってゲノム配列と照らし合わせて、どこから転写が始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる。Cap Analysis Gene Expressionの略。 - 2.肝細胞がん
肝臓がんの一種。肝細胞を由来とする悪性腫瘍で、肝臓がんの70~85%を占める。慢性肝炎、肝硬変が進行すると合併して発症する。主要な危険因子は慢性のB型、C型肝炎ウイルスの感染や飲酒など。 - 3.転写マップ
DNAを鋳型としてRNAが合成されるときの始まりの位置(転写開始点)、と合成されたRNAの量をCAGE法によって特定し、ゲノム上に地図として示したもの。 - 4.RNA-seq法
組織や細胞で発現している全RNA(トランスクリプトーム)を解析する手法の1つ。mRNAやncRNAの断片的な配列情報(約50-125塩基)を網羅的に取得し、ゲノム配列と対応させることで、遺伝子発現量の定量や新たな転写配列の発見を行う。RNA sequencingの略。 - 5.LTR配列
レトロウイルスに特徴的な、ウイルスゲノムの両端に位置する繰り返し配列。宿主ゲノムに挿入されると強い転写活性を発揮し、ウイルスゲノムの発現を促進する機能を持つ。ヒトゲノムに存在するレトロトランスポゾンにおいては、LTRの転写活性は抑制されている場合が多いと考えられている。LTRはLong Terminal Repeatの略。 - 6.ノンコーディングRNA(ncRNA)
non-coding RNA。メッセンジャーRNA(mRNA)と異なり、タンパク質の設計図として用いられないRNAの総称。エピジェネティクス(塩基配列に依存しない遺伝子の調節機構)や転写、翻訳といった生物の活動の中枢をなす反応、幹細胞性の維持など、さまざまな働きに関与するncRNAが次々に報告されており、その重要性に注目が集まってきている。 - 7.開始コドン
メッセンジャーRNA(mRNA)がタンパク質に翻訳される際に、翻訳の開始点となる3塩基のこと。 - 8.プロモーター
ゲノムDNA上でRNAに書き写される領域の近くにあり、遺伝子を発現させる機能を持つ領域(配列)。
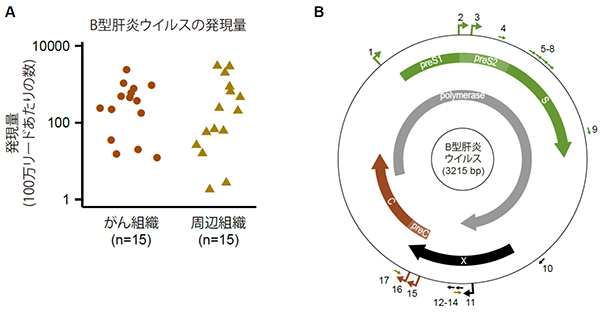
図1 B型肝炎ウイルス(HBV)の発現量と転写開始点
- A.15人の肝細胞がん患者で確認されたHBVゲノムの転写。丸と三角はそれぞれ、患者ごとのがん組織と周辺組織での発現量を示し、発現量の個人差は数千倍に及ぶことが分かる。
- B.CAGE法で明らかになったHBVゲノムの17箇所の転写開始点。円内の太い矢印はタンパク質をコードする連続した読み枠(オープンリーディングフレーム)を表し、英語名はそれらのタンパク質産物の名称を指す。円外の数字と矢印は、転写開始点を示す。矢印(大)は発現が高いもの(合計1000リード以上)を、矢印(小)は発現が低いもの(100-1000リード)を表す。
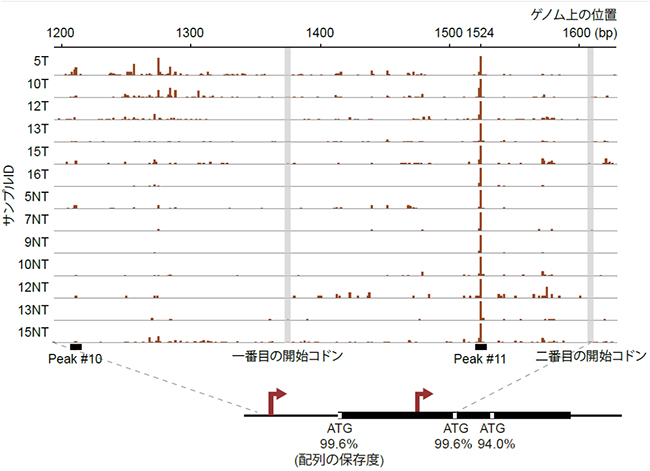
図2 X遺伝子上の新しい転写開始点
上:X遺伝子の発現が一定以上(100万リード当たり1リード以上)あった13サンプル(がん組織はTで表された6サンプル、周辺組織はNTで表された7サンプル)について、X遺伝子の転写開始点をゲノム上にマッピングした。縦のバーは、その位置から始まる転写産物のピークを示す。
下:X遺伝子のmRNAの構造。転写開始点を矢印で、開始コドンをATG(アデニン・チミン・グアニン)で示す。三つの開始コドンのうち、1番目と2番目の保存度が99.6%と高いのに対し、3番目の保存度は94.0%とわずかに低い。
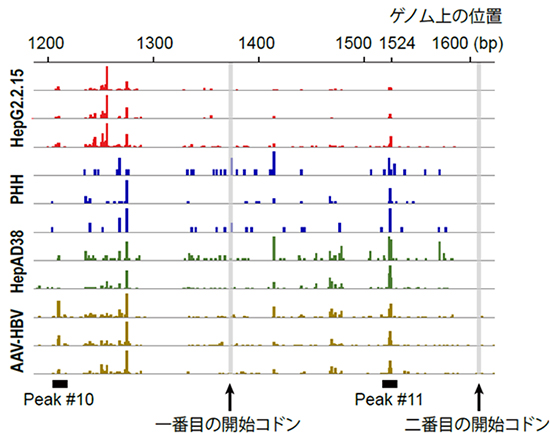
図3 実験モデルシステムにおけるX遺伝子の転写開始点
HBVによる実験モデルシステムである、ヒト肝臓がん由来細胞株(HepG2.2.15、HepAD38)、ヒト肝細胞の初代培養細胞(PHH)、マウスの肝臓に強制的にHBVを感染させたもの(AAV-HBV)に対して、CAGE法による解析を行い、X遺伝子の転写開始点を調べた。肝臓がん患者のサンプルから同定された新規転写開始点であるpeak#11は、これらの実験モデルシステムでも検出された。
