要旨
理化学研究所(理研)ライフサイエンス技術基盤研究センター構造・合成生物学部門超分子構造解析研究チームの関根俊一チームリーダー、江原晴彦研究員らの共同研究チーム※は、細胞内で転写を行っている状態のRNAポリメラーゼ[1]IIの立体構造を明らかにしました。
RNAポリメラーゼII(Pol II)は、真核生物の核内でメッセンジャーRNA(mRNA)の転写を担う酵素で、多数のサブユニットからなる巨大なタンパク質複合体です。転写の過程においてPol IIは、さまざまなタンパク質と結合することでさらに巨大な複合体となって機能しています。しかし、それらがどのような姿をしているかはほとんど分かっていません。近年、クライオ電子顕微鏡[2]技術の進歩により、従来は解析が難しかった生体分子複合体の立体構造を調べることができるようになりつつあります。本研究では、最先端のクライオ電子顕微鏡解析およびX線結晶構造解析[3]を組み合わせた統合的手法により、細胞内で転写を行っているPol II複合体(転写伸長複合体)の姿を明らかにしました。
転写伸長複合体は、Pol IIと4種類の転写伸長因子で構成されます。転写伸長因子はPol IIの表面の広い範囲に分布し、Pol IIの機能に重要な箇所を重点的に補強・再構築し、DNAの結合とRNA合成に特化した複合体を完成させていることが明らかになりました。転写伸長因子の結合により、約30塩基対の長さのDNAをカバーするトンネルが形成されます。また、因子の一部はRNAの排出口周辺に結合し、RNAの効率的な排出を助けています。これらの特徴は、転写中の一時停止やDNAの脱落を防ぎ、効率的な転写伸長を行う転写伸長複合体の性質をよく表しています。また、本研究で明らかにした転写伸長複合体の構造は、転写のON/OFF制御やmRNAプロセシング[4]などPol II特有の機能を支える構造基盤を明らかにした点でも重要です。
本成果をもとに今後、転写制御やその破綻による疾患メカニズムについての研究が発展するものと期待できます。
本研究は、米国の科学雑誌『Science』(9月1日号)に掲載されるのに先立ち、オンライン版(8月3日付け:日本時間8月4日)に掲載されます。
本研究は、日本学術振興会 科学研究費補助金(基盤研究(B)「転写因子によるRNAポリメラーゼの構造変化と転写制御のメカニズム」)代表 関根俊一および日本医療研究開発機構が所管する創薬等ライフサイエンス研究支援基盤事業の支援を受けて行われました。
※共同研究チーム
理化学研究所
ライフサイエンス技術基盤研究センター 構造・合成生物学部門
超分子構造解析研究チーム
チームリーダー 関根 俊一(せきね しゅんいち)
研究員 江原 晴彦(えはら はるひこ)
タンパク質機能・構造研究チーム
チームリーダー 白水 美香子(しろうず みかこ)
上級研究員 重松 秀樹(しげまつ ひでき)
研究員 横山 武司(よこやま たけし)
横山構造生物学研究室
上席研究員 横山 茂之(よこやま しげゆき)
背景
生命の設計図である遺伝情報は、細胞の核内にあるDNAに塩基の配列として保存されています。生物は遺伝子、つまりDNA上で遺伝情報が記された領域を、適切なときに適切な器官や組織、細胞で働かせることにより、その体を形成、維持し活動しています。
遺伝子を働かせるためには、遺伝子のDNA配列がまず、RNAにコピー(転写)される必要があります。そのRNAがタンパク質の設計図となるメッセンジャーRNA(mRNA)である場合、mRNAの配列をもとにタンパク質が合成(翻訳)されます。この第一段階でDNAからRNAへの転写をつかさどるRNAポリメラーゼは、全ての生物にとって必須の酵素です。ヒトを含め真核生物には3種類のRNAポリメラーゼが存在し、そのうちRNAポリメラーゼII(Pol II)がmRNAの転写を担当しています。Pol IIは12個のタンパク質(サブユニット)からなる分子量約50万の巨大な複合体です。
2002年、米国のロジャー・コーンバーグ博士(2006年ノーベル化学賞受賞)らは、X線結晶構造解析によりPol IIの立体構造を解明し、Pol IIが“カニのはさみ”のような形状をしていることを示しました。一方、細胞内のPol IIは、転写因子など多数のタンパク質と結合し、さらに巨大な複合体を形成して働いていることが知られています。しかし、実際に細胞内で働いているPol IIの姿は、これまでよく分かっていませんでした。
近年、試料の結晶化を必要としないクライオ電子顕微鏡による観察技術が著しく進歩し、これまで結晶化が難しかったタンパク質や巨大な生体分子複合体の立体構造の解析が可能になってきました。この手法を用いて、Pol IIについても、転写の開始時に形成される巨大な複合体(転写開始複合体)を中心に構造研究が行われています。一方で、RNAの伸長中に形成される巨大な複合体(転写伸長複合体)の構造はこれまで明らかにされていませんでした。
研究手法と成果
共同研究チームは、単細胞真核生物である酵母の一種(メタノール資化酵母)から取り出したPol IIと、人工合成したDNA、RNA、および4種類の転写伸長反応を制御するタンパク質(転写伸長因子Elf1[5]、Spt4[6]、Spt5[6]、TFIIS[7])を組み合わせ、転写伸長複合体を試験管内で再構成しました。創薬等ライフサイエンス研究支援基盤事業[8]の拠点である当センターのクライオ電子顕微鏡装置を用いたクライオ単粒子解析、および、大型放射光施設「SPring-8」[9]の放射光を用いたX線結晶構造解析を行い、両者を組み合わせた統合的手法により細胞内で転写を行っている巨大なPol II複合体(転写伸長複合体)の構造を明らかにしました(図1)。
転写伸長因子は、Pol II表面の広い範囲に分布しており、機能的に重要な部位に重点的に結合することで、それらの機能部位を補強・再構築し、転写伸長に特化した複合体を完成させていることが明らかになりました。Pol IIは単独でもDNAと結合できますが、“カニのはさみ”のようなDNAが結合する溝は開いたままで閉じることはできません。四つの転写伸長因子のうち、Elf1とSpt4、Spt5は、DNAが結合した溝を外側からカバーして、完全に閉じたトンネルを形成することが分かりました(図1上左)。Elf1はPol IIの進む方向(下流側)でDNAを取り込む「DNA導入トンネル」を作り出します(図2左)。Spt4とSpt5は上流側に「DNA送出トンネル」を新たに確立します(図2中)。結果として、DNAは約30塩基対の長さにわたってタンパク質でできたトンネルを通リ抜けるかたちになり、規定された空間内で精密なRNAの転写が可能になっていることが分かりました(図1下)。
一方、RNAの出口もPol II単独では十分に完成していません。Spt5はRNA排出口に結合して「RNA送出トンネル」を完成させ、効率的なRNA送出を可能にしていることが分かりました(図2右)。このように、転写伸長因子がPol IIに欠けている構造を補強・拡張し、DNAとRNAの通路を完全に独立したものとして確立することにより、機能的な転写伸長複合体が完成することが明らかになりました。これにより、転写中の一時停止やDNAの脱落を防ぎ、効率的な転写が可能になると考えられます。
本研究で明らかにしたPol II転写伸長複合体の構造は、転写開始時に形成される開始複合体の構造とは大きく異なっています(図3)。転写開始時には、基本転写因子[10]と呼ばれる一群のタンパク質がPol IIに結合して開始複合体を形成し、Pol IIが最初にDNAに結合するのを助けます。転写開始から伸長のステージへの移行の際には、基本転写因子と転写伸長因子の全面的な交換が行われ、 Pol IIはRNA伸長に特化した異なる形の複合体へと変貌を遂げることが分かりました。このPol II転写伸長複合体は、転写のON/OFF制御のターゲットであるとともに、転写と連携したmRNAプロセシングなどの足場になっており、本研究はそれらの構造基盤を初めて解明したという点でも画期的な成果です。
今後の期待
本研究では、最先端のクライオ電子顕微鏡解析とX線結晶構造解析を組み合わせて、細胞内で働いている状態を再現した巨大複合体の構造解析に成功しました。Pol II転写伸長複合体は、転写のON/OFF制御のターゲットであると同時に、mRNAプロセシングなど、多くの重要なイベントが行われる足場となっています。今回明らかとなった構造はそれらの構造基盤を提供するもので、この構造を出発点として多くの生命現象の理解が進むものと期待できます。また、がんやウイルス感染症などの疾患は、多くの場合、転写制御の破綻や撹乱に基づいて起こるため、今後の研究の進展により、発症のメカニズムや治療法の解明につながると期待できます。
Pol II転写伸長複合体に代表される巨大複合体は構造解析の難度が非常に高く、これまで取り残されていた創薬対象のひとつです。本研究で確立した構造解析技術をさまざまな巨大複合体やタンパク質間相互作用の解析に応用し、細胞内に近い状態の構造データを取得することで、創薬対象の拡大が期待できます。
原論文情報
- Haruhiko Ehara, Takeshi Yokoyama, Hideki Shigematsu, Shigeyuki Yokoyama, Mikako Shirouzu and Shun-ichi Sekine, "Structure of the complete elongation complex of RNA polymerase II with basal factors", Science, doi: 10.1126/science.aan8552
発表者
理化学研究所
ライフサイエンス技術基盤研究センター 構造・合成生物学部門 構造生物学グループ 超分子構造解析研究チーム
チームリーダー 関根 俊一(せきね しゅんいち)
研究員 江原 晴彦(えはら はるひこ)
 関根俊一チームリーダーと江原晴彦研究員
関根俊一チームリーダーと江原晴彦研究員
お問い合わせ先
理化学研究所 ライフサイエンス技術基盤研究センター
広報・サイエンスコミュニケーション担当 山岸 敦(やまぎし あつし)
Tel: 078-304-7138 / Fax: 078-304-7112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
お問い合わせフォーム
AMED事業に関するお問い合わせ先
日本医療研究開発機構(AMED)創薬戦略部 医薬品研究課
Tel: 03-6870-2219
20-DDLSG-16 [at] amed.go.jp(※[at]は@に置き換えてください。)
補足説明
- 1.RNAポリメラーゼ
DNAの塩基配列を読み取って、相補的なRNAを合成する酵素で、遺伝子発現(セントラルドグマ)の第一段階をつかさどる生命活動に必須の酵素。複数のタンパク質(サブユニット)が集合してできた巨大な複合体で、細菌からヒトまで共通した“カニのはさみ”のような形をしている。中央にできた溝にDNAを挟み込み、およそ10塩基対のDNAをほどいて「転写バブル」を形成し、一方の鎖を鋳型にしてRNAを合成する。 - 2.クライオ電子顕微鏡
タンパク質などの生体試料を観察するために開発された電子顕微鏡。タンパク質などの試料を含んだ溶液を薄く展開し、液体エタン(-183℃~-160℃)中で急速凍結して試料をごく薄い氷の層に閉じ込めた上、液体窒素温度(-196℃)で電子顕微鏡により観察する。試料を染色固定する方法に比べてニつの利点がある。一つ目は、低温で電子線を照射するためタンパク質試料の電子線による損傷が軽減される。二つ目は、タンパク質試料を生理的な(自然な)状態に近い条件で観察できる。 - 3.X線結晶構造解析
物質の結晶を作り、それにX線を照射して得られる回折データを解析することにより、物質の内部構造を調べる方法。タンパク質の構造を原子レベルの分解能で詳細に解明するための最も有力な方法の一つである。 - 4.mRNAプロセシング
真核生物のmRNAはまず前駆体としてPol IIにより転写され、5'末端キャップ構造の付加、スプライシングによるイントロンの除去、3'末端のポリA配列の付加などを受けて成熟する。これらのプロセシングは転写後に起きるのではなく、転写の開始、伸長、終結と密接に関わることが分かっている。 - 5.Elf1
真核生物と古細菌の一部に保存された転写伸長因子。Spt4やSpt5、TFIISなどの転写伸長因子と合成致死性を示す因子として同定された。細胞内で転写中のPol IIに共存していることが知られているほか、クロマチン構造の維持への関与も見いだされている。 - 6.Spt4、Spt5
Spt4とSpt5は、複合体(Spt4/Spt5)として機能する転写伸長因子。Spt4は真核生物と古細菌に、Spt5は真核生物と古細菌だけでなくバクテリアにも保存されている。転写中に起こるPol IIの休止を抑制し、プロセッシブな転写的伸長を保証する。高等真核生物では、DSIFという名で知られており、転写のON/OFF制御において中心的な役割を果たしている。また、CTRと呼ばれるC末端のフレキシブルな領域を持っており、これを介して複数の他のタンパク質をPol IIに呼び込む役割も果たす。 - 7.TFIIS
Pol IIのRNA切断活性を増強させる転写伸長因子。ミスマッチ塩基の取り込みなどにより転写が一時休止すると、Pol IIはDNA上を後退しながらRNAを分解した後、転写を再開することができる。バクテリアにも同様の機能を持つGreA、GreBが存在する。 - 8.創薬等ライフサイエンス研究支援基盤事業
日本医療研究開発機構(AMED)が所管する、大型実験施設の外部開放および最先端技術を用いた研究支援事業。ライフサイエンス研究の成果を医薬品等の実用化につなげることを目的とする。ライフサイエンス技術基盤研究センターは本事業において、低温電子顕微鏡での構造解析が可能な試料調製と、ゲノミクス解析、インシリコスクリーニングを支援している。 - 9.大型放射光施設「SPring-8」
理研が所有する、兵庫県の播磨科学公園都市にある第三世代放射光施設。SPring-8の名前はSuper Photon ring-8GeVに由来する。放射光(シンクロトロン放射)とは、電子を光とほぼ等しい速度まで加速し、電磁石によって進行方向を曲げたときに発生する細くて強力な電磁波のこと。SPring-8では、遠赤外から可視光線、軟X線を経て硬X線に至る幅広い波長域で放射光を得ることができるため、原子核の研究からナノテクノロジー、バイオテクノロジー、産業利用や科学捜査まで幅広い研究が行われている。 - 10.基本転写因子
RNAポリメラーゼIIがRNAの転写を開始する際に必要とされる一群の転写因子タンパク質。TFIIA, TFIIB, TFIID (TBP), TFIIE, TFIIF, TFIIHが存在する。( 図3左(5FZ5)はTFIIA, TFIIB, TBP, TFIIE, TFIIFが結合した状態の複合体)
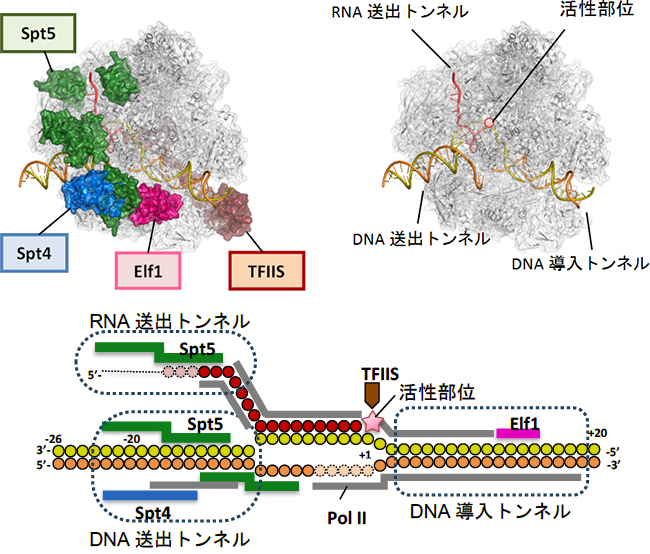
図1 RNAポリメラーゼll(Pol II)転写伸長複合体の構造
上)本研究で明らかにした四つの転写伸長因子(Elf1、Spt4、Spt5、TFllS)を結合したPol II転写伸長複合体の構造(左)。Pol II構造を灰色とし、他の分子を色分けしている。転写伸長因子を結合しない状態では、DNAが広い範囲でむき出しになっており、RNAを支持する構造も弱い(右)。なおこの図の配置では、Pol IIは右方向に進みながら転写伸長を行う。
下) Pol II転写伸長複合体における転写伸長因子、DNA、RNAの相互作用をまとめた図。DNAの鋳型鎖において、リボヌクレオチドが付加される位置を+1としている。Pol II(この図では省略)は右方向に進みながら転写伸長を行う。赤丸はRNAのリボヌクレオチド、黄丸は鋳型鎖DNAのデオキシリボヌクレオチド、橙丸は非鋳型鎖DNAのデオキシリボヌクレオチドを示す。中央部分でDNAが開いた構造は、転写バブルを表す。
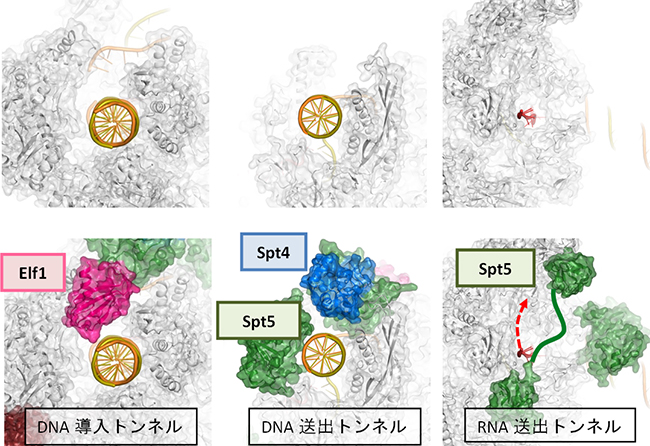
図2 転写伸長因子によって形成される新たな構造
転写伸長因子は、Pol IIと一体化してRNA伸長に特化した複合体を形成する。左はDNA導入トンネル、中央はDNA送出トンネル、右はRNA送出トンネルを示す。上段は、転写伸長因子を取り除いた構造を比較のために示したもの。
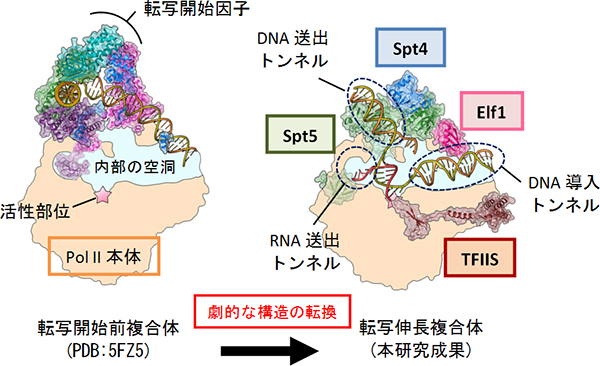
図3 転写開始前複合体から大きく変貌する転写伸長複合体
転写開始の段階から伸長の段階にかけて、Pol II複合体の構造は劇的な変化を遂げる。Pol II転写伸長複合体は、転写のON/OFF制御のターゲットであるとともに、転写と連携したmRNAプロセシングなどの足場になっている。
