理化学研究所(理研)生命機能科学研究センター合成生物学研究チームの真野智之研修生(研究当時)、山田陸裕上級研究員、上田泰己チームリーダー、東京大学大学院医学系研究科機能生物学専攻システムズ薬理学教室の史蕭逸助教らの共同研究グループは、全脳全細胞解析[1]を可能にするプラットフォームであるクラウド[2]システム「CUBIC-Cloud[3]」を開発しました。
本研究成果は、脳全体の遺伝子の働きやネットワーク構造などの膨大な3次元データをクラウド上で保管・解析し、データ駆動型[4]の神経科学を推進するための基盤技術として、神経科学の発展に大きく貢献するものと期待できます。
CUBIC-Cloudは、組織透明化技術[5]であるCUBIC[6]で得られた全脳全細胞データを取り込み、複数の脳画像の位置合わせ(レジストレーション)[7]、定量解析、可視化といった機能をグラフィカルユーザーインターフェース[8]とともに提供します。全ての計算はクラウドで実行されるため、強力な計算機環境を持たない研究者でも使用可能です。また、CUBIC-Cloudで実行した解析結果はクラウドを通じて世界の研究者に共有・公開することができ、将来的には多数の全脳データを用いたデータマイニング[4]の可能性を提供しています。
本研究は、科学雑誌『Cell Reports Methods』(6月21日付:日本時間6月22日)に掲載されます。またCUBIC-Cloudの利用にあたっては、CUBIC-Cloudのホームページでユーザー登録を受け付けています。
背景
近年、生体組織を3次元的に観察することを可能にするさまざまな組織透明化技術が発表されています。上田泰己チームリーダーらは、2014年に第一世代CUBIC技術を発表しました注1)。その後、組織透明化・3次元観察技術の発展により、神経科学の研究においてマウス全脳の高解像な画像を取得することが普及してきました。しかし、このような研究の中で、大量の画像データを効率的に解析し、さらには他の研究者と共有するためのソフトウェアがしばしばボトルネックとなっていました。
- 注1)2014年11月7日プレスリリース「マウスを丸ごと透明化し1細胞解像度で観察する新技術」
研究手法と成果
共同研究グループは、パブリッククラウドを利用したマウス全脳解析プラットフォームである「CUBIC-Cloud」を開発しました。まずユーザー側では、CUBIC技術で透明化処理したマウス脳を光シート顕微鏡[9]で撮影し、画像解析ソフトで細胞検出までを行います。このデータをCUBIC-Cloudにアップロードすると、ブラウザ上で、複数の脳の位置合わせ(レジストレーション)、定量解析、可視化といった機能を簡単に使うことができます(図1)。
全ての計算はクラウドで実行されるため、強力な計算機環境を持っていない研究者も使用することができます。また、CUBIC-Cloudで実行した解析結果はクラウドを通じて世界の研究者に共有・公開することができるため、将来的には多数の全脳データを用いたデータ駆動型の研究が可能になります。
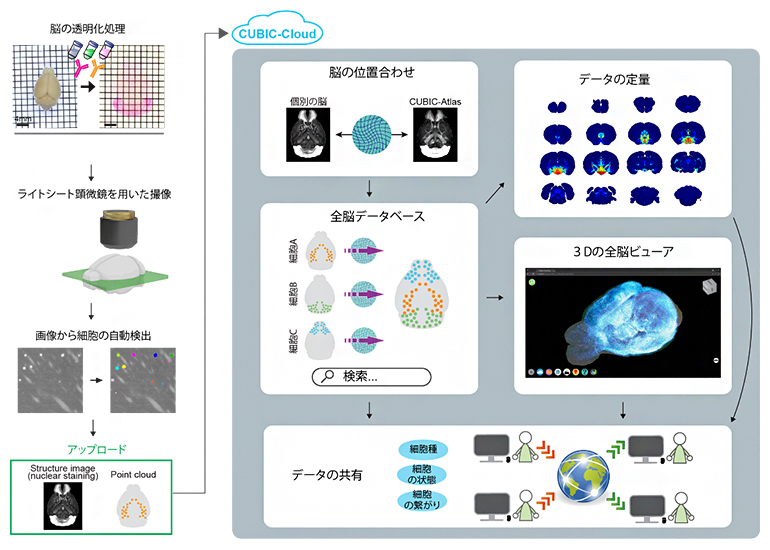
図1 CUBIC-Cloudの概要図
CUBIC-Cloudによる全脳解析パイプラインの概要。マウス脳を透明化処理後、光シート顕微鏡で撮像し、取得した画像から細胞の点を一つ一つ検出した。ユーザーは点検出した画像をCUBIC-Cloudにアップロードする。クラウド上で、脳と脳の位置合わせを行い、さまざまな解析をすることが可能である。インタラクティブな3D全脳ビューアも提供している。
さらに、CUBIC-Cloudの性能を実証するために、60個体以上のマウスの全脳を解析しました(図2)。その結果、さまざまな種類の神経細胞(ニューロン)が脳のどこにあるのか、認知症モデルマウスにおけるアミロイドβプラーク[10]沈着の定量解析、実験的に脳内炎症を起こした際の脳神経細胞の活動の変化などを捉えることに成功しました。
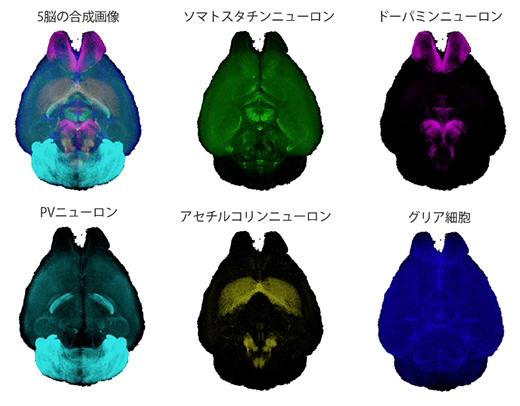
図2 CUBIC-Cloudを用いた解析の実施例
マウス全脳の4種の神経細胞(ソマトスタチンニューロン、ドーパミンニューロン、パルブアルブミン陽性(PV)ニューロン、アセチルコリンニューロン)およびグリア細胞を抗体染色で可視化し、CUBIC-Cloud上で一つの脳の上に合成したもの(左上)。これによって、さまざまな脳画像の比較が可能となる。左上は5種類の細胞の合成画像である。
今後の期待
CUBIC-Cloudは、全脳解析をより簡便に実行するためのソフトウェア基盤を神経科学コミュニティーに提供します。今後、多くの研究者が本プラットフォームを利用することで全脳データの収集・解析がさらに加速し、より統一的な脳の理解に貢献するものと期待できます。
補足説明
- 1.全脳全細胞解析
複数の領域で構成される脳全体について、1細胞レベルの解析を行うこと。 - 2.クラウド
インターネットを通じてユーザーにサービスを提供する形態。 - 3.CUBIC-Cloud
理研で開発された透明化・全細胞関連技術の社会実装を担う株式会社CUBICStarsが、運営する有料のクラウド型全脳全細胞解析サービス。本研究成果の神経科学分野における重要性と公共性を鑑み、2021年中にアカウント登録を行う無料ユーザーに2脳分の全脳全細胞解析サービスを無料で提供予定。 - 4.データ駆動型、データマイニング
仮説検証により実験を進める仮説駆動型手法に対し、研究対象に関する精密で大量の系統的データの解析から新しい知識を得る研究手法をデータ駆動型、あるいはデータマイニングと呼ぶ。 - 5.組織透明化技術
生体組織を透明にし、内部観察を可能とする手法。古くは100年ほど前から使用されてきたが、2000年前後からさらなる開発が進んだ。組織透明化には組織を通過する光の散乱および吸収を解消することが重要であり、組織内の光散乱物質(主に脂質)や光吸収物質(主に色素)を除去したのち、溶媒と組織内の屈折率を均一化(屈折率調整)する操作が行われる。 - 6.CUBIC
理研の研究チームが2014年に開発した、3次元イメージング、画像解析を組み合わせた全臓器・全身全細胞解析のためのパイプライン。CUBICは、Clear, Unobstructed Brain/Body Imaging Cocktails and Computational analysisの略。 - 7.位置合わせ(レジストレーション)
本研究では、核染色画像を用いて脳を構成する全ての細胞核の座標を抽出し、全脳の3次元構造をデータ化したマウス脳アトラス(CUBIC-Atlas)に対して、個々の脳サンプルから得られた蛍光抗体染色画像を1細胞レベルでマッピングすること。 - 8.グラフィカルユーザーインターフェース
コンピュータのユーザーインターフェースの一つで、ユーザーにとっての使いやすさを重視し、情報の提示において、マウスなどによる画面上の簡単な操作によって指示を送ることができるようにした手法。 - 9.光シート顕微鏡
レーザー光をシート上に広げて透明化サンプルの側面から照射し、サンプルの上からカメラで撮影することで、透明化サンプル内の特定の平面(光学切片)を撮影することができる顕微鏡。サンプルをZ方向に動かして光学切片を連続撮影し、高速に3次元画像を取得できる。 - 10.アミロイドβプラーク
Aβ(アミロイドベータ)は、40~43個のアミノ酸が連なってできたペプチド(タンパク質断片)である。アミロイド前駆体タンパク質(APP)が、βセクレターゼやγセクレターゼと呼ばれる酵素によって切断されることで生じる。アルツハイマー型認知症では、脳内のAβが凝集して線維状(Aβプラーク)になり、脳に沈着することが良く知られている。
共同研究グループ
理化学研究所 生命機能科学研究センター
合成生物学研究チーム
チームリーダー 上田 泰己(うえだ ひろき)
(東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室教授)
上級研究員 山田 陸裕(やまだ りくひろ)
客員研究員 洲﨑 悦生(すさき えつお)
(研究当時 東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室 准教授)
(現 順天堂大学医学研究科 教授)
テクニカルスタッフⅠ 清水 知佳(しみず ちか)
研修生(研究当時) 真野 智之(まの ともゆき)
(研究当時 東京大学 大学院医学系研究科 システムズ薬理学教室 大学院生)
(現 沖縄科学技術大学院大学 特別研究員)
比較コネクトミクス研究チーム
チームリーダー 宮道 和成(みやみち かずなり)
東京大学
大学院医学系研究科 機能生物学専攻 機能生物学専攻 システムズ薬理学教室
助教 史 蕭逸(シ・ショウイ)
(理化学研究所 生命機能科学研究センター 合成生物学研究チーム 客員研究員)
大学院生 昆 一弘(こん かずひろ)
大学院農学生命科学研究科 応用生命化学専攻 生物化学研究室
教授 東原 和成(とうはら かずしげ)
研究支援
本研究は理化学研究所運営費交付金(生命機能科学研究)で実施し、科学技術新機構機構(JST)戦略的創造研究推進事業さきがけ「組織3D染色による細胞の網羅的解析技術の開発 (研究代表者:洲崎悦生) 」、JST-ERATO東原化学感覚シグナルプロジェクト(研究総括:東原和成)、日本学術振興会(JSPS)科学研究費補助金基盤研究(B)「臓器全体のタンパク質発現・修飾・活性状態の3D検出法開発 (研究代表者:洲崎悦生) 」、同新学術領域「網羅的3次元観察技術による細胞のダイバーシティー検証 (研究代表者:洲崎悦生)」、同基盤研究(S)「哺乳類生体リズム振動体の設計 (研究代表者:上田泰己) 」、同特別研究員奨励費 「個体の表現型を支える細胞システム理解のためのレジストレーション手法の開発(特別研究員:真野智之)」、公益財団法人武田科学振興財団 武田報彰医学研究助成「全脳イメージング技術を用いた次世代中枢薬理学の創成(研究代表者:上田泰己)」、Human Frontier Science Program grant 2018 「Sleep, the clock, and the brain: a neuromathematical approach(研究代表者:上田泰己)」、日本医療研究開発機構(AMED)先端的バイオ創薬等基盤技術開発事業「先端的医療技術に対する全臓器・全身スケールでの評価技術基盤の開発(研究開発代表者:上田泰己)」、同革新的技術による脳機能ネットワークの全容解明プロジェクト「マーモセット脳の3次元観察・解析に資する基盤技術開発(研究開発代表者:上田泰己)」、ANRI基礎科学スカラーシップ(研究代表者:真野智之)の支援を受けて行われました。
原論文情報
- Tomoyuki Mano, Ken Murata, Kazuhiro Kon, Chika Shimizu, Hiroaki Ono, Shoi Shi, Rikuhiro G. Yamada, Kazunari Miyamichi, Etsuo A. Susaki, Kazushige Touhara, Hiroki R. Ueda*, "CUBIC-Cloud provides an integrative computational framework toward community-driven whole-mouse-brain mapping", Cell Reports Methods, 10.1016/j.crmeth.2021.100038
発表者
理化学研究所
生命機能科学研究センター 合成生物学研究チーム
チームリーダー 上田 泰己(うえだ ひろき)
研修生(研究当時) 真野 智之(まの ともゆき)
上級研究員 山田 陸裕(やまだ りくひろ)
東京大学 大学院医学系研究科 機能生物学専攻 システムズ薬理学教室
助教 史 蕭逸(シ・ショウイ)
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
東京大学 医学部 総務チーム(総務担当)
Tel: 03-5841-3304
Email: ishomu [at] m.u-tokyo.ac.jp
産業利用に関するお問い合わせ
AMED事業に関するお問い合わせ先
日本医療研究開発機構(AMED)
創薬事業部 医薬品研究開発課 先端的バイオ創薬等基盤技術開発事業
Email: sentan-bio [at] amed.go.jp
疾患基礎研究事業部 疾患基礎研究課
革新的技術による脳機能ネットワークの全容解明プロジェクト
Tel: 03-6870-2286 / Fax: 03-6870-2243
Email: brain-m [at] amed.go.jp
※上記の[at]は@に置き換えてください。
