理化学研究所生命機能科学研究センターバイオインフォマティクス研究開発チームの林哲太郎技師、二階堂愛チームリーダーらの研究チーム※が開発した「1細胞完全長トータルRNA[1]シーケンス法『RamDA-seqTM』[2]」がこのたび商用化につながりました。
日本国内では、ゲノム医療[3]の保険適用が開始され、患者ごとに数十の遺伝子を解析して適切な治療法や薬剤を選択する方向に進んでいます。さらに近年、がんなどの疾患では個々の細胞で性質が異なり、その治療で1細胞ごとの全遺伝子解析の有効性が示されてきました。しかし、検体が微量なため、検出できる遺伝子数が少なく、遺伝子配列の一部しか計測できないため、疾患の原因となる変異を漏れなく計測できない問題がありました。
研究チームでは、この課題を克服した『RamDA-seqTM』を開発しました。この方法は、RNA全長を検出でき、非ポリA型RNA[4]も検出できるため、既存法と比べ多くの遺伝子や変異を計測できます。遺伝子配列に変異が起きるようながんなどを対象に、全遺伝子レベルのゲノム診断への貢献が期待できます。
研究チームは、本技術の製品化のため、東洋紡株式会社へ技術指導を行いました。東洋紡株式会社は、理研から本技術の特許実施のライセンスを受け、「GenNext® RamDA-seqTM Single Cell Kit」および「RT-RamDATM cDNA synthesis Kit[5]」の発売につながりました。
関連リリース
背景
2019年6月1日より、日本国内においてもがんゲノム医療の保険適用が開始され、個々の患者ごとに数十の遺伝子を解析し、適切な治療法や薬剤を選択する方向に進んでいます。さらに近年の研究成果から、がんにおいて個々の細胞レベルでも性質に差異があり、その治療においてはシングルセル(1細胞)レベルでの全遺伝子解析(1細胞RNA-seq[6])の有効性が示されてきました。しかし、その解析は対象が微量なため、実用化には技術的課題が多く残されています。
まず、既存の手法では検出できる遺伝子数が少ないという問題がありました。また非ポリA型のRNAを捉えられません。このため、疾患の原因や診断のマーカーに利用できる遺伝子数は、さらに少なくなります。次に、既存の手法ではRNAの一部の配列しか計測できないため、疾患の原因となるRNAの変異を漏れなく計測することができず、研究や診断の精度が十分に得られませんでした(図1)。
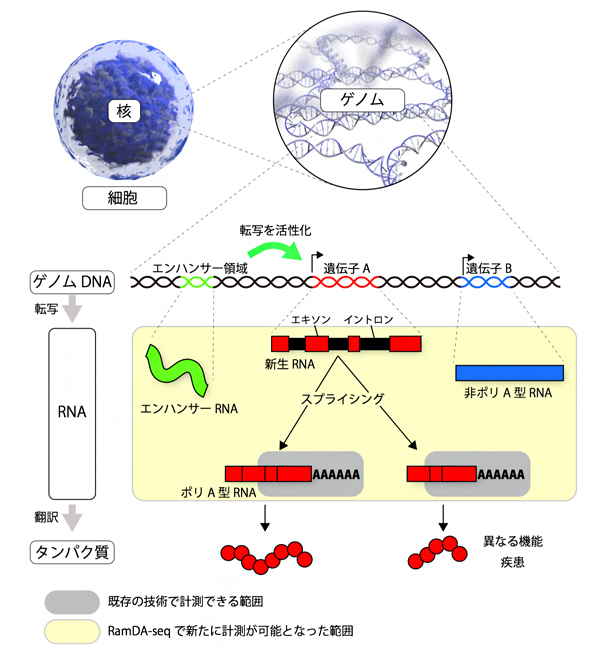 図1 ゲノムから遺伝子、RNA、タンパク質まで
我々の身体を形成する全ての細胞は、共通の遺伝情報を持つ。この遺伝情報をゲノムといい、細胞核内にあるDNAにその情報は保存されている(ゲノムDNA)。ゲノムDNA上には遺伝情報が記録されている遺伝子領域とそうでない領域があり、遺伝子領域からはRNAが転写される。転写された直後のRNA(新生RNA)にも遺伝情報を持つ領域(エキソン)と持たない領域(イントロン)が存在するため、スプライシングという機構でイントロンが取り除かれ成熟したRNAとなる。成熟RNAは、末端にアデニン(A)が連続的に付加されてポリA型RNAになるものと、Aが付かない非ポリA型RNAになるものに分類される。ポリA型RNAとなる遺伝子の多くは、タンパク質に翻訳されて機能するが、非ポリA型RNAとなる遺伝子は、一部の例外を除いてタンパク質に翻訳されずRNAのままで機能する。スプライシングが変化すると、RNAの長さと配列が変わり、翻訳されるタンパク質の機能が変わったり、疾患の原因となる。また、遺伝子領域外には、遺伝子の転写活性を調整するエンハンサー領域があり、最近になって、エンハンサー領域から転写されるRNA(エンハンサーRNA)が発見された。
図1 ゲノムから遺伝子、RNA、タンパク質まで
我々の身体を形成する全ての細胞は、共通の遺伝情報を持つ。この遺伝情報をゲノムといい、細胞核内にあるDNAにその情報は保存されている(ゲノムDNA)。ゲノムDNA上には遺伝情報が記録されている遺伝子領域とそうでない領域があり、遺伝子領域からはRNAが転写される。転写された直後のRNA(新生RNA)にも遺伝情報を持つ領域(エキソン)と持たない領域(イントロン)が存在するため、スプライシングという機構でイントロンが取り除かれ成熟したRNAとなる。成熟RNAは、末端にアデニン(A)が連続的に付加されてポリA型RNAになるものと、Aが付かない非ポリA型RNAになるものに分類される。ポリA型RNAとなる遺伝子の多くは、タンパク質に翻訳されて機能するが、非ポリA型RNAとなる遺伝子は、一部の例外を除いてタンパク質に翻訳されずRNAのままで機能する。スプライシングが変化すると、RNAの長さと配列が変わり、翻訳されるタンパク質の機能が変わったり、疾患の原因となる。また、遺伝子領域外には、遺伝子の転写活性を調整するエンハンサー領域があり、最近になって、エンハンサー領域から転写されるRNA(エンハンサーRNA)が発見された。
研究チームは、これらの課題を解決するため、1細胞レベルで従来は検出の難しかった多様なRNAの全長を計測する『RamDA-seqTM』を開発し、論文を出版しました注1、注2)。本手法は、遺伝子配列に変異が起きるようながんなどを対象に、全遺伝子レベルのゲノム診断への貢献が期待されています。
この手法は、研究チームが発表した論文や実験手順書を参考に、世界中で利用されていますが、より簡便に安定して利用できるよう試薬キットの市販化が求められていました。
- 注1)Tetsutaro Hayashi, Haruka Ozaki, Yohei Sasagawa, Mana Umeda, Hiroki Danno and Itoshi Nikaido, "Full-length total RNA sequencing from single cells uncovers dynamics of recursive splicing and enhancer RNAs", Nature Communications, doi: 10.1038/s41467-018-02866-0
- 注2)2018年2月14日プレスリリース「1細胞から多種多様なRNAのふるまいを計測 -1細胞完全長トータルRNAシーケンス法の開発に成功-」
研究手法と成果
既存の1細胞RNA-seq法では、1細胞が持つごくわずかなRNAをDNAシーケンサーで検出するため、逆転写反応[7]と分子の増幅が必要になります。オリゴdTプライマー[8]のみを用いて逆転写反応するため、ポリA型RNAしかcDNA合成できず、また途中でcDNA合成が止まった場合、全長の検出が困難でした。一方、RamDA-seqTM法では、Not-so-random(NSR)プライマー[9]を使用してランダムプライミング[10]するため、ポリA配列以外からも合成が可能であり、非ポリA型RNAを検出できます。また、NSRプライマーはRNAの様々な場所に結合し逆転写が開始されるため、RNA全長からシーケンスリードを得られます。さらにRamDA-seqTM法は、逆転写と同時にcDNAが増幅されます。そのため、増幅用のPCR[11]アダプターをcDNAに付加する必要がありません。そのためにアダプター付加の失敗による遺伝子の捉え漏らしや、PCRによる増幅バイアスを抑えられます。
研究チームは、本技術の製品化のため、研究・診断薬メーカーである東洋紡株式会社へ技術指導を行いました。東洋紡株式会社は、理研から本技術の特許実施のライセンスを受け、RamDA-seqTM法の試薬キット「GenNext® RamDA-seqTM Single Cell Kit」および「RT-RamDATM cDNA synthesis Kit」を開発しました(表1、図2)。
![]() 左右にスクロールできます
左右にスクロールできます
| 特性 | 既存の1細胞RNA解析法 | RamDA-seq |
|---|---|---|
| 検出遺伝子の全長性 | 長鎖RNAで全長が捉えられない | 全長をカバーするリードが得られる |
| 実験間のばらつき | 検出漏れや増幅の偏りが発生 | 検出漏れや増幅の偏りが少ない |
| 非ポリA型RNAの検出 | 検出不可 | 検出可能 |
| 検出遺伝子数 | 15,000 | 25,000 |
表1 RmaDA-seqTMの特長
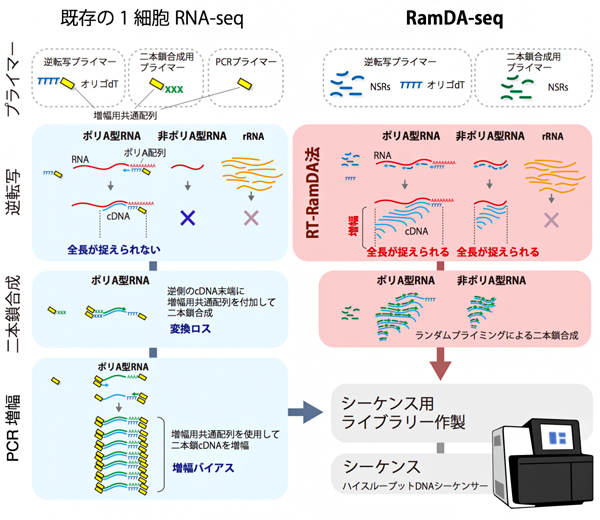 図2 RamDA-seqTMの原理
図2 RamDA-seqTMの原理
今後の期待
本製品の利用により、1細胞レベルの遺伝子発現量の変動やRNA配列の変異を全遺伝子規模で解析することで、基礎研究からゲノム医療など様々な分野の発展に貢献すると期待できます。
一方、研究チームは、これまで数々の1細胞RNA-seq法とそのデータ解析手法の研究開発で、世界をリードしてきました注3、注4、注5)。これらの研究成果をアカデミアだけでなく、産業界を通じて社会にも還元していきます。
- 注3)2013年7月25日プレスリリース「細胞1個の遺伝子発現を網羅的に定量化する「Quartz-Seq法」を開発」
- 注4)2018年3月31日プレスリリース「数千個の1細胞からRNA量と種類を正確に計測 -細胞機能を網羅的・高精度・低コストに同定可能に-」
- 注5)2019年2月25日プレスリリース「高速検索エンジン「CellFishing.jl」を開発 -大規模1細胞データベースから類似細胞を瞬時に検出する手法-」
補足説明
- 1.RNA、ゲノム、遺伝子
ゲノムは細胞が持つ全遺伝情報であり、DNA(デオキシリボ核酸)という生体高分子に4種類の塩基の配列として記録されている。ゲノムの中の遺伝情報が記録(コード)された領域が遺伝子である。遺伝子領域の情報は、DNAを鋳型としてRNA(リボ核酸)が合成(転写)されることで読み出される。 - 2.1細胞完全長トータルRNAシーケンス法『RamDA-seq』
多様なRNAの発現量と全長を1細胞で計測する方法。リボゾーマルRNA(rRNA)を除いたRNAを用いることで、rRNA以外の非ポリA型及びポリA型の全てのRNAとその全長を検出することができるRNAシーケンス法。ヒストンmRNA、長鎖ノンコーディングRNA、新生RNA、環状RNA、エンハンサーRNAなどを検出できる。大量のRNAを用いたトータルRNAシーケンスでは、最初にrRNAを除去するステップがあるが、その際、RNAのロスが生じる。また操作が煩雑となるため、1細胞に適応することは難しい。RamDA-seq(ラムダセック) は、Random Displacement Amplification sequencingの略。 - 3.ゲノム医療
ゲノム情報を利用した個別化医療のひとつ。ゲノム情報に基づき、ひとりひとりの体質に合った治療を行う医療のこと。特に、がんは複数の遺伝子の変異を検査し、治療を行う必要ある。このようながん遺伝子検査は、限られた遺伝子のみを検査するため、診断精度や診断できるがんに限りがある。そのため全遺伝子やRNAを対象として、診断を行う研究がさかんに行われている。 - 4.非ポリA型RNA
タンパク質をコードするmRNAと異なり、RNAの3'末端にポリA配列(アデニン塩基が連続してつながったもの)を持たないRNAの総称。ヒストンタンパク質をコードするヒストンmRNAは例外的にポリA配列を持たない。 - 5.RT-RamDA法
逆転写反応中にRNAから直接、全遺伝子増幅を可能にした新しい核酸増幅法。DNAを鋳型とする従来の増幅法と異なり、増幅用の共通配列を必要としない。このため、逆転写プライマーに増幅用配列を付加する必要がなく高効率な逆転写が可能となる。また、少ないステップで増幅が完了するため、従来法と比べて収率が高く高感度である。RT-RamDAは、Reverse Transcription with Random Displacement Amplification の略。 - 6.1細胞RNA-seq
1細胞中に含まれるRNAを、ハイスループットDNAシーケンサーを用いて配列決定し、網羅的かつ定量的にその量や種類を決定する方法。微量なRNAを用いるため、微量RNAからの逆転写反応とシーケンス可能な量までcDNAを増幅させる二つのステップが必要である。 - 7.逆転写反応
RNAを鋳型とし相補的なDNA(cDNA)を合成する反応。RNAウイルスが宿主細胞に進入し自己複製する際に働くRNA依存性DNA合成酵素である逆転写酵素を利用する。逆転写酵素がcDNA合成を行うには、RNA配列に相補的に結合した短いDNA鎖(逆転写プライマー)を出発点とする必要がある。 - 8.オリゴdTプライマー
逆転写プライマーの一つ。ポリA型RNAの末端に付加されるポリA配列に結合するチミン塩基が20個程度連続的につながったDNA鎖。非ポリA型RNAには結合できない。 - 9.Not-so-random(NSR)プライマー
ランダムプライマーの一種で、ランダムプライマーからリボゾーマルRNA(rRNA)を認識する配列を除いたプライマー。これを用いて逆転写反応を行うとrRNAからのcDNA合成を抑制できる。rRNAは、細胞内に存在するRNAの9割以上を占めるが、どのような細胞でも存在しており、計測しても細胞種特異的な機能はわからない。 - 10.ランダムプライミング法、ランダムプライマー
ランダムプライマーは逆転写プライマーの一つ。アデニン、チミン、シトシン、グアニンの4種類の塩基がランダムに6塩基がつながったDNA鎖。RNAの配列に関係なく結合できるためポリA型RNAと非ポリA型RNA両方に結合することができる。また、RNAの末端だけでなく、RNAの全面に結合することができる。ランダムプライミング法はランダムプライマーを用いた逆転写反応で、RNAの捉え漏れの低減やRNAの全長をcDNAに変換できるメリットがある。 - 11.ポリメラーゼ連鎖反応(PCR)
熱耐性を持つDNA依存性DNA合成酵素を用いて、鋳型DNAとPCRプライマーの結合、相補DNAの合成、二本鎖DNAの解離の3ステップを繰り返すことで、DNAを連続的かつ指数関数的に増幅させることができる反応。鋳型DNAの長さや配列に依存した増幅バイアスが生じる。また、短いDNAは増えやすく、長いDNAは増えにくい。さまざまな種類のRNAを由来とするcDNAを増幅させるには、PCRプライマーが結合するための共通配列を予め逆転写プライマーに付加させておく必要がある。PCRはPolymerase Chain Reactionの略。
研究チーム
理化学研究所 生命機能科学研究センター バイオインフォマティクス研究開発チーム
技師 林 哲太郎(はやし てつたろう)
基礎科学特別研究員 尾崎 遼(おざき はるか)
上級研究員 笹川 洋平(ささがわ ようへい)
テクニカルスタッフI 梅田 茉奈(うめだ まな)
チームリーダー 二階堂 愛(にかいどう いとし)
研究支援
本技術開発の研究は、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業(創薬等支援技術基盤プラットフォーム事業:PDIS)「超微量RNAシーケンス技術の支援と高度化」、再生医療実現拠点ネットワークプログラム「iPS・分化細胞集団の不均質性を1細胞・全遺伝子解像度で高速に測定する技術の開発」の支援を受けて行われました。また、本研究の一部は、科学技術振興機構(JST)戦略的創造研究推進事業CREST「統合1細胞解析のための革新的技術基盤」の研究課題「臓器・組織内未知細胞の命運・機能の1細胞オミクス同時計測」の支援を受けました。
発表者
理化学研究所 生命機能科学研究センター バイオインフォマティクス研究開発チーム
技師 林 哲太郎(はやし てつたろう)
チームリーダー 二階堂 愛(にかいどう いとし)
 林 哲太郎
林 哲太郎
 二階堂 愛
二階堂 愛
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
産業利用に関するお問い合わせ
AMED事業に関するお問い合わせ先
日本医療研究開発機構(AMED)
創薬戦略部 医薬品研究課
Tel: 03-6870-2219 / Fax: 03-6870-2244
E-mail:20-DDLSG-16 [at] amed.go.jp
戦略推進部 再生医療研究課
Tel: 03-6870-2220 / Fax: 03-6870-2243
E-mail:saisei [at] amed.go.jp
※上記の[at]は@に置き換えてください。
