2009年3月11日
理化学研究所
タンパク質の立体構造の解明を加速する新規技術の開発に成功
-大腸菌でヨード原子を含む人工アミノ酸をタンパク質に組み込むシステムを開発-
ポイント
- ヨードチロシンを組み込む大腸菌タンパク質合成システムを世界で初めて確立
- タンパク質のX線結晶構造解析に簡易な新手法を提供
- タンパク質の立体構造解析を加速し、創薬開発の構造薬理学に貢献
要旨
独立行政法人理化学研究所(野依良治理事長)は、大腸菌を使って人工アミノ酸をタンパク質に導入する新規技術を確立し、大量生産したタンパク質を活用し結晶化することでX線結晶構造解析を容易にするシステムの開発に成功しました。これは、理研生命分子システム基盤研究領域の横山茂之領域長、村山和隆研究員(現・東北大学准教授)、拡張遺伝暗号システム研究チームの坂本健作チームリーダーらの成果です。
多くの薬は、標的タンパク質に「鍵と鍵穴」のように結合し、その働きを変化させることで作用します。タンパク質上の「鍵穴」に相当する部分の立体構造が分かると、その形に合わせて薬を開発することができるため、標的となるタンパク質の立体構造は、薬を開発する上でとても重要な情報になります。タンパク質の立体構造を知るために広く用いられているのが、タンパク質を結晶化し、結晶にX線を照射する「X線結晶構造解析」と呼ばれる手法です。このとき、構造解析の目印になる原子をタンパク質に導入します。今回開発した技術では、これまでに用いられてきたセレン※1原子やイオウ原子よりも3~10倍強いシグナルを発し、セレン原子のような毒性がなく扱いやすいヨード原子を目印として用いました。本研究では、大腸菌でのタンパク質合成において、ヨード原子をヨードチロシンという人工アミノ酸の形で部位特異的に取り込ませることに成功しました。この技術は、ヨードチロシンに親和性の高い、古細菌由来の酵素の変異体を用いることで初めて可能になりました。今回開発したシステムを用いると、通常の実験室に設置可能な小型のX線発生装置でも、タンパク質の立体構造を解析することができます。また、今回の技術は大腸菌を使うため、ヨードチロシンを取り込んだタンパク質を、通常の研究室の設備で大量に作ることが可能です。このため、多くの研究者が設備の制約を受けず、安価で実行できる研究手段を手に入れることができるようになります。これにより、タンパク質の立体構造解析が加速し、薬の開発やさまざまなライフサイエンス研究に貢献するものと期待できます。
本研究成果は、わが国で推進している「タンパク3000プロジェクト」および、「ターゲットタンパク研究プログラム」の一環として行ったもので、米国の科学雑誌『Structure』オンライン版(3月10日付け:日本時間3月11日)に掲載されます。
背景
生命を創造し、維持する生命現象の基本となっているタンパク質を理解するためには、その形(立体構造)を知ることが大事です。代謝などを担うタンパク質の代表である酵素は、「鍵と鍵穴」の関係によって、作用する化学物質(基質)を見分けて働きかけます。このような酵素と基質との関係は、酵素の立体構造を知ることで初めて正しく理解することができます。また、多くの薬は、タンパク質のさまざまな機能を促進または阻害するなどして、その働きを変化させることで作用します。薬が正しく作用するためには、標的タンパク質だけを選び出し、タンパク質上の「鍵穴」にうまくはまり込むことが必須です。つまり、タンパク質の立体構造を知ることが、薬の開発を大きく前進させることになります。
立体構造を知るには、いくつかの手法があります。一般的に活用されているX線結晶構造解析という方法は、タンパク質を結晶化させ、その結晶にX線を照射して得られるデータを解析し、立体構造を明らかにします。このとき、効率よく、しかも正しくデータを解析するためには、目印となる原子をタンパク質に導入しておく必要があります。現在、タンパク質のX線結晶構造解析では、(1)セレン原子をタンパク質に導入した後に、タンパク質を結晶化させ、(2)放射光施設のような大型設備によって発生させた強力なX線を結晶に照射し、(3)多波長異常分散法※2によって構造を明らかにする手法が、一般的で広く利用されています。近年では、複数のデータ・セットを収集する多波長異常分散法の代わりに、データ・セットを1つだけ収集でき、解析しやすい単波長異常分散法※2も用いられるようになっています。
ヨード原子は、X線の照射によりセレン原子よりも3倍近く強い異常散乱シグナルを発することから、従来から、目印となる原子として注目されてきました。ヨード原子を利用することで、通常の実験室にも設置可能な小型X線発生装置を用いて、単波長異常分散法によって立体構造を解明することが可能になります。さらに、ヨード原子には、セレン原子のような毒性がなく扱いやすいことも特徴となっています。2004年に、米国の研究グループが、ヨード原子を人工アミノ酸の形でタンパク質に導入しX線結晶構造解析を行いましたが、このときタンパク質に導入した人工アミノ酸は、ヨード・フェニルアラニンでした。タンパク質を構成するアミノ酸のフェニルアラニンは、タンパク質内部に多く存在しており、フェニルアラニンにヨード原子を導入することは、タンパク質の構造に影響を与えることが多いため、立体構造解析に適さないと考えられています。そこで、研究チームは、タンパク質の表面に多く存在するアミノ酸のチロシンにヨード原子を導入することを試みました(図1)。これまでに、大腸菌由来のチロシルtRNA※3合成酵素(TyrRS)※4を改変して、チロシンの代わりにヨードチロシンをタンパク質に取り込ませることに成功しています(2008年8月19日プレス発表)。しかし、この大腸菌酵素由来のTyrRS変異体は、真核生物の中ではヨードチロシンを取り込んだタンパク質を生産することができますが、大腸菌の中で働かせると、酵素TyrRSによる正しいタンパク質の合成を阻害するので、大腸菌を使った研究には使用できません。大腸菌は、タンパク質を大量に発現させる手法として、広く安価に利用されています。研究チームは、大腸菌でタンパク質にヨードチロシンを導入することを目指し、古細菌※5由来のTyrRSを改変することを試みました。
研究手法と成果
DNAの情報からタンパク質を合成※6するためには、コドン・tRNA・酵素を含むさまざまな分子が1つのまとまったシステムを構成し、生物の中で正しく機能することが必要です。そのため、ヨードチロシンという新しい人工アミノ酸の導入が可能で、しかも、タンパク質合成を正しく行うことのできる大腸菌タンパク質合成システムを構築しなければなりません。
- 1.ヨードチロシンを導入できるタンパク質合成システムを確立
研究チームでは、まず古細菌TyrRSのチロシン結合ポケットを構成しているアミノ酸残基を選んで、ほかのアミノ酸に置き換えるという作業を行い、TyrRS変異体を多数作成しました。その中から、チロシンには結合せず、ヨードチロシンとだけ結合する変異体「iodoTyrRS-mj」を選び出すことに成功しました。さらに、この変異体の立体構造を解明し、iodoTyrRS-mjがどのようにしてヨードチロシンを識別しているのかを明らかにすることができました(図2)。ヨードチロシンのヨード原子は、チロシン結合ポケットにある5つのアミノ酸残基の間にはまり込むようにして、酵素に結合しています。このような相互作用は、ヨード原子を持っていないチロシンの場合には見られないため、チロシンがほとんどiodoTyrRS-mjとは結合しないことが説明できます。興味深いことに、ヨードチロシンが古細菌のiodoTyrRS-mjに結合する向きと、大腸菌TyrRS変異体に結合する向きは、ちょうど反対になっていることも分かりました(図3)。
次にiodoTyrRS-mjと、終止コドン※7の1つ、アンバー・コドンに対応するように改変した古細菌チロシンtRNAを発現させた大腸菌を用いて、ヨードチロシンをタンパク質の望みの位置に導入できるかを調べました。ヨードチロシンは、ほかの通常のアミノ酸と同様に大腸菌の生育用培地にあらかじめ加えておきました。このタンパク質を質量分析法によって調べたところ、DNA上のアンバー・コドンの位置に対応したアミノ酸の場所に、ヨードチロシンが正しく組み込まれていることが分かりました。
本研究では、この大腸菌内のタンパク質合成システムに、新たなコドンと、古細菌由来の改変酵素、古細菌のtRNAを追加し、ヨードチロシンをタンパク質に組み込むことができるようなシステムに拡張することができました。 - 2.ヨードチロシンを導入し、構造未知のタンパク質の立体構造を決定
そこで、いよいよ構造の分かっていないタンパク質にヨードチロシンを導入して、X線結晶構造解析によって立体構造を解明する実験を行いました。今回、対象とするタンパク質には、分子量2万程度の比較的小さな分子ながら立体構造が知られていない、高度好熱菌※5の酵素の1つである「アセチルトランスフェラーゼ」を選びました。このタンパク質は、5箇所にチロシン残基を持っていますが、3箇所についてそれぞれヨードチロシンに置き換えた酵素を作製できました。このとき、ヨードチロシンに置き換えたいチロシンの位置を、DNA上でアンバー・コドンに変化させています。これら3つのヨードチロシン含有タンパク質をそれぞれ精製して結晶化を行ったところ、2つについて結晶化に成功し、X線結晶構造解析に進みました。その結果明らかとなったヨードチロシン含有タンパク質の立体構造は、ヨードチロシンがいずれもタンパク質の表面に存在していました(図4)。ヨードチロシンに置き換えができなかったチロシン残基は、今回の立体構造解析の結果から、タンパク質の内部に存在していることが分かりました。
さらに、ヨードチロシンを入れずに結晶化した、天然型のアセチルトランスフェラーゼの立体構造も明らかにし、ヨードチロシンの入っている変異体と比較しました。検証の結果、2つの構造はぴったりと一致し、ヨードチロシンを入れたタンパク質の立体構造がもとの構造を変えていないことが確認できました。つまり、ヨードチロシンを導入した構造は、そのまま正しい天然型構造と見なすことができます。
この結果から、チロシンのように比較的タンパク質表面に存在しているアミノ酸をヨードチロシンに置き換えることが、立体構造を知るためには好ましいと分かりました。すでに報告されているヨード・フェニルアラニンを導入するシステムは、フェニルアラニンがタンパク質内部に見られ、構造を変えてしまう可能性が高いことと比べても、ヨードチロシンの優位性が示されたことになります。
今後の期待
本研究で開発したヨードチロシン導入のシステムは、X線結晶構造解析法を用いる広範囲の研究に多くの利点と利用しやすさをもたらすことが期待できます。このシステムを大腸菌で実現したことは、タンパク質を大量に生産することを可能にし、結晶化とX線結晶解析実験を安価に行うことにつながります。実際に、本研究では、大腸菌を含んだ生育培地1リットルあたり数mgのタンパク質、すなわち結晶化してX線結晶解析を行うために十分な量のタンパク質を得ることができました。また、毒性のないヨードチロシンを使用することで安全性を高め、廃液処理なども容易に行うことが可能となりました。
本システムで明らかにしたアセチルトランスフェラーゼの立体構造は、検証の結果、ヨードチロシンを導入した構造は天然型構造と見なせることが分かりました。このシステムが標準手法となると、高度な手法と設備を要求されるX線結晶構造解析法が、より身近な研究手法となり、タンパク質の立体構造解析が加速し、医薬品の開発や多くのライフサイエンス研究に貢献するものと期待されます。
発表者
理化学研究所
生命分子システム基盤研究領域 領域長
東京大学大学院理学研究科生物化学専攻 教授
横山 茂之(よこやま しげゆき)
Tel : 045-503-9196 / Fax : 045-503-9195
拡張遺伝暗号システム研究チーム
チームリーダー 坂本 健作(さかもと けんさく)
Tel : 045-503-9459 / Fax : 045-503-9458
お問い合わせ先
横浜研究推進部 企画課Tel : 045-503-9117 / Fax : 045-503-9113
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.セレン
元素番号34、元素記号はSe。セレンはごく微量だけ人体に必要とされるが、必要量を超えて摂取すると毒性を示す。X線結晶構造解析では、メチオニンの中のイオウ原子がセレン原子に置き換わったセレノメチオニンを使用するが、使用する量は明らかに毒性を示す量である。 - 2.多波長異常分散法、単波長異常分散法
タンパク質の結晶にX線を照射すると、結晶を通過することで回折した光のパターンを得ることができる。このデータからタンパク質の立体構造を解明するためには、回折した光の位相を決める必要があるが、タンパク質中に導入した重金属原子を手がかりにして位相を決定する。異なる波長のX線を利用して得られる複数のデータ・セットから立体構造を解明する方法を多波長異常分散法と呼ぶ。1つの波長のX線を用いて得られるデータ・セットから構造を解明する方法が単波長異常分散法である。1つのデータ・セットを得るだけでよいという点で単波長異常分散法は有利である。 - 3.tRNA
転移リボ核酸の略号。1つの生物種には、アミノ酸ごとに1~数種類のtRNAが存在する。tRNAはそれぞれ対応するアミノ酸を末端に結合し、リボソームまで運搬する。リボソームはタンパク質の工場であり、tRNAによって運び込まれたアミノ酸をつなげてタンパク質の生産している。 - 4.チロシルtRNA合成酵素(TyrRS)
チロシンに対応するtRNAに、チロシンを結合させる酵素がチロシルtRNA合成酵素である。TyrRS(tyrosyl-tRNA synthetase)と省略される。それぞれのアミノ酸に対してこのような合成酵素が存在する。 - 5.古細菌、高度好熱菌
すべての生物は、真核生物、真正細菌、古細菌の3つのグループに分類される。古細菌の多くは温泉や海底などの極端な環境で生育しており、酵素の性質などがほかの2つのグループと異なることが多い。真正細菌の中にも、温泉などの高温下で増殖するものがおり、これらは高度好熱菌と呼ばれている。 - 6.タンパク質合成のしくみについて
タンパク質はアミノ酸がつながった分子で、どのアミノ酸がタンパク質の中のどの位置に現れるかは、DNA配列によって決まる。DNAの中で、1つのアミノ酸に対応する3つの塩基を1単位として「コドン」と呼ぶ。コドンとアミノ酸を橋渡しするのがtRNAで、例えば、チロシンというアミノ酸の場合、チロシンに対応したtRNA(チロシンtRNA)がタンパク質合成で行っている配列上の位置にチロシンを運ぶ。そこでチロシンtRNAは、チロシンに対応したコドンを読み取って、チロシンを1つ前のアミノ酸に結合する。TyrRSは、あらかじめチロシンtRNAに、チロシンを結合させておく働きをする。
本研究では、ヨードチロシンをtRNAに結合させるiodoTyrRS-mjを作り、このiodoTyrRS-mjと同様に、古細菌由来のチロシンtRNAの両方を大腸菌の中で発現させることで、ヨードチロシンをタンパク質中に組み込むことを実現した。ただし、チロシンtRNAをそのまま使うと、チロシン・コドンの位置でヨードチロシンがタンパク質に組み込まれるため、古細菌のチロシンtRNAを改変して、どのアミノ酸にも対応付けられていない終止コドンの1つ、アンバー・コドンを読み取るようにし、アンバー・コドンの位置でヨードチロシンがタンパク質に組み込まれるようにした。 - 7.終止コドン
DNAは4つの文字(塩基)が並んだ物質だが、タンパク質が合成されるときは、3文字ずつに区切ってアミノ酸と対応付けられている。この3文字のまとまりをコドンと呼ぶ。コドンの中には、アミノ酸とは対応付けられていない3つのコドンがあり、この3つのコドンは終止コドンと呼ばれ、タンパク質の合成を終了させる目印になっている。3つの終止コドンにはそれぞれ名前がつけられており、本研究で使用しているアンバー・コドンはその1つである。アンバー・コドンを人工アミノ酸のコドンとして使うとき、タンパク質を終了させるためには、ほかの2つの終止コドンを使用する。
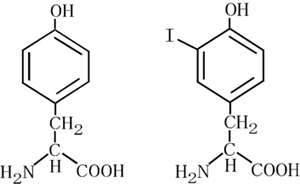
図1 チロシン(左)とヨードチロシン(右)の化学構造
Iがヨード原子。
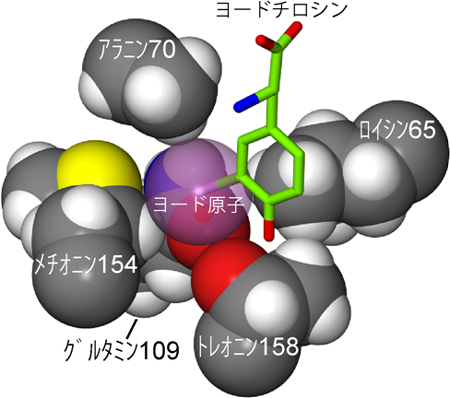
図2 ヨード原子が酵素のアミノ酸残基にぴったりと囲まれている様子
ヨード原子に接している酵素(iodoTyrRS-mj)の5つのアミノ酸残基(アラニン70、メチオニン165、グルタミン109、トレオニン158、ロイシン65)を抜き出して表示した。アラニン70とは、「酵素中の70番目の位置にあるアラニン」を意味している。これらのアミノ酸残基は充填モデルで表示されている。ヨードチロシンは、逆立ちした形で、緑色のスティック・モデルで表示されている。ヨード原子の部分だけは紫色で原子半径を示した。ヨード原子がアミノ酸残基とぴったり接していることが分かる。
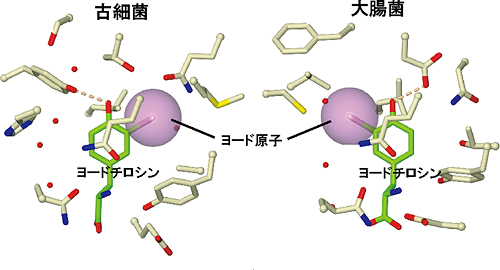
図3 2つのTyrRS変異体に結合したヨードチロシン
古細菌由来のTyrRS変異体(iodoTyrRS-mj)(左)と大腸菌TyrRS変異体(右)のアミノ酸結合のポケットと、ポケットに結合したヨードチロシン分子を示した。白いスティックで示された部分は酵素の一部分であり、結合ポケットを形作っている。緑のスティックはヨードチロシンであり、その中のヨード原子は紫の球で原子半径を示している。この図では、古細菌と大腸菌のTyrRS変異体の結合ポケットを同じ向きから眺めている。ヨード原子が、この2つの変異体で、反対向きにポケットに収まっていることが分かる。
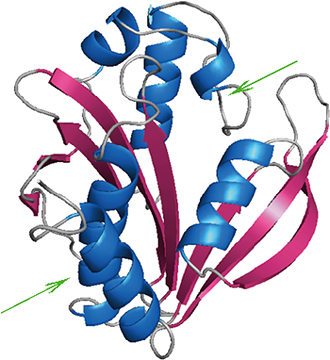
図4 ヨードチロシンの導入によって解明したアセチルトランスフェラーゼの立体構造
2次構造単位を色分けして示している。青がαへリックス、赤がβシートである。構造解析に成功した変異体タンパク質のヨードチロシンの導入位置を矢印で示す。
