2010年9月30日
独立行政法人 理化学研究所
国立大学法人 東京大学大学院理学系研究科
tRNAにわざと誤ったアミノ酸を付加して修正する巧妙な仕組みを解明
-tRNAに直接に付加できないアミノ酸を利用するためのリスク管理機構を発見-
ポイント
- グルタミンの付加されたtRNAを生成する巨大複合体の立体構造解析に成功
- 第1の酵素が誤ったアミノ酸をtRNAに付加する瞬間を初めてとらえる
- 誤りを速やかに修正するために第2の酵素がすぐそばで待機していることを発見
要旨
独立行政法人理化学研究所(野依良治理事長)と国立大学法人東京大学(濱田純一総長)は、グルタミンというアミノ酸を運ぶ役割の転移RNA(tRNA)※1と、それに働く2種類の酵素が巨大複合体「グルタミン・トランスアミドソーム」を作ることを発見し、X線結晶構造解析によって、まず第1の酵素がtRNAにグルタミン以外のアミノ酸を付加し、第2の酵素がこの誤りを素早く修正してグルタミンに変換するという巧妙なメカニズムを持つことを詳細に解明しました。これは、理研生命分子システム基盤研究領域の横山茂之領域長(東京大学大学院理学系研究科構造生物学社会連携講座兼任教授)、東京大学大学院理学系研究科構造生物学社会連携講座の伊藤拓宏特任助教(理研生命分子システム基盤研究領域システム研究チーム客員研究員)による研究成果です。
生物は、20種類のアミノ酸を材料にタンパク質を合成しています。各アミノ酸は、それぞれに対応する転移RNA(tRNA)に付加され、リボソーム(タンパク質合成工場)へと運搬されて、タンパク質合成に用いられます。アミノ酸をtRNAに付加するアミノアシルtRNA合成酵素(aaRS)※2は、アミノ酸ごとに用意され、アミノ酸とtRNAとが正しいペアを生成するように働きます。しかし、多くの真正細菌や古細菌※3では、アミノ酸の一種であるグルタミン(Gln)のaaRS(GlnRS)が存在しません。代わりに、別のアミノ酸であるグルタミン酸(Glu)のaaRS(GluRS)は、その働きが拡張され、tRNAGluにもtRNAGlnにも区別せずに働き、tRNAGlnにとっては誤ったアミノ酸であるGluを付加する第1の酵素として機能し、わざと誤ったペア(Glu-tRNAGln)を作ります。続いて、GatCAB※4という第2の酵素が、tRNAGln上でGluを正しいアミノ酸(Gln)へと修正します。しかし、この方式では、GatCABの働きに過不足があると、Glu-tRNAGlnやGln-tRNAGluという誤ったペアがタンパク質合成に使われるという2重のリスクを抱えます。また、Glu-tRNAGlnは不安定なため、アミノ酸の修正を素早く行う必要があります。
今回の研究では、tRNAGlnとGluRS、GatCABの3者が安定した巨大複合体『グルタミン・トランスアミドソーム』を形成することを発見しました。さらに、この3者複合体の中で、第1の酵素であるGluRSが誤ったアミノ酸(Glu)をtRNAGlnに付加している様子、第2の酵素であるGatCABが、tRNAGluにはなくtRNAGlnのみにある特徴を認識して強固に結合している様子、GatCABが、GluRSのすぐそばに位置し、誤ったペアGlu-tRNAGlnが生成されると直ちに修正できるように待ち構えている様子、などを初めて3Dでとらえ、2重のリスクを回避する巧妙な仕組みを明らかにしました。さらに、GluRSとGatCABが協調して構造を変化させ、素早く修正できることも分かりました。
グルタミンは、生命の進化の過程で活用されるようになった比較的新しいアミノ酸と考えられています。今回の成果は、生命がグルタミンをタンパク質の構成因子として獲得したメカニズムを検証する基盤や、新しい機能を持ったアミノ酸をタンパク質に組み込む技術基盤を提供します。また、GatCABがピロリ菌などの病原菌の生育に欠かせない酵素であることから、新たな抗菌剤開発への応用も期待できます。
本研究成果は、「ターゲットタンパク研究プログラム」、および「日本学術振興会 科学研究費補助金 基盤研究(A)」の一環として行われたもので、英国の科学雑誌『Nature』(9月30日号)に掲載されます。
背景
遺伝情報に基づいて作られるタンパク質の生成は、生命の根幹をなす重要な反応です。DNAに書き込まれている遺伝情報は、まず伝令RNA(mRNA)へと転写されます。mRNA中の3文字の核酸配列(コドン)は、リボソーム上で1つのアミノ酸へと変換され、アミノ酸の連なったタンパク質が合成されます。mRNA中のコドンと1つのアミノ酸を対応させている分子がtRNAです。20種類のアミノ酸には、それぞれに対応するtRNAが存在します。ヒトを含む真核生物と一部の細菌では、アミノ酸ごとに対応する20種類のアミノアシルtRNA合成酵素(aaRS)が存在し、対応するtRNAにアミノ酸を正確に付加することで、正しいペア(アミノアシルtRNA)を生成します。この機構によって遺伝情報に対応した正確なタンパク質合成が保障されています。
しかし、多くの真正細菌、古細菌では、20種類のaaRSのうち、アミノ酸の一種であるグルタミン(Gln)をtRNAGlnに付加するaaRS(GlnRS)が存在しません。代わりに、グルタミン酸(Glu:Glnのアミノ基(-NH2)がヒドロキシル基(-OH)となっている)を材料とした2段階の反応でGlnが付加されたtRNAGln(Gln-tRNAGln)を作ります。そのような生物では、まず、Gluに対応するaaRS(GluRS)の働きが拡張されてtRNAGluにもtRNAGlnにも区別せずに働き(非識別型GluRS)、tRNAGlnにとっては「誤った」アミノ酸であるGluを付加して、Glu-tRNAGlnという誤ったペアを中間体として生成します。次に、GatCABというアミド基転移酵素※5が、tRNAGln上でGluのヒドロキシル基をアミノ基へと変換することによってGluをGlnへと修正し、正しいペアであるGln-tRNAGlnを最終産物として生成します。これは、わざと間違えて修正するというユニークなメカニズムです。
一方で、この方式は、GatCABが、tRNAGlnにのみ働き、tRNAGluには働かないことが必要であり、もしもGatCABの働きに過不足があると、生物は2重のリスクを抱えることになります。1つは、中間体のGlu-tRNAGlnがリボソームへと運搬されることで、Glnの位置にGluが導入されて誤ったタンパク質を合成してしまうことです(①)。もう1つは、GatCABが間違えてGlu-tRNAGluに作用することで、Glu-tRNAGluのGluがGlnに変換され、Gln-tRNAGluができ、逆にGluの位置にGlnが導入されてしまうことです(②)。GluはGlnとは異なり、マイナスの電荷を持ち、両者はタンパク質中でまったく違う役割を持つため、でき上がった異常なタンパク質が生物にとって致命的な事態を招くことになりかねません。生物が、このようなリスクを冒してまでGlnを利用しようとするほど、アミノ酸の1つ1つは大切な役割を持っていて、その利用にはリスクを回避する巧みなリスク管理のメカニズムが必要となります。
また、中間体のGlu-tRNAGlnは、生体内では不安定であることが知られており、GatCABによる修正は迅速に行われなければなりません。緩慢な修正をしていては、中間体が分解され、エネルギーを浪費することになります。従って、正しいペアであるGln-tRNAGlnが生成される過程には、何らかの特別な仕組みが存在することが予想されていましたが、具体的な証拠はありませんでした。
今回、研究グループはtRNAGlnとそれに働く2種類の酵素GluRS、GatCABを用いて、Gln-tRNAGlnの生合成メカニズムの解明に挑みました。
研究手法と成果
研究グループは、真正細菌(Thermotoga maritima:サーモトーガ・マリティマ)由来のtRNAGlnとGluRS、GatCABを用い、ポリアクリルアミドゲル電気泳動※6により、それらの結合状態を解析する実験を行いました。その結果、3者が安定した巨大な複合体を形成することを発見し、これを『グルタミン・トランスアミドソーム』と命名しました。
次に、グルタミン・トランスアミドソーム(以下、トランスアミドソーム)の3次元(3D)構造を明らかにするために、X線結晶構造解析に適した結晶を作製することに成功し、大型放射光施設のSPring-8※7のビームラインBL41XUと高エネルギー加速器研究機構(KEK)のPhoton FactoryでX線回折実験を行い、トランスアミドソームの3D構造を解明しました。
トランスアミドソームの3D構造から、実際にGluRSとGatCABがtRNAGlnに対して同時に結合し、トランスアミドソームを構築している様子を詳細に明らかにすることができました(図1)。また、この3D構造では、GluRSがtRNAGlnと相互作用し、「誤ったアミノ酸」であるGluをtRNAGlnに付加しようとしている瞬間、つまり、放置すれば、中間体のtRNAGlnがリボソームへと運搬されてしまうリスク(①)を冒している瞬間を初めてとらえることに成功しました。この相互作用様式から、非識別型GluRSが、本来の基質ではないtRNAGlnを、本来の基質であるtRNAGluと同じように認識してアミノアシル化できる仕組みが明らかになりました。
一方、トランスアミドソーム中のGatCABは、そのテールボディ※8が、tRNAGlnのL字型の外側のDループと呼ばれる部分の構造の違いを手がかりに、Glu-tRNAGlnとGlu-tRNAGluとを見分けていることを発見しました。また、テールボディ以外の触媒ボディ※8は、tRNAGlnに働きかけるために接してはいませんでしたが、その一部がGluRSと相互作用し、「留め金」がかかったように固定されていました。すなわち、今回とらえたトランスアミドソームのGatCABは、GluRSがtRNAGlnにGluを付加しようとしているときに、GluRSから中間体のGlu-tRNAGlnを受け渡されるのを待ち構えている状態であることが分かりました。これらの結果は、GatCABが、同じGluRSの作り出すGlu-tRNAGlnおよびGlu-tRNAGluのうち、Glu-tRNAGlnを、Gln-tRNAGlnへと確実に修正する一方で、働いてはいけないGlu-tRNAGluは修正しないように管理していることを示しています。こうして、中間体のGlu-tRNAGlnがリボソームへと運搬されてしまうリスク(①)とGatCABが作用すべきGlu-tRNAGlnをGlu-tRNAGluと間違えるリスク(②)の2重のリスクを回避する巧妙な仕組みを明らかにしました。
GluRSの反応が完了した後にGlnへと修正されるべきGluは、tRNAGlnの末端部分に付加されていますが、どのようにしてGatCABがその末端部分を受け取るのでしょうか。GatCABとGluRSの構造情報を詳しく調べると、GatCABとGluRSのそれぞれに、蝶番(ちょうつがい)のような構造を見いだしました。すなわち、GatCABとGluRSは、蝶番で曲がることによって、tRNAGlnに結合したまま(トランスアミドソームを形成したまま)で、触媒ボディを動かすことができ、Glu-tRNAGlnの末端部分から遠ざけたり、近づけて働かせたりすることができるという予想外のメカニズムの存在を発見しました(図2)。これにより、素早く確実な修正が可能になっていると考えられます。
これまでのGluRS、tRNAGln、GatCABに関する立体構造研究から、1分子のtRNAGlnの末端部分に対して、GluRSとGatCABが同時に結合しようとすると互いに衝突してしまうことが分かっていました。そのため、GluRSがtRNAGlnから離れた後でGatCABが結合すると考えられており、「トランスアミドソーム」のような3者複合体が継続的に形成されることはないと思われていました。しかし実際には、GluRSとGatCABの蝶番構造により、互いに協調し合うことでトランスアミドソームの形成が可能であり、今まで機能不明だった部分構造は、「留め金」としてトランスアミドソームを安定化していることが分かりました。
これらをまとめると、GatCABはGluRSおよびtRNAGlnとトランスアミドソームを形成することによって、リスクを管理し、巧妙にGln-tRNAGlnを作りだします。まず、GluRSがtRNAGlnに「誤った」アミノ酸であるGluを付加しているときには、GatCABがすぐそばで待機していて、確実に修正できるようにすることにより、Glu-tRNAGlnを間違って手放してしまうリスクを回避しています。GluRSは、中間体のGlu-tRNAGlnを生成すると、自分の触媒ボディを素早く移動させ、グルタミン酸の付加されたtRNAの末端部分から離れます。続いて、待機しているGatCABがその触媒ボディをさらに近づけて、tRNAの末端部分を受け取り、GluをGlnへと修正します。一方、GluRSが本来の基質であるtRNAGluに「正しい」アミノ酸であるGluを付加しているときには、GatCABは結合せず(トランスアミドソームを形成せず)、過剰な間違った修正を起こすリスクも回避していました。こうして、正しいペアであるGln-tRNAGlnを生成するための、このような巧妙なメカニズムの存在を初めて明らかにすることができました(図3)。
今後の期待
今回の研究では、Gln-tRNAGlnという正しいペアが生成される過程で中心的な役割を果たすトランスアミドソームが存在することを発見し、その立体構造を明らかにしました。また、トランスアミドソームに含まれる2種類の酵素(GluRSとGatCAB)は、互いに協調するための構造変化が可能な設計になっており、酵素間でのダイナミックな基質tRNAGlnの受け渡し機構を明らかにすることができました。この発見は、基質となる1つの生体高分子に連続反応を担う2つの酵素が同時に結合して連携する状態の立体構造を初めて明らかにしたという点でも大きな意義があり、今後の多くの研究の基礎となります。
このような複雑なメカニズムを用い、「2重のリスク」を冒してまで、グルタミンというアミノ酸をタンパク質に組み込む必要があることは、タンパク質の機能を発揮するために、アミノ酸の種類にさまざまなバラエティーがあることの重要性を強く示しています。グルタミンは、生命の進化において比較的新しくタンパク質合成に用いられるようになったアミノ酸であると考えられており、生命が新たなアミノ酸をタンパク質の構成成分として獲得したメカニズムを検証するための基盤となります。グルタミン以外にも、特殊な元素であるセレンを含むアミノ酸(セレノシステイン)の生合成※9を行う際に、「誤ったアミノ酸をわざと付加してから修正する」という「2重のリスクを冒す」という戦略がとられており、各段階の酵素反応の仕組みは明らかになりつつあります。しかし、複数の酵素が連携して働く仕組みはまったく明らかにはなっていません。そのような研究にも、今回の研究成果が基礎として大きく役立つと期待されます。
また今回、進化の過程で生物が必要に応じて利用可能なアミノ酸の種類を増やしていくメカニズムの一端を解明したことで、工学的な応用ができるようになります。すなわち、タンパク質の生合成においてさまざまなアミノ酸を利用可能にすることによって、新しい機能を担うタンパク質を創製する基盤技術開発や、それらを通じた新しいタンパク質医薬などの有用タンパク質の開発にもつながると期待できます。現在は、利用したい有用アミノ酸を直接tRNAに結合させる新たな特異性を持つaaRSを作りだすタンパク質工学的な研究開発が行われていますが、今後は、「誤ったアミノ酸を付加してから修正する」という戦略をとった研究開発が行われることが考えられます。
また、GatCABと「非識別型」のGluRSは、ピロリ菌などの病原菌の増殖に欠かせない酵素で、ヒトなどの真核生物には存在しないことから、それらをターゲットとする新たな作用機構の抗菌剤開発への応用が期待されます。
発表者
理化学研究所
生命分子システム基盤研究領域
領域長 横山 茂之(よこやま しげゆき)
Tel: 045-503-9196 / Fax: 045-503-9195
お問い合わせ先
横浜研究推進部 企画課Tel: 045-503-9117 / Fax: 045-503-9113
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.転移RNA(tRNA)
転移リボ核酸の略号。DNAから転写されて生成したmRNAには、塩基配列であるアデニン(A)、グアニン (G)、シトシン(C)、ウラシル(U)のうち3つが1組(コドン)となって1つのアミノ酸がコードされている。コドンとアミノ酸を対応付けるアダプター分子がtRNAである。tRNAはL字型の立体構造をしており、L字の角の外側の部分はDループおよびTループと呼ばれる領域が形作っている。L字型の一番先にはコドンと塩基対合することができるアンチコドンが存在し、反対側の先に位置するA76リボースに、対応するアミノ酸が付加された後に、リボソームに運ばれる。mRNAのコドンとtRNAのアンチコドンがリボソーム上で対合すると、tRNAに結合していたアミノ酸同士が次々とつながり、タンパク質が合成される。また、20種類のアミノ酸には、それぞれに対応するtRNAが存在する。ここでは、例えば、グルタミン(Gln)に対応するtRNAは、tRNAGlnと記した。 - 2.アミノアシルtRNA合成酵素(aaRS)
タンパク質を構成するアミノ酸は主に20種類存在する。20種類のアミノ酸のそれぞれに対してアミノアシルtRNA合成酵素が20種類存在し(アスパラギン酸に対応するAspRS、リシンに対応するLysRSなど)、ATPのエネルギーを利用してアミノ酸を活性化したのち、対応するtRNAのCCA末端に付加する(アミノアシル化)。 - 3.真正細菌、古細菌
すべての生物は真核生物、古細菌、真正細菌の3つのグループに分類される。真核生物の特徴は細胞内に核を持ち、細胞のそれ以外の部分からは膜で仕切られていることである。核の中には遺伝情報が書き込まれたDNAが収められている。一方、核を持たない生物は、古細菌と真正細菌とに分類され、地球上のあらゆる環境に生息しており、その代謝系は多種多様である。古細菌は、その遺伝情報が真正細菌よりも真核生物に近いといった特徴を持ち、極限環境に生息しているものが多い。真正細菌とはいわゆる細菌(バクテリア)のことで、大腸菌、サルモネラ菌、腸炎ビブリオ菌、コレラ菌、赤痢菌などを含む。 - 4.GatCAB
GatはGlutamyl-tRNA amidotransferase(グルタミル‐tRNAアミド基転移酵素)の略号。GatCおよびGatA, GatBという3つのタンパク質からなるヘテロ3量体であり、GatAはドナーとなる分子からアンモニアを引き抜く活性を、GatBはGatAによって生成したアンモニアとATPを用いてtRNA上のグルタミン酸をグルタミンへと変換する活性を持つ。GatAとGatBはGatCと共に複合体を形成しており、アンモニアを通すためにGatAの活性部位とGatBの活性部位との間にトンネルが存在する。 - 5.アミド基転移酵素
基質のカルボキシル基にアミノ基を転移し、アミド基を生成する反応を触媒する酵素。GatCABの場合には、グルタミン酸側鎖のカルボシキル基をアミド基へと変換し、結果としてグルタミン酸をグルタミンへと変換する。 - 6.ポリアクリルアミドゲル電気泳動
アクリルアミドの重合体であるポリアクリルアミド中で、核酸やタンパク質などを電気泳動し、それらを分離する方法。 - 7.大型放射光施設SPring-8
兵庫県の播磨科学公園都市にある世界最高の放射光を生み出す理化学研究所の施設。SPring-8の名前はSuper Photon ring 8 GeVに由来する。放射光とは、電子を光とほぼ等しい速度まで加速し、電磁石によって進行方向を曲げた時に発生する、絞られた強力な電磁波のこと。SPring-8では、この放射光を用いて、ナノテクノロジー、バイオテクノロジーや産業利用まで幅広い研究を行っている。 - 8.テールボディと触媒ボディ
GatBに存在する2つの蝶番により、GatCABは3つのまとまった領域に分けられる。そのうちtRNAのL字の角の部分を認識しているGatBのヘリカルドメインとテールドメインの2つの領域をあわせてテールボディと命名し、GatCABのそれ以外の部分を触媒ボディと命名した (図1参照)。 - 9.セレンを含むアミノ酸(セレノシステイン)の生合成
21番目のアミノ酸として知られるセレノシステイン(Sec)の生合成経路においても、誤ったアミノ酸をわざと付加してから修正するという戦略がとられている。すなわち、セレノシステインに対応するtRNA(tRNASec)に、まず誤ったアミノ酸であるセリンが付加され、さらにtRNASec上でセリン部分がリン酸化され、最後にセレンを受け取り最終産物であるSec-tRNASecができ上がる(2010年8月13日プレス発表)。
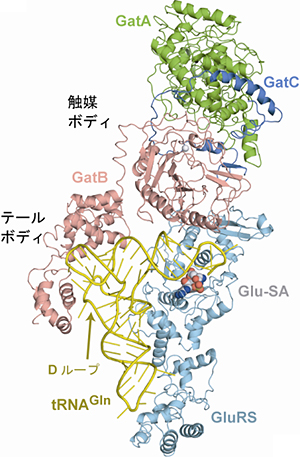
図1 グルタミン・トランスアミドソームの立体構造
tRNAGln(黄色)にGluRS(水色)とGatCAB(緑、青、ピンク)が同時に結合している。GatCABは、そのテールボディにおいて、tRNAGlnを認識している。一方で、触媒ボディはtRNAGlnに接してはいなかったが、その一部は、GluRSと相互作用し、「留め金」がかかったように固定されている。これにより、GatCABがGluRSによって生成される中間体Glu-tRNAGlnを受け渡されるのを待ち構えていることが判明した。
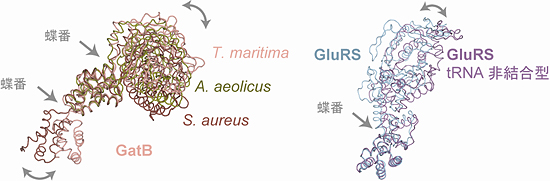
図2 GatBとGluRSに見いだした蝶番構造
解明した3D構造から、それらがどのように生体内で機能を発揮しているかを明らかにするため、3種類の生物種のGatBの構造を重ね合わせたところ、2個所(左図中矢印)の蝶番構造を見いだした。また、tRNA非結合型のGluRSとトランスアミドソーム中のGluRSの構造を重ね合わせたところ、1個所(右図中矢印)の蝶番構造を見いだした。この構造により、GatCABとGluRSは、tRNAGlnに結合したまま(トランスアミドソームを形成したまま)で、触媒ボディを動かすことが可能となる。
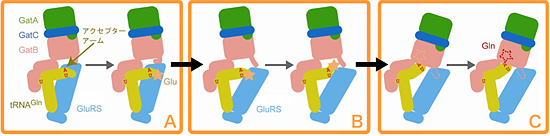
図3 GluRSからGatCABへの反応中間体Glu-tRNAGlnの受け渡し機構
GluRSは中間体のGlu-tRNAGlnを生成する(A)。次にGluRSは、自らの触媒ボディを素早く移動させ、アクセプターアームと呼ばれるGluの付加されたtRNAの末端部分から離れる(B)。その後、待機しているGatCABが、自身の触媒ボディをさらに近づけて、tRNAの末端部分を受け取り、GluをGlnへと修正する(C)。
