2012年9月21日
独立行政法人理化学研究所
網羅的な遺伝子発現情報を3次元空間に描き出す新手法を開発
―トモグラフィーを応用して、遺伝子発現量を迅速・高精度に3次元で再現―
ポイント
- 従来法より検体数1,000分の1、測定時間10分の1、費用100分の1で達成
- ハンチントン病遺伝子の発現量とその疾患による脳障害部位に相関があることを確認
- 36,558種類の遺伝子発現量の立体地図をウェブで公開
要旨
理化学研究所(野依良治理事長)は、網羅的に測定した遺伝子発現情報を3次元空間に再構成する新手法「トランスクリプトームトモグラフィー」を開発し、生体内のどの部分で、どの遺伝子が、どれだけ発現しているかを、ウェブ上の仮想空間で描くことに成功しました。この手法で作成したマウスの脳遺伝子発現データベース(ViBrism-DB)を、9月20日、ウェブ上に公開しました。これは理研基幹研究所(玉尾皓平所長)生物情報基盤構築チームの於保祐子(おほゆうこ)客員研究員(NPO法人BReNt-ブレインリサーチネットワーク代表)、横田秀夫チームヘッド、理研情報基盤センターの姫野龍太郎センター長らによる共同研究グループの成果です。
計算機科学の技術を応用して生物学の問題を解こうとする生物情報科学(バイオインフォマティクス)は、数値化された生命情報を扱います。近年の技術革新により、遺伝子やタンパク質などの分子の発現量は網羅的に測定できるようになりました。しかし、これらの情報を3次元的の画像情報として数値化し表現することは困難でした。共同研究グループが開発したトランスクリプトームトモグラフィーは、独自の生体情報画像化システム「3D-ISM」と、マイクロアレイ※1などの網羅的かつ高速な発現量測定技術を組み合わせたものです。3次元の異なる方向で切り出した切断片の測定結果を、トモグラフィー※2を用いて画像情報とともに仮想空間に再構成します。実際に、マウスのほぼ全ての遺伝子の発現量を脳全体で調べ、36,558種類の発現地図を3次元空間に再構成したところ、神経難病であるハンチントン病※3遺伝子が高発現している分布と、その疾患の影響による脳障害の分布が似ていることが明らかになりました。技術面や費用、作業量などの問題により、組織全体の遺伝子発現量を3次元的にまとめたデータベースはごく限られています。この手法を使うと、従来法に比べ、検体数は1,000分の1、測定時間10分の1、費用100分の1程度で数値情報を3次元化できるため、研究者が各自の研究テーマごとにデータベースを作成し、解析することも可能です。さらに、同じ検体でタンパク質など遺伝子以外の生体内高分子の測定も可能なので、疾患モデル動物の詳細かつ網羅的な解析に応用でき、さまざまな疾患の解明に役立つと期待できます。
本研究成果は、米国のオンライン科学雑誌『PLOS ONE』(9月19日付け:日本時間9月20日)に掲載されました。
背景
生物の設計図にあたる全遺伝情報(ゲノムDNA)の解読が進み、ヒトやマウスなどでは、1つの細胞の中に約数万種類のタンパク質をコードする遺伝子や生命活動に重要な役割を担うRNA分子が存在していることが分かってきました。ゲノムDNAの一部を鋳型にしてRNAが合成されることを転写(トランスクリプション)と呼び、転写によって合成されたRNAのことを転写産物(トランスクリプト)と呼びます。RNAの情報に基づいてタンパク質が合成されますが、一部のRNAは、RNA分子として存在し生命活動を維持するために重要な働きをします。細胞中にはさまざまなRNAがそれぞれの量で存在しており、このようなRNA分子の総体を「トランスクリプトーム」と呼びます。近年の技術進歩により、ごく少数の細胞から、その中にあるほぼ全ての転写産物について網羅的に測定できるようになりました。例えばマイクロアレイやRNA-シークエンス法などの技術を用いると、複雑な形をした臓器や生体全体を対象としてトランスクリプトームを調べることができます。このとき、測定に使った細胞がどこにあったかという位置情報を正確に知る必要がありますが、従来の方法は位置測定が2次元的で、正確な3次元化は困難でした。測定対象をサイコロ状に切断する方法も試みられましたが、位置合わせが困難な上、測定精度を上げようとサイコロのサイズを小さくすると、試料数が膨大になるという欠点がありました。そのため、トランスクリプトーム解析で得られる網羅的なデータを、迅速・高精度に3次元空間に再構成する技術が求められていました。
研究手法と成果
研究チームは、生体の内部構造を観察するための3次元内部構造顕微鏡「3D-ISM」を開発してきました。3D-ISMは、カミソリのような鋭い刃で生体組織試料などの対象を5ミクロン程度にごく薄く切断し、そのときの切断面の画像情報を得る、という繰り返し過程を自動制御するシステムです。この過程を対象全体に繰り返し、得られた数千枚の画像情報をコンピュータ上で積み重ねると、外形と内部の構造を3次元で捉えることができます。出来上がった3次元画像は、コンピュータ上の仮想空間で再現しているにもかかわらず、対象が実空間にある時と同じように、どの方向からのどの断面でも観察することができます。
2003年から、バイオインフォマティクスの方法論を新たに導入して開発したトランスクリプトームトモグラフィーは、3D-ISMの観察過程で切り出されたごく薄い切断片を数百枚ずつ集めて分画とし、これをマイクロアレイなどの網羅的かつ高速な発現量測定技術で解析する新手法です。少なくとも3つの生体組織試料を用意し、縦・横・高さ方向の分画について定量計測してトモグラフィーにより解析すると、遺伝子の発現量について立体的な分布が得られます(図1)。実際に、実験動物として最もよく使われるマウスの脳を用いて、脳全体を1ミリ刻みで平均10分画(6~13分画)に分け、各分画についてマイクロアレイでほぼ全ての遺伝子の発現量を解析、36,558種類の発現分布地図を作成しました(図2)。その精度は、従来の2次元的な方法で作成、公開されているアレンブレインアトラス(Allen Brain Atlas: ABA)とほぼ同程度だったにもかかわらず、作成に要した脳試料数はABAが数千に対し本手法は6つ、費用はABAが数億円に対し本手法は数百万円、時間はABAが1年に対し本手法は1カ月と、格段に効率が良くコストもかからない方法であることが分かりました。また、マウス脳の一部を実際にヒトの手で丁寧に切り取り、その場所の遺伝子発現量を網羅的に測定したブレインスターズ(BrainStars:理研発生・再生科学総合研究センター 機能ゲノミクスユニット, T. Kasukawa, et al, 2011 PLOS ONE)と比較しても、測定結果はよく一致しました。特に、実際に使用した試料数はほぼ同数ですが、ブレインスターズが特定の脳領域だけを対象としているのに対し、本手法は脳全体を同等の精度でくまなく測定することができました。
さらにこの方法は、測定した1つひとつの遺伝子について、同一の3次元空間で定量的に解析することを可能にしました。その結果、難治性の脳疾患であるハンチントン病の遺伝子Httが高発現している分布と、その疾患による脳の障害が起きている部分に相関があること、また、この疾患の進行を遅らせる可能性があると知られている遺伝子Bdnfが多く発現する脳部位は、Httが高発現していてもその障害の程度が低い傾向にあることが分かりました(図3)。
今後の期待
トランスクリプトームトモグラフィーで作成した3次元情報は、MRIなどの他の生物学的情報と同一の3次元空間で解析できます(図4)。特に脳科学の分野では、標準的な脳空間をウェブ上に設定して、そこに多くの情報を再現することで、研究成果を統合して複雑な脳機能の理解を深めようというデジタル脳図譜作成プログラム※4が加速しています。開発した手法は、こうした新しい脳科学研究に、重要な役割を果たすと期待できます。また、研究者が各自の研究テーマごとにトランスクリプトームデータベースを作成し、ウェブ上に公開した標準空間にアップロードすると、研究者間でこれらを比較検討し、解析することも可能となります。これは、現在ゲノム解析について1次元で行われている研究を3次元に拡大するものであり、研究の相乗効果によって革新的な成果が生まれることが予想できます。さらに、同じ検体から、遺伝子以外の生体内高分子の測定も可能で、生命現象や病気の状態を分子レベルで理解することができます。特に、疾患モデル動物などの詳細かつ網羅的な解析に応用できるため、さまざまな疾患の解明に貢献すると期待できます。
原論文情報
- Yuko Okamura-Oho, Kazuro Shimokawa, Satoko Takemoto, Asami Hirakiyama, Sakiko Nakamura, Yuki Tsujimura, Masaomi Nishimura, Takeya Kasukawa, Koh-hei Masumoto, Itoshi Nikaido, Yasufumi Shigeyoshi, Hiroki R. Ueda, Gang Song, James Gee, Ryutaro Himeno, Hideo Yokota “Transcriptome tomography for brain analysis in the web-accessible anatomical space”PLOS ONE Doi: 10.1371/journal.pone.0045373
発表者
理化学研究所
基幹研究所 先端技術基盤部門
生物情報基盤構築チーム
客員研究員 於保 祐子(おほ ゆうこ)
(NPO法人BReNt-ブレインリサーチネットワーク代表)
チームヘッド 横田 秀夫(よこた ひでお)
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.マイクロアレイ
生物の遺伝子発現を網羅的に解析する一般的な方法。塩基配列が知られている多種類の遺伝子のDNAをプローブとして、プレート上に規則正しく貼付けておく。調べたい細胞からRNAを採取し、蛍光標識する。これをプレート上に貼付けられたDNAと反応させ、蛍光強度を読み取ることにより、RNAの発現量を網羅的かつ定量的に調べることができる。 - 2.トモグラフィー
物理探査、医療診断などで用いられる逆解析技術の1つで、その多くは対象領域を取り囲む形で走査線を配置し、内部の物性の分布を調べる。 - 3.ハンチントン病
主に中年以降に発症する常染色体優性遺伝の遺伝性慢性進行性の疾患。臨床症状は主に不随意運動で、知能障害、精神症状を伴う。1983年にハンチントン病遺伝子が第4染色体短腕に存在することが示され、1993年にその原因遺伝子(ハンチンチン遺伝子)が同定された。この原因遺伝子の一部の塩基配列に存在するCAG繰り返し配列が正常より長いために引き起こされる。CAG繰り返し配列が翻訳されるとグルタミン鎖となり、正常より長くなったグルタミン鎖を病因にもつ疾患群をポリグルタミン病と呼ぶ。 - 4.デジタル脳図譜作成プログラム
ウェブ上に仮想の脳空間を設定して、実際の生体から得られた生体高分子情報を仮想の共通空間に記述する。これらをデータベース化し、情報の共有化を図ることで、研究の進展や健康増進につなげようという活動(デジタル脳図譜作成プロプラム:Digital Brain Atlasing Program)が、OECDの報告書に沿ってINCF(国際ニューロインフォーマティクス統合機構)を中心に国際連携として進められている。
日本語での紹介: BReNt-プロジェクト紹介
マウス脳のウェブ上共通空間は最初にプログラム会議が開催されたスウェーデンの小島にちなんで、Waxholm Space (WHS)と呼ばれている。
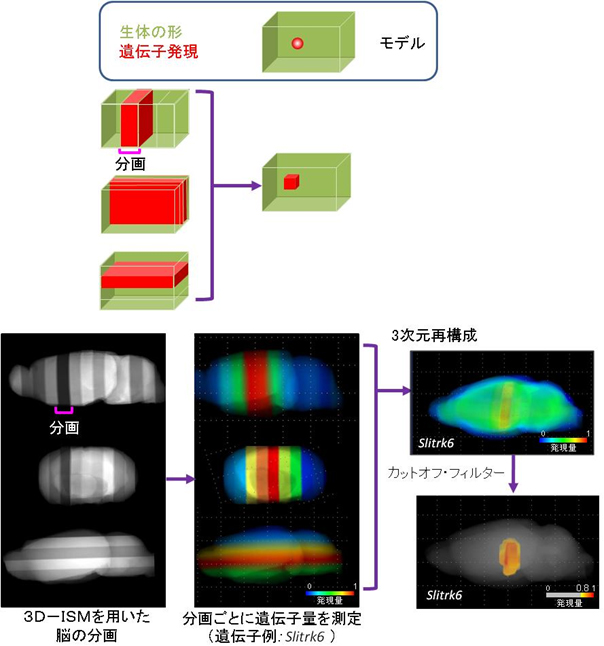
図1 トランスクリプトームトモグラフィーの原理と実画像
- (上)
トランスクリプトームトモグラフィーの原理
数万種類の遺伝子について分画ごとに発現量を測定する。多方向(最少直行3方向)からの結果をトモグラフィーで3次元再構成して分布を推定する。 - (下) 実際にマウスの脳を用いて、遺伝子Slitrk6を測定した例。測定した全ての遺伝子について同様の操作を行い、発現地図を作る。
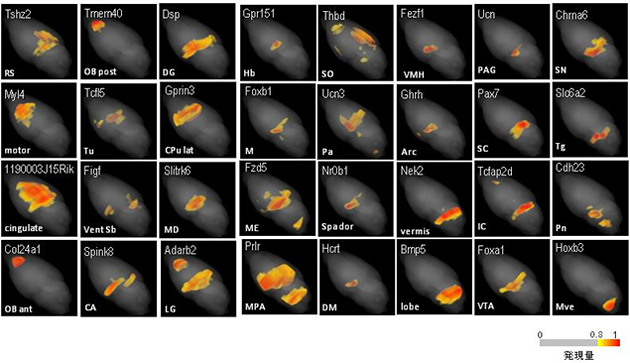
図2 脳を俯瞰する形で見た発現地図
計測した36,558遺伝子発現分布地図のうち、32遺伝子の分布例を示す(パネル上:遺伝子名、下:主な発現の脳領域名)。
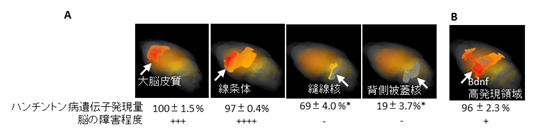
図3 ハンチントン病遺伝子(Htt)発現量と脳障害程度の関係
- A: 遺伝子Httが多く発現する脳部位(赤い部分)と既知の脳の障害程度に相関がある。
- B: 病気の進行を遅らせる可能性のある遺伝子Bdnfが多く発現する脳部位(赤い部分)は、Httが多く発現(96±2.3%)していても障害の程度が低い(+)傾向にある。
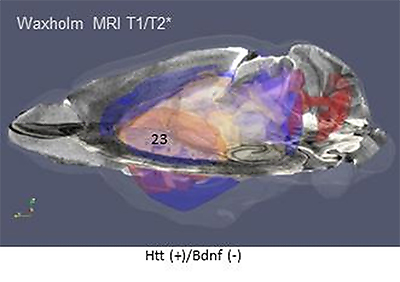
図4 ウェブ上のマウス脳共通空間(WHS: Waxholm Space)でMRI画像に遺伝子発現解析を統合した図
ハンチントン病によって最も障害を受けると考えられるHtt強発現Bdnf低発現領域を、マウス脳MRI画像に基づき脳部位ごとに色分けすると(赤~青)、線条体(23)などの障害部位がどの程度含まれるかが分かる。
図1~4は 2012 Yuko Okamura-Oho et al., PLOS ONE, DOI: 10.1371/journal.pone.0045373を著者改変
