2013年10月3日
理化学研究所
薬の効果がもたらす遺伝子発現の変化を網羅的・定量的に捉える
-抗がん剤投与時のプロモーター活性の変化をゲノム全域で解析することに成功-
ポイント
- 理研独自の「非増幅deepCAGE法」で薬剤作用のプロモーター活性を測定
- 従来法では難しかった弱い薬剤作用を定量的に捉えることに成功
- 未解析薬剤の標的タンパク質や作用機序の解析などに広く応用が可能
要旨
理化学研究所(理研、野依良治理事長)は、薬剤の作用を遺伝子発現量の変化(プロモーター活性[1]の変化)として網羅的・定量的に捉えることに初めて成功しました。これは、理研ライフサイエンス技術基盤研究センター(渡辺恭良センター長)機能性ゲノム解析部門(ピエロ カルニンチ部門長)の鈴木治和グループディレクターらと、理研社会知創成事業予防医療・診断技術開発プログラム(林崎良英プログラムディレクター)の川路英哉コーディネーターらの共同研究グループによる成果です。
新薬開発において創薬候補物質が標的細胞の遺伝子発現をどのように変化させるかを捉えることは、薬理作用を解明するうえで重要です。従来はこの目的のためにマイクロアレイ法[2]が広く用いられてきましたが、検出感度が低く、測定できる遺伝子の種類が限定されるなどの理由で、十分な定量解析が行えませんでした。
理研が開発した独自技法「非増幅deepCAGE法[3]」は、細胞中のmRNAを少量でも偏りなく網羅的に捉え、遺伝子の転写開始点の直上流にあるプロモーター活性を測定できます。さらに、塩基配列を高速に解析できる次世代シーケンサーを用いることで、解析対象の細胞に存在する全mRNAの種類と量が解析可能となります。
共同研究グループは、非増幅deepCAGE法を用いて、抗がん剤を投与する前と投与した後のがん細胞のプロモーター活性を解析しました。その結果、薬剤の作用により個々のプロモーター活性が促進あるいは抑制されたかを、定量的かつ感度良く捉え、2次元上のグラフ(プロモーター活性プロファイル)として示すことができました。また、標的分子が異なる2種類の抗がん剤によるプロモーター活性変動の相違は、2つのプロファイルの比較で明瞭に観察できました。さらに、2種類の抗がん剤のプロモーター活性プロファイルを組み合わることで、同じ情報伝達経路内の別の分子に作用するもう1種類の抗がん剤のプロモーター活性プロファイルを表せることも分かりました。これは、既知の薬剤のプロモーター活性プロファイルから、未知の薬剤の作用を推定することが可能であることを示します。
これらの結果は、非増幅deepCAGE法を用いた遺伝子発現の定量解析が、既存の薬剤の標的タンパク質や作用機序の解明などに広く応用可能であり、新薬開発での薬理学分野への貢献が期待できることを示しています。本成果は、科学雑誌『CPT Pharmacometrics and Systems Pharmacology』(9月25日付け:日本時間9月26日)に掲載されました。
背景
新薬の開発では、候補薬物の作用を細胞レベルでモニターし、それらの薬効や安全性を評価することが、基本的な作業となっています。特に、薬剤投与の前後で細胞に発現している遺伝子をゲノム全域にわたって捉え、薬剤の作用を遺伝子発現量の変化として定量的に捉える手法が注目されています。これまでは、マイクロアレイ法を用いた解析が盛んに行われてきました。マイクロアレイ法は、測定試料(細胞から抽出したRNAから合成したDNA)と、予めデザインされたDNAプローブとのハイブリダイゼーション[4]により遺伝子発現の有無やその発現量などの情報を検出しますが、測定するRNAの種類、測定感度、測定値の定量性の範囲などにおいて制約がありました。そのため、ゲノム全域において遺伝子発現変化の十分な定量的解析ができませんでした。
CAGE(Cap Analysis of Gene Expression)法は、理研が開発した独自の技法です。この特徴は、mRNAの末端の塩基配列(CAGEタグ)を調べて1塩基の精度でゲノム上に存在する転写開始点[5]を網羅的に同定することにより、その直上流に存在するプロモーターを同定し、かつ、その位置からスタートする転写物の発現量(プロモーター活性)も測定できることです。CAGE法は、理研が主催する国際科学コンソーシアムFANTOM[6]でのゲノム全域な解析に用いられました。さらにFANTOMではCAGEの技術を次世代シーケンサーと組み合わせることでdeepCAGE法を開発し、血球系細胞の分化過程におけるプロモーターレベルでの転写制御ネットワーク解析に成功しました注1)。また最近では、ヒトゲノムの全ての機能要素の解析を目指したENCODEプロジェクト[7]において、プロモーター活性を示す指標としてCAGEデータが使われました注2)。
これまで共同研究グループはCAGE法を用いて、ヒトやマウスの正常組織、培養細胞のプロモーター活性解析を行なってきましたが、薬剤作用に関する詳細なプロモーター活性解析は未着手でした。その理由として、①100万個以上のCAGEタグが必要なこと、②シーケンス用サンプルを調製する際にPCRを用いるためわずかなプロモーター活性の変化を厳密に捉えるのが技術的に困難なこと、などが挙げられます。しかし、①については、次世代シーケンサーの登場により、deepCAGE法を用いて数100万のCAGEタグを短時間、かつ低コストで得られるようになりました。また、②についても、PCRを用いない非増幅deepCAGEライブラリー調製法の開発に成功し、上記の制約はなくなりました。今回、共同研究グループは、薬剤作用がもたらす微弱な遺伝子発現の変化を網羅的・定量的に捉えるため、詳細なプロモーター活性解析に挑みました。
- 注1) 2009年4月20日プレスリリース
- 注2) 2012年9月6日プレスリリース
研究手法と成果
本研究では、ヒト乳がん由来の細胞株であるMCF-7細胞をモデルとし、3種類の抗がん剤(U0126、ワルトマニン、ゲフィチニブ)をそれぞれ投与した時のがん細胞のプロモーター活性の変化を調べました。3つの薬剤は、がん細胞の増殖に関わる情報伝達経路の阻害剤です。U0126とワルトマニンは、それぞれRas-ERKおよびPI3K-Aktと呼ばれる情報伝達経路を阻害します。一方ゲフィチニブは、EGFRと呼ばれるレセプターキナーゼを標的として阻害しますが、その結果EGFR下流の主要な情報伝達経路であるRas-ERKおよびPI3K-Akt情報伝達経路も阻害します(図1)。各薬剤は、阻害効果が現れる濃度レベルで、できる限り低い濃度を用いました。
非増幅deepCAGE法によるゲノム全域のプロモーター活性解析の結果、共同研究グループはMCF-7細胞から1万個あまりのプロモーターを同定することができました。これらのプロモーター活性を3回の独立した実験で測定しましたが、その結果実験間での再現性が極めて高いことが分かりました。そこで、薬剤を投与しなかった細胞と抗がん剤を投与した細胞を比較し、統計的に有意なプロモーター活性の変化が見られた遺伝子を同定しました。具体的には、U0126では139種、ワルトマニンでは168種、ゲフィチニブは157種のプロモーター活性に変化が見られました(図2左)。この中には、すでに論文報告されているErbB3などのがん遺伝子も含まれており、今回の実験結果が正しいことが示されました。
次に、異なる情報伝達経路に標的分子を持つU0126とワルトマニンの薬剤作用の違いを、本技法で検出できるか検討しました。それぞれを投与した細胞のプロモーター活性の変動を比較解析したところ、阻害する情報伝達経路に応じたプロモーター活性変動の相違が明瞭に観察されました。一方、同じ試料をマイクロアレイ法で解析した結果では、この相違をうまく捉えることができませんでした(図2右)。
さらに、ゲフィチニブ投与によるEGFRの阻害効果が、EGFRの下流に存在する2つの主要な情報伝達経路(Ras-ERK、PI3K-Akt)の阻害剤であるU0126とワルトマニンそれぞれの阻害効果で説明できるかを検討しました。その結果、ゲフィチニブ投与時の阻害プロファイルは、U0126とワルトマニンそれぞれの阻害プロファイルによって表せることが分かりました(図3)。
今後の期待
次世代シーケンサーを利用した非増幅deepCAGE法により、薬剤作用をプロモーター活性プロファイルとして、初めて定量的かつ感度良く捉えることができました。このことは、新薬開発だけでなく、これまで未知であった既存薬剤の標的タンパク質や作用機序を解析するために、広く利用できることから、新薬開発への応用が期待できます。また、臨床現場における薬効配合剤や複数薬剤投与の評価への応用が可能であり、今後の薬理学に貢献するライフサイエンス技術として期待できます。
原論文情報
- Kazuhiro Kajiyama, Mariko Okada-Hatakeyama, Yoshihide Hayashizaki, Hideya Kawaji, Harukazu Suzuki. "Capturing drug responses by quantitative promoter activity profiling." CPT Pharmacometrics and Systems Pharmacology, 2013, doi: 10.1038/psp.2013.53
発表者
理化学研究所
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 オミックス応用技術研究グループ
グループディレクター 鈴木 治和 (すずき はるかず)
独立行政法人理化学研究所
社会知創成事業
予防医療・診断技術開発プログラム
コーディネーター 川路 英哉 (かわじ ひでや)
お問い合わせ先
独立行政法人理化学研究所 ライフサイエンス技術基盤研究センター
チーフ・サイエンスコミュニケーター 山岸 敦 (やまぎし あつし)
Tel: 078-304-7138 / Fax: 078-304-7112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.プロモーター活性
プロモーターは、遺伝子の転写開始に必要なゲノム上の特定領域やその塩基配列をさす。どの遺伝子からどれくらいの量の転写が起きるかは、転写因子と呼ばれるタンパク質とプロモーターの結合によって制御されている。遺伝子の転写開始点はプロモーターのすぐ下流にあるため、細胞から抽出したmRNAの5’末端の塩基配列を定量的に解析することで、特定のプロモーターからの転写産物の量を知ることができる。これをプロモーター活性と呼ぶ。同じ遺伝子でも、プロモーター活性は細胞の種類や状態によって異なる。 - 2.マイクロアレイ法
既知の遺伝子の1本鎖DNA断片を高密度にスライドガラスなどに並べ、貼付けたものをマイクロアレイと呼ぶ。これに、組織や細胞から抽出したmRNAを逆転写反応で合成したDNAをハイブリダイゼーションさせることで、特定の遺伝子の発現量を検出する方法。 - 3.非増幅deepCAGE法
理研が開発した独自の技法であるCAGE法(Cap Analysis of Gene Expression)を発展させた新しいゲノム解析技術。特定の細胞で発現しているほぼ全ての遺伝子のプロモーター活性を網羅的・定量的に検出することができる。 - 4.ハイブリダイゼーション
DNAやRNAを構成する5種類の塩基は、Aに対してT(RNAの場合はU)が、Gに対してCが水素結合する性質を持つ。ある1本鎖のDNAやRNAに対して、これら対応する塩基とその並びを入れ替えた配列を持つ1本鎖を相補鎖と呼ぶ。相補鎖どうしは溶液中で2本鎖を形成する性質をもち、この反応をハイブリダイゼーションと呼ぶ。 - 5.転写開始点
ゲノムDNA上で、mRNAの転写が始まる部位。転写とは、DNAの塩基配列を相補的にRNAに写し取る反応のこと。遺伝子情報によるタンパク質合成過程の第一段階である。転写開始点に相当するmRNAの末端は、キャップ(Cap)と呼ばれる構造をとる。 - 6.FANTOM
2000年に、理研ゲノム科学総合研究センター 遺伝子構造機能研究グループ(旧・オミックス基盤研究領域、現・理研ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門)が中心となって結成した。哺乳動物(マウス)の遺伝子を網羅的に機能注釈することを主眼とする国際的研究コンソーシアム(Functional ANnoTation Of Mammalian cDNA)の略称。現在は活動範囲を拡大し、遺伝子ネットワークの解明に取り組んでいる。オーストラリア、シンガポール、スウェーデン、南アフリカ、イタリア、ドイツ、ギリシャ、スイス、英国、米国などを含む全世界の15カ国から、51の研究機関などが参加している。 - 7.ENCODEプロジェクト
ポストゲノム戦略として米国で立ち上げられたプロジェクトで、米国NIH(衛生研究所)の国立ヒトゲノム解析研究所 (National Human Genome Research Institute)を中心として、2003年9月から正式に開始された計画。‘ENCODE’とは、Encyclopedia of Human DNA Elements(ヒトDNAの百科事典)から命名されており、完全解読されたヒトゲノム上に、遺伝子の機能を担う領域をすべて書き込んで、全ヒトゲノム(DNA)の百科事典を作成することを目指している。
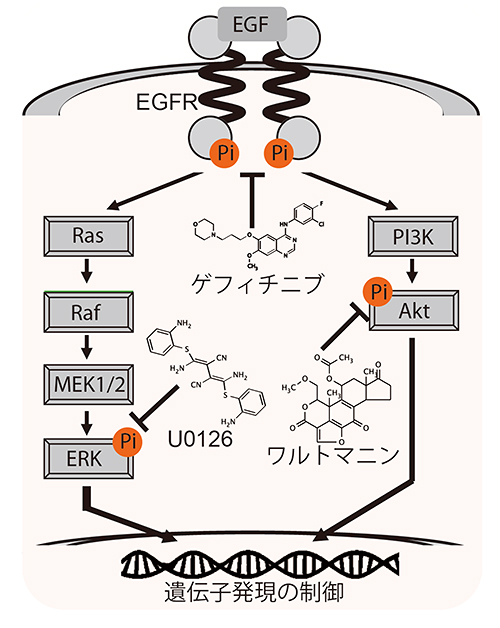
図1 本研究で用いた抗がん剤の標的となる情報伝達経路
増殖因子であるEGFが細胞膜上の受容体(EGFR)に結合すると、EGFRの細胞内にあるキナーゼ(リン酸化酵素)部分が活性化する。EGFRの下流では、ERKとAktの2つのリン酸化酵素が別々の経路を介して活性化される。ゲフィチニブ(Gefitinib)、U0126、ワルトマニン(Wortmannin)のそれぞれの薬剤が阻害する標的分子をT字バーで示す。
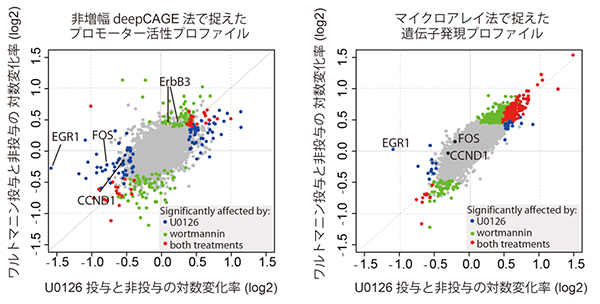
図2 非増幅deepCAGE法と従来のマイクロアレイ法で得た遺伝子発現プロファイルの比較
ヒト乳がん由来の細胞株であるMCF-7細胞に、U0126(横軸)、ワルトマニン(縦軸)のいずれかを投与した時の遺伝子発現量の変化を対数グラフで示した。グラフの点は個々の遺伝子を表し、特にU0126投与で影響を受けた遺伝子を青、ワルトマニン投与で影響を受けた遺伝子を緑、どちらの薬剤でも影響を受けた遺伝子を赤で示した。対角線に近いものほど両薬剤投与で類似した変化が測定されたものとなる。従来のマイクロアレイ法では対角線付近に遺伝変化の測定値が集中するのに対し(右)、非増幅deepCAGE法では、2種類の薬剤が個々の遺伝子の発現に与える影響の違いを捉えた結果となった(左)。
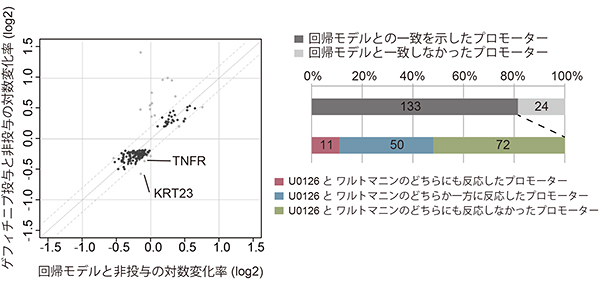
図3 ゲフィチニブで変化した157遺伝子と、U0126およびワルトマニンの阻害効果の関係
- 左: ゲフィチニブを投与した時に顕著な反応を示した157遺伝子の発現変化を縦軸で示し、横軸は、それらの遺伝子の変化量をU0126とワルトマニンそれぞれを投与した時のデータから統計学的手法(回帰モデル)で求めた値を示す。実験値と回帰モデルがよく一致した133遺伝子を黒丸で示し、一致しなかった24遺伝子を灰色で示した。TNFRとKRTは、U0126とワルトマニンの標的分子とは独立した情報伝達経路でゲフィチニブによる阻害を受けることが知られている遺伝子の例。
- 右: 実験値と推定値がよく一致した133遺伝子について、U0126とワルトマニンのどちらからどのような影響を受けているかを示したグラフ。
