ポイント
- アラニンの遺伝暗号を解読する酵素・tRNA複合体の構造解析に成功
- 二重らせんにわずかな変形を持つRNAだけを選択する巧妙な仕組みを解明
- 非反応性複合体を介した基質選択という新しい原理を発見し、応用を可能に
要旨
理化学研究所(理研、野依良治理事長)は、遺伝暗号解読の主要なプロセスで全く新しい分子メカニズムが働いていることを発見しました。これは、理研横山構造生物学研究室の横山茂之上席研究員、永沼政広特別研究員、理研ライフサイエンス技術基盤研究センター 構造・合成生物学部門の関根俊一チームリーダーと、米国のスクリプス研究所、トーマスジェファーソン大学との共同研究グループによる成果です。
主に20種類のアミノ酸が連なったタンパク質は、私たちの体の重要な構成要素です。タンパク質の合成過程では、遺伝子の塩基配列が、トランスファーRNA(tRNA)[1]の仲介[2]により、遺伝暗号の規則に従い特定のアミノ酸へと翻訳されます。遺伝暗号の規則を実現しているのは、個々のアミノ酸に対応する20種の「アミノアシルtRNA合成酵素(aaRS)[3]」です。aaRSは対応するアミノ酸と、そのアミノ酸の運搬を担うtRNAとを厳しく選択して結合し、反応させます。1988年に、アミノ酸の1つのアラニンで、tRNA選択の決め手(決定因子)がtRNAの二重らせん部分にある変則的塩基対(G・U塩基対)[4]であることが発見されました。しかし、アラニンに対応するaaRS「アラニルtRNA合成酵素(AlaRS)」によるtRNA選択のメカニズムには多くの謎があり、その実態は解明されていませんでした。
共同研究グループは、大型放射光施設「SPring-8」[5]と放射光科学研究施設「フォトンファクトリー」[6]を用いて、AlaRSとそのtRNAとの複合体のX線結晶構造を解析し、tRNA選択の詳細なメカニズムの解明に成功しました。アラニンと反応するtRNAの末端部分が、天然型ではAlaRSの活性部位に向かって入って行くのに対し、天然型のG・U塩基対をA・U塩基対に入れ替えた変異型tRNAでは、AlaRS上の「分岐点」で向きを変え、活性部位から遠く離れて行き「非反応性複合体[7]」となることが分かりました。変異型tRNAは、天然型tRNAとほぼ同じ強さでAlaRSと結合するのにもかかわらず、反応速度が2桁も遅く、事実上、選択されません。この反応速度の差だけに基づくtRNA選択の原理は、これまで説明できませんでした。今回、結晶構造の解析に成功したことにより、天然型tRNAでは、G・U塩基対に起因するわずかな変形が、末端部分の位置をずらして分岐点を超えさせ、反応を2桁も速くすることが分かりました。非反応性複合体を介した基質選択は新規の基本的概念です。今後、これを応用することで、人工アミノ酸を遺伝暗号に組み込むなど、新技術の開発につながると期待できます。
本研究成果は、文部科学省ターゲットタンパク研究プログラム、文部科学省創薬等支援技術基盤プラットフォーム事業の一環として行われたもので、英国の科学雑誌『Nature』オンライン版(6月11日付け:日本時間6月12日)に掲載されました。
背景
私たちの体を構成するタンパク質は生命を営むうえで重要な分子で、主に20種類のアミノ酸できています。全てのタンパク質は、それぞれの遺伝子の塩基(A・G・T・C)の配列として暗号化されて記録された情報(遺伝暗号)に従って、アミノ酸が数珠つなぎになって合成されます。
DNAの遺伝子領域から転写されたメッセンジャーRNA(mRNA)[8]の塩基の(A・G・U・C)配列は、3つの塩基ずつに区切られ、その3塩基の並び方が1つのアミノ酸の種類を指定する暗号になっており、暗号の最小単位を意味する「コドン」と呼ばれています。mRNAのコドンは、タンパク質合成の場である細胞小器官「リボソーム」で、順番に、トランスファーRNA(tRNA)の「アンチコドン」と呼ばれる部分の3塩基の配列によって認識されます。同時に、tRNAが指定のアミノ酸を運んできていて、タンパク質が合成されます。このように、tRNAがコドンとアミノ酸との間を仲介することによって、遺伝子の塩基配列は、タンパク質のアミノ酸配列に「翻訳」されています。
コドンとアンチコドンの間の認識は、DNAの複製や転写における情報の受け渡しと似た、RNA分子間のワトソン・クリック型塩基対[9]などによって行われます。それに対して、アミノ酸はRNAとは全く異なるタイプの分子であり、それらをどのようにして結びつけるかが、遺伝暗号における情報変換の本質といえます。
アミノ酸とtRNAとを反応させ、結びつける役割を担うのが、「アミノアシルtRNA合成酵素(aaRS)」です。それぞれのアミノ酸に対して、専用のaaRSが存在し、数多くのアミノ酸とtRNAの中から、自分に対応するものだけを厳密に選択して反応させます。つまり、コドンとアミノ酸との対応関係を決める役割を担っているのがaaRSといえます。aaRSによるアミノ酸の認識については、これまで多くのX線結晶構造解析が行われ、対応するアミノ酸をどのようにして選び出すか、その認識機構が解明されてきました。四半世紀ほど前に、aaRSによるtRNAの認識の決め手となる目印(決定因子[10])が、tRNA中の少数のヌクレオチド(DNAやRNAを構成する単位)であることが発見されました。また、多くのaaRSは、tRNAのアンチコドンを決定因子として認識すると考えられていました。
aaRSの1つである「アラニルtRNA合成酵素(AlaRS)」は、アミノ酸の1つアラニンとアラニン専用のtRNA(tRNAAla)を選択して反応させ、アラニルtRNAAlaを生成します。1988年、AlaRSはtRNAAlaのアンチコドンではなく、アクセプターステム[11]と呼ばれるtRNAの二重らせん構造の3番目にある変則的塩基対(G・U塩基対:G3・U70)を決定因子として認識することで、tRNAAlaとその他のtRNAを区別し、選択することが発見されました(図1左、右)。このG3・U70塩基対をA3・U70やG3・C70などのワトソン・クリック型塩基対に置換すると、AlaRSによってアラニルtRNAAlaが生成されなくなります。これと反対に、他のtRNAにG3・U70塩基対を導入するとアラニルtRNAが生成されます。当時、tRNAの決定因子はアンチコドンであると思われていたため、tRNAAlaの決定因子がアクセプターステムに存在するというのは大発見でした。また、DNAと異なり、RNAの二重らせん中の塩基対は、その主溝側からタンパク質が近づくことは困難で、塩基対の種類を認識することはできないと考えられています。
AlaRSに対するG3・U70塩基対の重要性の発見から20年以上もの間、AlaRSがどのようにして活性部位から離れたただ1つの塩基対に強く依存してtRNAAlaを区別しているのかは、分からないままでした。さらに、G3・U70塩基対をA3・U70などのワトソン・クリック型塩基対に置換すると、AlaRS-tRNAAla間の結合の強さ(親和性)に影響を与えることなく、反応速度が2桁も遅くなることが分かっています。このような親和性ではなく反応速度を変えることによる基質選択メカニズムは、酵素による選択的RNA認識メカニズムという観点からも興味深い謎でした。
研究手法と成果
共同研究グループは、AlaRSがG3・U70塩基対を決定因子としてtRNAAlaを選択するメカニズムを明らかにするために、①AlaRSと天然型のtRNAAla(tRNAAla/GU)が結合した状態の結晶構造と、②AlaRSとG3・U70をA3・U70に置換した変異型のtRNAAla(tRNAAla/AU)が結合した状態の結晶構造の解明に取り組みました。X線結晶構造解析は、理研の大型放射光施設「SPring-8」、高エネルギー加速器研究機構の放射光科学研究施設「フォトンファクトリー」で行いました。
解析の結果、①・②ともAlaRSは二量体を形成し、tRNAAla1分子が結合していることが分かりました(図2左)。tRNAAlaのアクセプター/Tステム[1]はAlaRSに包み込こまれるように相互作用しており(図2右)、tRNAのアンチコドンを認識するaaRSとは全く異なる結合様式でした。AlaRSは、アクセプターステムの二重らせんを主溝と副溝の両側から挟み、深い主溝を押し広げて、決定因子の位置を挟み付けるように認識します。これにより、G3・U70塩基対もA3・U70塩基対も、U70の位置が同じようになるよう、精密に位置決めされていました(図3)。これらの結合様式は、天然型のtRNAAla/GUと変異型のtRNAAla/AUとでほぼ同じであり、それらとAlaRSの親和性がほぼ同じでした。
G・U塩基対とA・U塩基対などのワトソン・クリック型塩基対は、異なる幾何学的構造をしているため、Uの位置を合わせると、GとAの位置は大きくずれて二重らせん構造にわずかな変形が生じます(図1下)。今回の構造解析によって、tRNAの末端アデノシン(CCA、アミノアシル化されるヌクレオチド)のAlaRS上での位置が、天然型と変異型のtRNAAlaで全く異なり、天然型ではAlaRSの活性部位に到達できるのに対し、変異型では、活性部位から約20Å(オングストローム、1Åは100億分の1メートル)も離れており活性部位に到達できないという、予想外の大きな違いが発見されました。
このG3・U70塩基対とA3・U70塩基対の構造のわずかな変形の影響は、tRNAの末端方向に伝わっていきますが、アクセプターステムの二重らせんが終わるまでは小さな違いに留まっています。詳細な構造を見てみると、アクセプターステムの二重らせんの終わりからtRNAの末端CCA鎖までは二重らせんを作らずフレキシブルな1本鎖となっており、天然型と変異型の経路は全く異なる方向へ配置されていることが分かりました。アクセプターステムの二重らせんの終わり付近にAlaRSの突起状の構造(セパレーター構造)があり、これが「分岐点」となって、tRNAの末端CCA鎖を2つの経路に振り分けていました(図4左)。この経路の配置から、G・U塩基対に特有のらせん構造の変形の影響は、アクセプターステムの二重らせんの主溝を広げて決定因子の位置を挟み付けるように認識することで末端方向に伝えられ、分岐点で大きく増幅されるという巧妙な仕組みによって、G3・U70塩基対を持つtRNAAlaの末端CCA鎖は活性部位に到達することができることが分かりました。一方、A3・U70などワトソン・クリック型の塩基対を持つtRNAの末端CCA鎖は、この仕組みが働かないため、活性部位とは全く異なる場所へ配置され、到達できないことが分かりました(図4右)。
変異型のtRNAAla/AUのAlaRSとの結合の様式では、反応すべき末端アデノシンが遠く離れた位置に隔離されていることから、「非反応性複合体」と呼ばれ、天然型のtRNAAla/GUの「反応性複合体」とは全く異なります。AlaRSのアミノアシル化の反応スキームに非反応性複合体の形成を含めてシミュレーションを行ったところ、変異型のtRNAAla/AUの場合に、天然型のtRNAAla/GUと比較して、親和性がほとんど違わないのに、反応速度が2桁も低下するという観測データを正確に再現できました。このような非反応性複合体を介した基質識別機構は、これまで報告されたことのない新しい概念です。
今後の期待
新たなtRNA選択メカニズムの発見によって、長い間謎のままだった、生命にとって必要不可欠な酵素反応の仕組みの説明が可能になりました。多くのaaRSはG3・U70よりも活性部位からさらに遠いアンチコドン配列を認識することでtRNAを選択しており、アンチコドンの変異が親和性に影響を与えることなく反応速度を変える例が多く知られています。そのようなaaRSの系でも、「非反応性複合体を介する基質識別機構」が働いているのか、全く異なるメカニズムがあるのかは謎のままです。今後、この非反応性複合体を介する基質識別機構を取り入れた酵素の設計などにより、人工塩基をもつtRNAの認識・選択が可能になれば、それらの人工コドンを人工アミノ酸に割り当てる遺伝暗号拡張技術の開発につながると予想され、有用な人工アミノ酸を活用するタンパク質工学の発展につながると期待できます。
原論文情報
Masahiro Naganuma, Shun-ichi Sekine, Yeeting Esther Chong, Min Guo, Xiang-Lei Yang, Howard Gamper, Ya-Ming Hou, Paul Schimmel, and Shigeyuki Yokoyama.
"The selective tRNA aminoacylation mechanism based on a single G・U pair".
Nature, 2014,doi:10.1038/nature13440
発表者
理化学研究所
上席研究員研究室 横山構造生物学研究室
上席研究員 横山 茂之(よこやま しげゆき)
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.トランスファーRNA(tRNA)、アクセプター/Tステム
tRNAは、transfer RNA(転移リボ核酸)の略号。4種類の塩基、アデニン(A)、ウラシル(U)、グアニン(G)、チミン(C)のヌクレオチドが70~100個連なってL字型の三次元構造をとる。標準的なL字構造は、アクセプターステム、Tステム、Dステム、アンチコドンステムの4つのステム(2重らせん構造)と、それらを結ぶループ(1本鎖構造)で構成される。20種のアミノ酸それぞれに専用のtRNAがあり、アミノ酸の種類ごとにアンチコドン配列(3連続の塩基)が割り当てられている。専用のaaRSによってCCA末端のAにアミノ酸が結合される。 - 2.仲介
mRNAのコドンとアミノ酸は、直接に相互作用することができないため、コドンをtRNAが認識し、アミノ酸をaaRSが認識し、tRNAとaaRSが互いを認識することによって、間接的に、コドンとアミノ酸が対応付けられる。このように、遺伝暗号の規則に従ったコドンとアミノ酸の間の対応付けは、tRNAとaaRSが仲介する。 - 3.アミノアシルtRNA合成酵素(aaRS)
タンパク質を構成するアミノ酸は主に20種類存在する。20種類のアミノ酸のそれぞれに対してアミノアシルtRNA合成酵素が20種類存在し(アスパラギン酸に対応するAspRS、リシンに対応するLysRSなど)、アデノシン三リン酸(ATP)のエネルギーを利用してアミノ酸を活性化したのち、対応するtRNAのCCA末端に付加する(アミノアシル化)。 - 4.変則的塩基対(G・U塩基対)
A・U、G・Cなどの天然型塩基対と比べて、変則的塩基対(G・U塩基対)では、Gが副溝側へ、Uが主溝側へとずれており、幾何学的に異なる。 - 5.大型放射光施設「SPring-8」
兵庫県佐用郡の播磨科学公園都市内にある大型放射光施設。公益財団法人高輝度光科学研究センター(JASRI)によって、運転・維持管理、及び、利用促進業務が行われている。
大型放射光施設「SPring-8」のホームページ - 6.放射光科学研究施設「フォトンファクトリー」
茨城県つくば市の筑波研究学園都市にある大型放射光施設。大学共同利用機関法人高エネルギー加速器研究機構の実験施設である。
放射光科学研究施設「フォトンファクトリー」のホームページ - 7.非反応性複合体
A3・U70をもつ変異型tRNAAlaは、G3・U70をもつ天然型tRNAAlaと異なり、そのCCA末端が活性部位とは異なる領域に捕捉されてしまうため、アミノアシル化されない。 - 8.メッセンジャーRNA(mRNA)
タンパク質を翻訳できる塩基配列情報と構造を持ったRNA。mRNAはDNAから写し取られた遺伝情報に従い、タンパク質を合成(翻訳)する。 - 9.ワトソン・クリック型塩基対
RNAはA、U、G、Cの塩基で構成されるが、AとU、GとCがペアとなる塩基対をワトソン・クリック型塩基対と呼ぶ。 - 10.決定因子
tRNAの認識の決め手となるヌクレオチド。20種のアミノ酸に対応するtRNAのそれぞれは、遺伝暗号の規則上で対応すべきaaRSによって選択されるための目印となるヌクレオチドを持つ。この目印となるヌクレオチド(決定因子)によって、どのaaRSによってアミノアシル化されるかが決まる。 - 11.アクセプターステム
tRNAは4つの二重らせん構造を持ち、そのうち、アミノ酸と反応する(アミノアシル化される)CCA末端に最も近い二重らせん構造をアクセプターステムと呼ぶ。
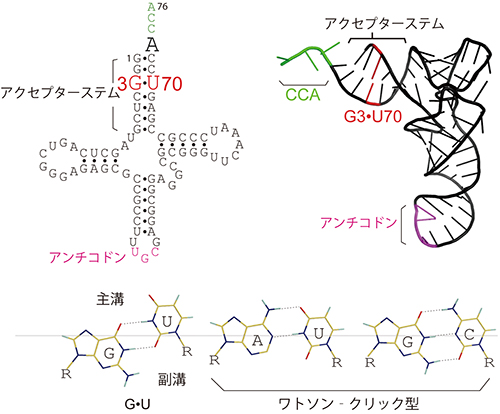
図1 tRNAAlaの配列と立体構造とG・Uとワトソン・クリック型塩基対
- 左: tRNAAlaの配列
- 右: tRNAAlaのL字型立体構造
- 下: G・U塩基対とワトソン・クリック型塩基対
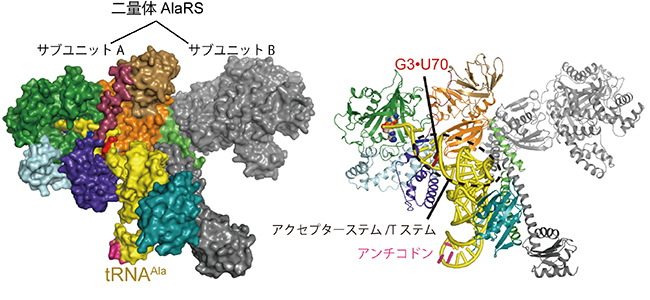
図2 AlaRSの全体構造
- 左:
AlaRS-tRNAAla複合体の表面モデル
サブユニットAとBが二量体を作り、tRNAAlaがサブユニットAにだけ結合している。 - 右:
AlaRS-tRNAAlaのリボンモデル
アクセプターステム/TステムがAlaRSによって取り囲まれるように相互作用している。
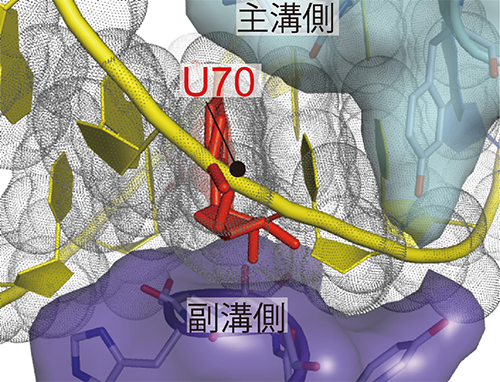
図3 AlaRSにより主溝と副溝の両側から挟まれるアクセプターステム
AlaRSは、アクセプターステム(黄色、原子の大きさをドットで示す)を、水色と紫色の構造を用いて、主溝と副溝の両側から挟み、深い主溝を押し広げて、決定因子(赤)の位置を挟み付けるように認識することで、G3・U70塩基対とA3・U70塩基対のU70の位置が同じようになるよう、精密に位置決めされる。
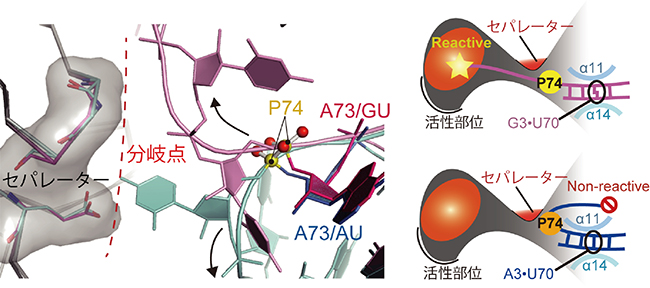
図4 選択的アミノアシル化のためのtRNAの末端CCA鎖分岐メカニズム
- 左:
分岐しているtRNAの末端CCA鎖の構造
AlaRSのセパレーター構造が「分岐点」となり、末端CCA鎖の経路が2つに分岐している。 - 右:
CCA末端鎖分岐メカニズムの概略図
G3・U70を持つtRNAAlaのCCA末端鎖は活性部位に向かうことができ、A3・U70を持つtRNAAlaのCCA末端は活性部位には到達できない。
