2015年5月5日
理化学研究所
カリフォルニア大学デイビス校
生体内の低分子化合物を網羅的に捉える解析プログラムを開発
-MS-DIALによる次世代メタボロミクス-
要旨
理化学研究所(理研)環境資源科学研究センター メタボローム情報研究チームの有田正規チームリーダー(情報・システム研究機構国立遺伝学研究所 教授)、津川裕司特別研究員と、カリフォルニア大学デイビス校 オリバー・フィーン教授らの共同研究チーム※は、生体内の低分子化合物[1]を網羅的に捉えて解析するメタボロミクス[2]用の統合解析プログラムMS-DIAL (Mass Spectrometry Data Independent AnaLysis)[3]を開発しました。
メタボロミクスとは、生体内の低分子化合物を測定し、食品の品質評価や疾患診断を行う技術です。測定には、主に液体クロマトグラフィータンデム質量分析装置(LC-MS/MS)[4]を用います。具体的には、クロマトグラフィー[5]で分離した化合物の構造を、質量分析装置を用いて同定します。しかし、クロマトグラフィーで数千のピーク[6]が見られる場合でも、ピークのほとんどは複数の化合物が重なりあったものであることから、個々の化合物構造を同定できる精度を持ったMS/MSスペクトル[7]は数百程度しか得られません。したがって、一度の解析では100~200程度の化合物構造しか同定できませんでした。
共同研究グループは、複数の化合物が重なりあったMS/MSスペクトルを個々のMS/MSスペクトルに分離するアルゴリズムを搭載した統合解析プログラムMS-DIALを開発しました。これによって、9種類の藻類のMS/MSスペクトルから1,023種の化合物を一斉に同定することに成功しました。MS-DIALは、化合物から検出した全てのMS/MSスペクトルから、個々の化合物のMS/MSスペクトルを選択的に取得するプログラムです。条件毎に都度測定を行う必要がないため、試料の過度な損失を回避することが可能です。本成果は、測定しうる化合物全てを捉えるという網羅性を備えた「次世代メタボロミクス」のための基盤技術となる成果です。
本研究は、JST国際科学技術共同研究推進事業(戦略的国際共同研究プログラム)の一環として行われ、英国の科学雑誌『Nature Methods』に掲載されるのに先立ち、オンライン版(5月4日付け:日本時間5月5日)に掲載されます。
※共同研究チーム
理化学研究所
環境資源科学研究センター メタボローム情報研究チーム
チームリーダー 有田 正規(ありた まさのり)(情報・システム研究機構国立遺伝学研究所 教授)
特別研究員 津川 裕司(つがわ ひろし)
統合生命医科学研究センター メタボローム研究チーム
上級研究員 池田 和貴(いけだ かずたか)
カリフォルニア大学デイビス校 ゲノムセンター
教授 Oliver Fiehn(オリバー・フィーン)
Associate Specialist Tomas Cajka(トーマス・チャイカ)
Assistant Project Scientist Tobias Kind(トビアス・キント)
学生(博士後期課程)Yan Ma(ヤン・マ)
カリフォルニア大学デイビス校
教授 Jean VanderGheynst(ジーン・ヴァンダゲーンス)
特別研究員 Brendan Higgins(ブレンダン・ヒギンス)
ライフィクス株式会社
代表取締役 金澤 光洋(かなざわ みつひろ)
背景
メタボロミクスとは、生体内の低分子化合物の微細な変動を網羅的かつ高解像度で捉える技術であり、熟練した試験者によってのみ可能であった食品の品質管理や難病疾患の早期発見を、誰でも簡単にできるようにする技術として注目されています。メタボロミクスの測定では、液体クロマトグラフィータンデム質量分析装置(LC-MS/MS)がよく使用されています。具体的には、クロマトグラフィーで分離した化合物の構造を、質量分析装置を用いて同定します。LC-MS/MSで化合物を同定するために必要な情報は、化合物の保持時間(試料の導入を開始してから化合物が溶出するまでに必要な時間)、精密質量、同位体比と、MS/MSスペクトルの4つです。中でもMS/MSスペクトルは化合物構造を同定する上で最も重要です。しかし、従来のLC-MS/MSでは走査スピードに問題があり、すべての化合物からMS/MSスペクトルを取得することは極めて困難でした。加えて、LC-MS/MSのデータベースに登録された標準品(基準となる化合物データ)のMS/MSスペクトル数が限られていたため、取得したスペクトルのデータとデータベースのデータが照合できないケースが大半を占めていました。そのため、一度の分析で100~200程度の化合物しか同定できませんでした。共同研究グループは、検出される全てのMS/MSスペクトルを取得する技術であるData Independent MS/MS Acquisition法(DIA法)[8]に注目しました。DIA法は、「理論的には」検出される全MS/MSスペクトルが取得可能です(図1)。しかし、得られるMS/MSスペクトルは複数の成分に由来する混合スペクトルであるという問題点があり、その混合スペクトルを個々のスペクトルに分離する方法はありませんでした。
そこで本研究では、複数の化合物が重なりあったMS/MSスペクトルを個々のMS/MSスペクトルに分離する解析プログラムの開発に取り組みました。同時に、LC-MS/MSのデータベースを増強するため、生体を構成する主要な化合物構造から予測される保持時間と理論MS/MSスペクトル[9]を網羅的に構築した新たなデータベースの開発にも取り組みました。
研究手法と成果
共同研究グループは、複数の化合物が重なりあったMS/MSスペクトルから個々のMS/MSスペクトルを取り出すための波形分離アルゴリズムを開発し(図2)、データの読み込みから統計解析まで迅速に実行可能な統合解析プログラム「MS-DIAL(Mass Spectrometry Data Independent AnaLysis)」を開発しました(図3)。MS-DIALにより、化合物から検出した全MS/MSスペクトルから個々ののMS/MSスペクトルを選択的に取得することが可能となりました。これにより、一度の解析で網羅的に低分子化合物を同定することができます。
共同研究グループは、生体を構成する主要な化合物である77,962の脂質分子種に対して、コンピュータプログラムを用いて予測保持時間と理論MS/MSスペクトルのデータベースを構築しました。構築したデータベースをMS-DIALに搭載したところ、1検体15分の分析で、9種の藻類から得た検体から1,023個の脂質分子種を同定することに成功しました。さらに、9種類の藻類について、同定した1,023個の脂質分子種それぞれの有無0、1の値を持った脂質フィンガープリント[10])を調べ分類した結果、古典分類学の系統樹と完全に一致する結果を得ました(図4)。分類学への応用は、形態観察や16S rRNA系統解析による系統樹[11]に存在する曖昧さを補う、もしくは全く新たな系統樹理論となりうるものです。例えば、昨今、藻類株UTEX2341はクロレラ種に属するかナンノクロロプシス種に属するか議論が分かれていましたが、脂質フィンガープリントによる系統樹はクロレラ種に属することを示唆しました(図4)。
さらに、共同研究グループは、MS-DIALに22,753の標準品MS/MSスペクトルおよび135,456脂質分子種の理論 MS/MSスペクトルを搭載し、網羅的な化合物同定基盤が整備された統合解析プログラムを開発しました。また、標準品の保持時間、精密質量、同位体比、およびMS/MSスペクトルを組み合わせた統合指標による独自の同定アルゴリズムにより信頼性の高い化合物同定が可能となりました。
今後の期待
MS-DIALの開発により、化合物から検出される全イオンのMS/MSスペクトルを用いた解析が可能になったことで、時間・場所を問わず、かつ誰が取得したデータであっても同じ同定結果が得られるようになりました。これにより、従来は必要なデータに合わせて個別に解析を行っていたため一過的な結果でしか議論ができなかったメタボロミクスについて、連続性を持った議論が可能となります。今後は、MS-DIALに搭載するデータベースの拡充を進める予定です。また、既存データベースであるMassBank[12]の拡充・整備を通じて、より多様なスペクトルデータに対応可能なプログラムの開発を進めます。
原論文情報
- Hiroshi Tsugawa, Tomas Cajka, Tobias Kind, Yan Ma, Brendan Higgins, Kazutaka Ikeda, Mitsuhiro Kanazawa, Jean VanderGheynst, Oliver Fiehn, and Masanori Arita, "MS-DIAL: Data Independent MS/MS Deconvolution for Comprehensive Metabolome Analysis", Nature Methods, doi: 10.1038/NMETH.3393
発表者
理化学研究所
環境資源科学研究センター メタボローム情報研究チーム
チームリーダー 有田 正規(ありた まさのり)
特別研究員 津川 裕司(つがわ ひろし)
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.生体内の低分子化合物
生体内で作られる分子量2,000以下の化合物を指す。低分子代謝産物とも呼ばれる。 - 2.メタボロミクス
生体内に含まれる全代謝産物を指す言葉であり、各代謝物の網羅的な比較解析により生体を解き明かそうとする学問分野を指す言葉でもある。 - 3.MS-DIAL (Mass Spectrometry - Data Independent AnaLysis)
当該研究により開発された解析プログラム。質量分析データを読み込み、化合物の同定・定量、さらには多検体試料の比較解析も可能である。 - 4.液体クロマトグラフィータンデム質量分析装置(LC-MS/MS)
液体クロマトグラフィーにより分離された化合物をイオンに変換し、質量分析装置を用いて化合物の検出および定量を行う装置。質量分析装置内でイオンを壊し(フラグメント化)、その生成イオン群(プロダクトイオン)を測定することを「MS/MSスペクトルを取得する」と言う。MS/MSスペクトルの情報から、化合物構造を同定・推定することが可能である。 - 5.クロマトグラフィー
化合物を分離する方法である。化合物の固定相に対する相互作用の違いにより、混合試料の分離を行う。移動相の種類によってガスクロマトグラフィー、液体クロマトグラフィーというように分類される。 - 6.ピーク
クロマトグラフィーで化合物を分離すると、「ピーク」としてその化合物が検出され、データとして保存される。液体クロマトグラフィーにより生体試料抽出物を分析すると、中にはゴミ(夾雑物)由来のピークも複数検出されることから、生体由来であることを強調したい場合には「生体由来の化合物ピーク」と言われることもある。 - 7.MS/MSスペクトル
タンデム質量分析装置では、代謝物由来のイオンそのものを検出するだけでなく、そのイオンを開裂させて生成されるイオン群(フラグメントイオン)を検出することが可能である。このフラグメントイオンスペクトルのことをMS/MSスペクトルと呼び、化合物同定では必須の指標として用いられる。 - 8.Data Independent MS/MS Acquisition法(DIA法)
従来のData “Dependent” MS/MS Acquisitionと対比して作られた言葉である。ここでは「ある一定の質量範囲に含まれる化合物由来イオンが混在した状態でMS/MSスペクトルを取得する」技術を指す。従来のように、1つ1つ化合物由来イオンを選択してMS/MSスペクトルを取得するのではなく、選択はせずとにかく多くのMS/MSスペクトルを取得するという意味(情報非依存的)から、このように名付けられている。 - 9.予測保持時間・理論MS/MSスペクトル
化合物の立体構造からコンピュータにより推定される予測保持時間(クロマトグラフィーにおける溶出時間)とMS/MSスペクトルのことを指す。 - 10.脂質フィンガープリント
フィンガープリントとは、ここではコンピュータで扱える0と1の2値データを意味する(コンピュータはこの0と1の組み合わせで色々な信号を認識します)。脂質フィンガープリントとは、生体試料にその脂質が含まれていたら1の値を、逆に含まれていなかったら(検出されなければ)0の値を入力した、2値データを指す。 - 11.16S rRNA系統解析
16Sもしくは18S rRNAとは、タンパク質合成酵素であるリボソームの小サブユニットのRNA塩基配列のことを言う。この塩基配列は多数の生物種で登録されており、種の同定や系統分類に広く用いられている。 - 12.MassBank
日本質量分析学会の公式データベースであり、標準品マススペクトルの公開および配布を行っている。
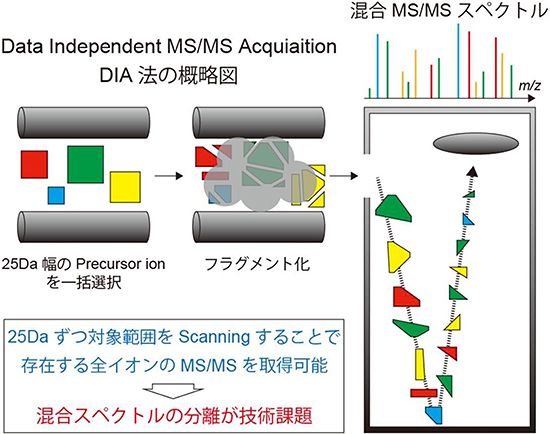
図1 Data Independent Acquisition(DIA)法の模式図
化合物由来イオンの対象範囲を25Da(たとえば100Daから125Daの代謝物)に設定した場合の模式図。一度にスキャンする範囲をある程度広げることで、最終的に存在する全イオンのMS/MSスペクトルを取得する。ただし、得られるスペクトルは複数の化合物に由来する、混合したMS/MSスペクトルである。
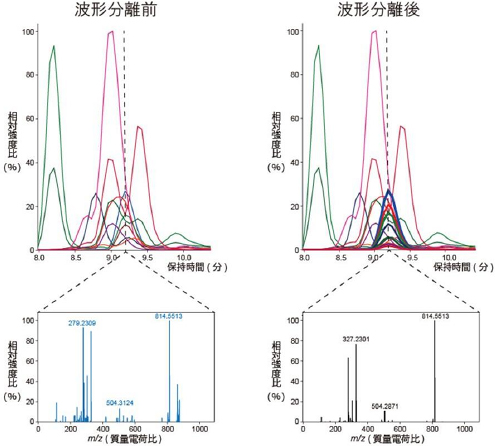
図2 MS-DIALによるMS/MSスペクトルの波形分離
DIA法で得られた混合MS/MSスペクトルがどのように波形分離され、個々のスペクトルが得られるかを示した。上図は質量電荷比の時間変化を示しており、下図は保持時間9.25分におけるMS/MSスペクトルを示している。波形分離前のMS/MSスペクトル(下図左)には、不必要な化合物由来の質量電荷比が含まれている。数学的な処理により必要なMS/MSスペクトルのみを抽出(上図右内太線)することで、必要な質量電荷比のみを得ることができる。下図左のグラフと下図右のグラフを比較すると、不要なデータが取り除かれ、個々のMS/MSスペクトルとして表示されていることが分かる。
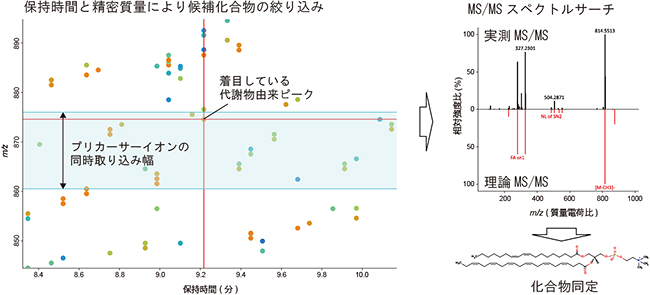
図3 MS-DIALにおける化合物同定の流れ
各スポットは検出された化合物由来ピークを示す。ターゲットとなる化合物の保持時間と精密質量の値から候補化合物の絞り込みを行う。さらに、データベースに存在する候補化合物群から、同位体比およびMS/MSスペクトルの類似度計算を行い、低分子化合物の同定(MS/MSスペクトルサーチ)を行う。
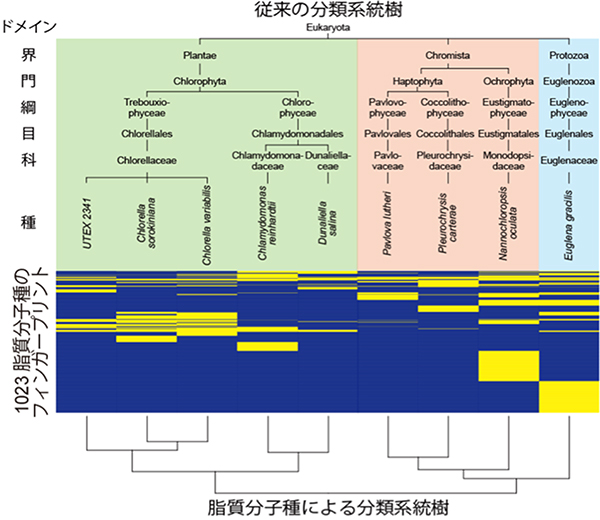
図4 脂質分子種による藻類の分類系統樹
16S rRNA系統解析による系統樹を上に、脂質分子種のフィンガープリントによる分類系統樹を下に示す。フィンガープリントとして示しているもののうち、黄色は当該藻類に対象脂質が「存在している」ことを示し、青色は「検出されなかった」ことを示す。左端のUTEX2341はクロレラ種に属するかナンノクロロプシス種に属するか議論が分かれていたが、今回の結果ではクロレラ種に属することを示唆した。
