2017年3月2日
理化学研究所
ハリー・パーキンス医療研究所
タンパク質をコードしないRNAをカタログ化
-機能的なncRNAの数は遺伝子の総数に匹敵-
要旨
理化学研究所(理研)ライフサイエンス技術基盤研究センターゲノム情報解析チームのピエロ・カルニンチ チームリーダー、ホン・ヂョン-チョウ研究員、予防医療・診断技術開発プログラムの林崎良英プログラムディレクター、川路英哉コーディネーターと、オーストラリア・ハリー・パーキンス医療研究所のアリスター・フォレスト教授らの国際共同研究グループは、27,919種の長鎖ノンコーディングRNA(lncRNA)[1]のヒトゲノム上での正確な位置や配列の特徴、細胞や組織での発現パターンを示した「ヒトlncRNAアトラス(地図)」を作成しました。このアトラスにより、19,175種のlncRNAが機能しており、そのうち1,970種は疾患に関与している可能性が示されました。
DNAから転写されるRNAには、タンパク質を作る情報を持ったメッセンジャーRNA(mRNA)と、タンパク質を作る情報を持たないノンコーディングRNA(ncRNA)があります。ncRNAのうち約200塩基以上の長さを持つ長鎖ノンコーディングRNA(lncRNA)の一部は、転写、翻訳、エピジェネティクス[2]など生体内の多様なプロセスに関与することが知られていますが、大部分のlncRNAの役割は分かっていません。lncRNAの機能解析が困難な理由として、mRNAと比較して発現レベルが低く発現パターンの解析が難しいこと、lncRNA同士で進化的に保存された配列が少ないこと、また転写開始点[3]の位置が正確に同定されていないことなどが挙げられます。
国際共同研究グループが作成した「ヒトlncRNAアトラス」は、FANTOM5プロジェクト[4]で得られたヒト長鎖RNAの5'末端[5]配列データを用いることで、lncRNAの転写開始点を正確に定めており、ヒトの代表的な細胞における発現パターンも記載しています。このアトラスをさまざまなデータセットと組み合わせた結果、19,175種のlncRNAに何らかの機能があることが示唆されました。ヒトゲノムには遺伝子が約2万あると考えられていますが、この結果はそれとほぼ同数あるいはそれ以上のlncRNAが機能している可能性を示しています。
ヒトlncRNAアトラスは、すでにインターネット上で公開されており、誰でも利用できます。lncRNA研究の有力なツールとして、現在進行中のFANTOM6プロジェクト[4]など世界各国が取り組んでいるlncRNAの機能解析を加速するものです。
本研究は、英国の科学雑誌『Nature』に掲載されるのに先立ち、オンライン版(3月1日付け:日本時間3月2日)に掲載されます。
背景
DNAから転写されるRNAには、タンパク質を作る情報を持った(タンパク質をコードする)メッセンジャーRNA(mRNA)と、タンパク質を作る情報を持たない(タンパク質をコードしない)ノンコーディングRNA(ncRNA)があります。ncRNAには、転移RNA(tRNA)[6]やリボソームRNA(rRNA)[6]などのほか、長さが18~24塩基程度のマイクロRNA(miRNA)[6]、約200塩基以上の長鎖ノンコーディングRNA(lncRNA)があります。lncRNAの一部は、転写や翻訳、エピジェネティクスの制御などを介して、細胞の分化やがん化、個体発生や疾患など生体の多様なプロセスに関与することが知られていますが、大部分のlncRNAの役割はよく分かっていません。
遺伝情報を保持するDNAが「ゲノム」として全ての細胞に等しく存在するのとは異なり、RNAやタンパク質などの生体分子は、それらの機能が必要とされる細胞や組織に発現します。従って、発現パターンや発現制御に関わるゲノム領域は、RNAの機能解析における重要な手掛かりとなります。しかし、大部分のlncRNAはmRNAと比較して発現レベルが低くその定量が難しいため、特徴的な発現パターンを見いだすのが困難でした。またRNAの発現を制御するDNA配列(プロモーター[7]やエンハンサー[8])は転写開始点(位置)の近傍に存在することが知られていますが、lncRNAの多くは転写開始点が正確に同定されていませんでした。そのため、ゲノムDNAの配列情報からlncRNAの制御領域を予想し、発現制御の観点からのアプローチは困難でした。ほかに、生存に重要な配列は種を超えて共通にみられる(進化的に保存されている)傾向があることを利用するアプローチもありますが、配列に明確な共通性がみられない機能性lncRNAも存在します。これらの理由からlncRNAの機能解析は困難でした。
研究手法と成果
国際共同研究グループはまず、FANTOM5で得られた正確な転写開始点の情報を、FANTOM5および公的データベースによる長鎖RNAの部分的な構造情報と統合した「ヒトlncRNAアトラス(地図)」を作成しました(図1A)。このアトラスは、27,919種のlncRNAで構成されており、lncRNAの転写開始点を正確に決定しているだけでなく、ヒトの代表的な細胞や組織における発現パターンも記載しています。
続いて、アトラスに遺伝学的情報やエピゲノム[2]情報を取り込むことで、lncRNAを分類しました(図1B)。その結果、遺伝子間領域に存在するlncRNAのほとんどは、遠位の転写制御領域であるエンハンサー領域から転写されるlncRNA(e-lncRNA[8])であること、またe-lncRNAは他のlncRNAよりも細胞種特異的に発現する傾向が強いことが分かりました。さらに、lncRNAの発現を制御する配列(転写開始点の近傍領域)や、転写されたlncRNA配列(エクソン[9]領域)について、ヒトと他の生物種(マウス、ラット、イヌ、ニワトリ)を比較したところ、ヒトlncRNAの64%は、進化的に保存された配列を持つことを見いだしました(図2)。
次にlncRNAの機能を推定するため、ヒトlncRNAに関する二つのリストを作成しました。一つは、特定の機能を持つ細胞や組織で発現するlncRNAのリストです。このリストから、例えば幹細胞で特異的に発現するlncRNAは多分化能の維持に関わるなどの推定が可能になります。もう一つは、疾患などのヒトの形質に関連する一塩基多型(SNPs)[10]の近傍に転写開始点を持つlncRNAのリストです。疾患関連SNPsの多くはゲノムの非コード領域に存在しており、lncRNAの転写を介して機能している可能性があります。これら二つのリストをもとに、特定の細胞種と疾患に共通するlncRNAが存在するか調べました。その結果、神経疾患に関連するSNPsを含むlncRNAは神経系組織に特異的に発現する傾向があるなど、疾患などとの関連が推定されるlncRNAが1,970種得られました。
続いて、lncRNAと発現量的形質座位関連SNPs(eQTL-SNPs)[11]の関係を調べました。eQTL-SNPsは、特定のmRNAの発現量に影響を与えるSNPであることから、その遺伝子発現を制御していることが示唆されています。eQTL-SNPsの近傍に転写開始点を持つlncRNAを調べたところ、3,166種が、そのSNPの影響を受けるmRNAと同じ発現パターンを示していました。これは、eQTL-SNPsがlncRNAの転写を介して、mRNAの発現量を制御している可能性を示唆しています。
以上の結果から国際共同研究グループは、ヒトlncRNAの19,175種に何らかの生物学的な機能があると推定しました(図2)。ヒトゲノムには遺伝子が約2万あると考えられていますが、この結果はそれとほぼ同数あるいはそれ以上のlncRNAが機能している可能性を示しています。
今後の期待
「ヒトlncRNAアトラス注)」はインターネットを通じて研究者が自由に利用でき、どのlncRNAに着目すべきかなどの検討をできます。現在進行中のFANTOM6プロジェクトでは、本研究で同定された機能を持つ可能性の高いlncRNAの情報をもとに、ncRNAの機能解析を進めていきます。
原論文情報
- Chung-Chau Hon, Jordan A. Ramilowski, Jayson Harshbarger, Nicolas Bertin, Owen J L Rackham, Julian Gough, Elena Denisenko, Sebastian Schmeier, Thomas M. Poulsen, Jessica Severin, Marina Lizio, Hideya Kawaji, Takeya Kasukawa, Masayoshi Itoh, A. Maxwell Burroughs, Shohei Noma, Sarah Djebali, Tanvir Alam, Yulia A. Medvedeva, Alison C Testa, Leonard Lipovich, Chi-Wai Yip, Imad Abugessaisa, Micka.l Mendez, Akira Hasegawa, Dave Tang, Timo Lassmann, Peter Heutink, Magda Babina, Christine A. Wells, Soichi Kojima, Yukio Nakamura, Harukazu Suzuki, Carsten O. Daub, Michiel J.L. de Hoon, Erik Arner, Yoshihide Hayashizaki, Piero Carninci, Alistair R R Forrest, "An atlas of human long non-coding RNAs with accurate 5’ ends", Nature, doi: 10.1038/nature21374
発表者
理化学研究所
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 LSA要素技術研究グループ ゲノム情報解析チーム
チームリーダー ピエロ・カルニンチ(Piero Carninci)
研究員 ホン・ヂョン-チョウ(Hon Chung-Chau)
予防医療・診断技術開発プログラム
プログラムディレクター 林崎 良英(はやしざき よしひで)
コーディネーター 川路 英哉(かわじ ひでや)
ハリー・パーキンス医療研究所 Systems biology and Genomics
教授 アリスター・フォレスト(Alistair Forrest)
(理化学研究所 客員主幹研究員)
 ピエロ・カルニンチ
ピエロ・カルニンチ
 ホン・ヂョン-チョウ
ホン・ヂョン-チョウ
 林崎良英
林崎良英
 川路英哉
川路英哉
 アリスター・フォレスト
アリスター・フォレスト
お問い合わせ先
理化学研究所 ライフサイエンス技術基盤研究センター
広報・サイエンスコミュニケーション担当 山岸 敦(やまぎし あつし)
Tel: 078-304-7138 / Fax: 078-304-7112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
お問い合わせフォーム
補足説明
- 1.長鎖ノンコーディングRNA(lncRNA)
タンパク質をコードしないノンコーディングRNAの一種。一般に、約200塩基以上のものを指す。全長にわたる保存性は高くないが、反復配列やウィルス由来の配列断片を含むものも多い。転写、翻訳、エピジェネティクスなど生体内の多様なプロセスに関与するものが知られている。 - 2.エピジェネティクス、エピゲノム
DNAの塩基配列に依存しない遺伝子の調節機構をエピジェネティクスと呼ぶ。エピジェネティクスの分子基盤は、DNAのメチル化やヒストンのメチル化/アセチル化によりゲノムの特定領域に可逆的につけられた「目印」であり、このメチル化やアセチル化の情報はしばしばエピゲノムと呼ばれる。 - 3.転写開始点
ゲノムDNAの塩基配列がRNAに転写される際、最初に写し取られる塩基。転写開始点の近傍は、遺伝子発現のタイミングや発現量の制御に重要な役割を持つ。 - 4.FANTOM5プロジェクト、FANTOM6プロジェクト
FANTOMは理化学研究所が主催する国際研究コンソーシアム。理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うことを目的に、理研ゲノム科学総合研究センターの林崎良英グループディレクター(現、理研予防医療・診断技術開発プログラム プログラムディレクター)が中心となり2000年に結成された。その成果は、iPS細胞(人工多能性幹細胞)の樹立研究など生命科学の広い分野に貢献している。5期目のプロジェクトとなるFANTOM5では、さまざまな哺乳類細胞のゲノム上の遺伝子制御部位の活性を測定し、転写状態やプロモーター活性の全容を明らかにする研究が進められた。現在のFANTOM6には20カ国、100以上の研究機関が参加し、ノンコーディングRNAの網羅的な機能解析に取り組んでいる。 - 5.5'末端
RNAはヌクレオシドの2'-リボースの5'位のヒドロキシ基と3'位のヒドロキシ基がリン酸ジエステル結合によって連結されてできている。RNAの5'位のヒドロキシ基側を5'末端と呼び、3'位のヒドロキシ基側を3'末端と呼ぶ。RNAの5'末端はDNA上の転写開始点に相当する。 - 6.転移RNA(tRNA)、リボソームRNA(rRNA)、マイクロRNA(miRNA)
tRNAとrRNAは、メッセンジャーRNA(mRNA)からタンパク質が翻訳される過程で機能するRNA。miRNAは、長さ18~24塩基程度の1本鎖RNAで、他の遺伝子の発現調節などの機能を担う。いずれもタンパク質へは翻訳されないが、その生合成のプロセスや機能はよく研究されている。 - 7.プロモーター(近位発現制御領域)
ゲノムDNA上でRNAに書き写される領域の近くにあり、遺伝子を発現させる機能を持つ部分をプロモーター領域(配列)という。 - 8.エンハンサー(遠位発現制御領域)、e-lncRNA
エンハンサー(enhancer)領域(配列)は、ゲノムDNA上で制御対象である遺伝子とは遠く離れた領域に位置し、遺伝子の転写効率を向上させる働きを持つ。エンハンサーが活性化する際、エンハンサー配列からノンコーディングRNAが転写されることが近年報告され、エンハンサーRNA(eRNA)もしくはe-lncRNAと呼ばれている。 - 9.エクソン
mRNAのスプライシング過程では、前駆体RNAからイントロンと呼ばれる領域が切り出され、残ったRNA(エクソン)がつなぎ合わさって成熟したmRNAとなる。lncRNAにもmRNAと同様のスプライシングを受けるものが数多く存在する。 - 10.一塩基多型(SNPs)
ヒトゲノムの配列は、全ての人々が同じというわけではなく、数百から約1,000塩基対の割合で1つの塩基の違いがある。その中でも人口中1%以上の頻度で存在するものを一塩基多型(SNP:single nucleotide polymorphism)という。一塩基多型には、単遺伝子疾患を引き起こしたり、多遺伝子疾患の発症を早めたりする変異から、個人の表現型に影響を及ぼさない中性的な多型までさまざまなものがある。さらに、代謝経路において差異を引き起こす一塩基多型も存在していて、薬物の応答性や副作用の個人間差に関係する。 - 11.発現量的形質座位関連SNPs(eQTL-SNPs)
背の高さや体重など連続的な数値で表される形質を量的形質と呼び、量的形質に関与する遺伝子座のことをQTL(Quantitative Trait Locus)と呼ぶ。mRNAの発現(expression)の強さも量的形質と見なすことができるため、発現量に影響を与えるSNPをeQTL-SNPsと呼ぶ。
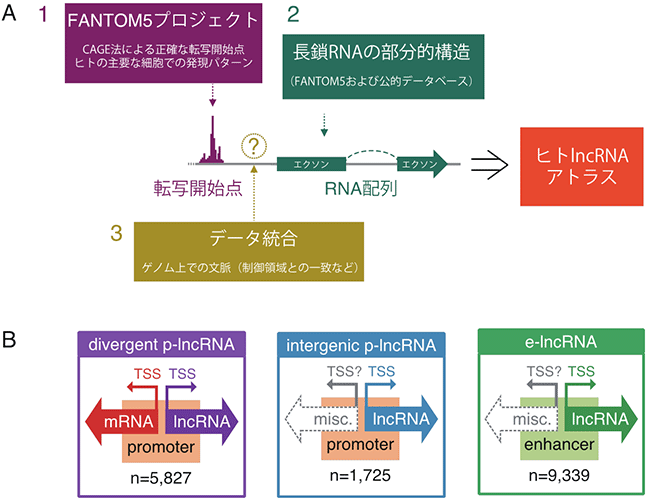
図1 ヒトlncRNAアトラスの概要
A:ヒトlncRNAアトラスのデザイン。FANTOM5で得られた正確な転写開始点の情報(1)と、長鎖RNAの部分構造情報(2)を、統合した(3)。
B:アトラスにより分類したlncRNAの三つのカテゴリ。「divergent p-lncRNA」および「intergenic p-lncRNA」はプロモーターに類似した配列に由来し、それぞれ、mRNAの転写開始点(TSS; Transcription start site)から(mRNAとは異なる転写産物として)転写されたもの、遺伝子から離れた領域(遺伝子間領域)に転写開始点を持つものを指す。「e-lncRNA」は、遺伝子間領域に転写開始点があるもののうち、エンハンサー領域から転写されるもの。
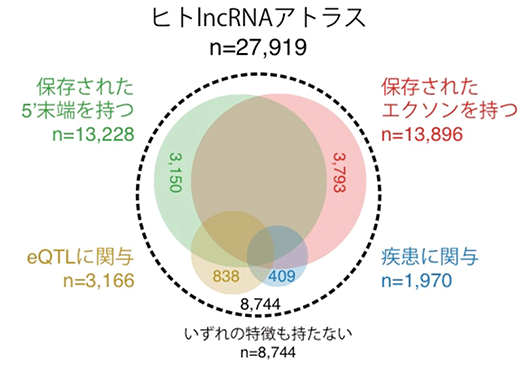
図2 ヒトlncRNAの機能推定
ヒトlncRNAアトラスに登録されている27,919種のlncRNAの内訳。保存された5’末端を持つもの(13,228種)、保存されたエクソン領域(13,896種)を持つもの、eQTLに関わるもの(3,166種)、疾患に関わるもの(1,970種)は、機能的なlncRNAであると考えられる。これらいずれかの特徴を持つlncRNAの総数は、19,175種となった。
