2017年8月22日
理化学研究所
ハリー・パーキンス医療研究所
マイクロRNAをより詳細にカタログ化
-発現パターンも網羅した地図で疾患に関わるmiRNA研究を加速-
要旨
理化学研究所(理研)ライフサイエンス技術基盤研究センターゲノム情報解析チームのデレック・デ・リー実習生(研究当時)、ゲノムデータ解析アルゴリズム開発ユニットのミヒル・デ・ホーン ユニットリーダー、トランスクリプトーム研究チームのピエロ・カルニンチ チームリーダー、予防医療・診断技術開発プログラムの林崎良英プログラムディレクターと、ハリー・パーキンス医療研究所のアリスター・フォレスト教授らの国際共同研究グループは、さまざまなヒト細胞で発現するマイクロRNA(miRNA)[1]を網羅的に記載したアトラス(地図)を作成しました。
DNAから転写されたRNAには、タンパク質の情報を含まないノンコーディングRNA(ncRNA)[2]が大量に存在します。miRNAと呼ばれる短い(21~23塩基)ncRNAは、標的となるメッセンジャーRNA(mRNA)に結合して翻訳抑制を行い、遺伝子を抑制的に制御します。miRNAは細胞の分化や個体発生などさまざまな生命現象に関与し、その発現異常はがんをはじめとする疾患と関係することから、多くの研究者がmiRNAの解析に取り組んでいます。miRNAの多彩な機能や疾患との関わりを解明する上で、miRNAの配列などをまとめたデータベースは重要な研究ツールですが、発現パターンや転写を制御するプロモーター[3]まで網羅したデータベースは存在しませんでした。
国際共同研究グループは、FANTOM5プロジェクト[4]で用いたヒトおよびマウスの、100種を超えるさまざまな初代培養細胞[5]に由来するRNAサンプルを利用し、miRNAの配列を詳しく解析しました。そして、既知のmiRNAだけでなく、新たなmiRNA候補の発現パターンやプロモーター配列を多数収録した「miRNA発現アトラス」を作成しました。miRNAは、前駆体であるpri-miRNAがさまざまなプロセスを経て成熟しますが、今回の解析から、miRNAの発現量は主としてpri-miRNAの転写の段階で制御されていることが示されました。また網羅的な発現解析から、特定の細胞種に顕著に発現するmiRNAや、特定の細胞種で発現が抑制されているmiRNAの存在が明らかになりました。
miRNA発現アトラス(英語)はインターネット上で公開されており、誰でも利用できます。これまで解析されていなかった細胞で発現しているmiRNAや、細胞種特異的に発現・抑制されているmiRNAを検索することが可能になるなど、miRNAの発現制御の解明につながる大きな手がかりとして、今後のmiRNA研究を加速させることが期待できます。
本研究は、国際科学雑誌『Nature Biotechnology』に掲載されるのに先立ち、オンライン版(8月21日付け:日本時間8月22日)に掲載されます。
背景
DNAから転写されるRNAには、タンパク質を作る情報を持った(タンパク質をコードする)メッセンジャーRNA(mRNA)と、タンパク質を作る情報を持たない(タンパク質をコードしない)ノンコーディングRNA(ncRNA)があります。ncRNAのうち長さが21~23塩基のマイクロRNA(miRNA)は、標的となる塩基配列を持つmRNAに結合してmRNAの分解を促進したり、翻訳を阻害する作用を示すことにより、遺伝子機能を抑制して制御する働きがあります。miRNAは、細胞の分化や発生などさまざまな生命現象に関与することが知られています。また、複数のmiRNAががん化を促進または抑制する機能を持つことが報告されており、多くの研究者がmiRNAの発現異常と疾患との関係の解明に取り組んでいます。
miRNAは、まずヘアピン構造[6]を持つ長いRNA(pri-miRNA)として転写されます(図1)。続いて、ヘアピン構造の基部を切断するRNA分解酵素Drosha[7]により、中間産物のpre-miRNAが切り出されます。pre-miRNAはさらにDicer[7]によって切断されて2本鎖のmiRNAとなった後、その片方もしくは両方が成熟miRNAとして機能します。これまでに報告されたmiRNAを集めたデータベース「miRBase注1)」には1,881種のヒトpre-miRNA配列と2,588個の成熟miRNAが収録されています。しかし、miRNAの発現パターンや、pri-miRNAの転写を制御するプロモーター配列まで網羅的に記載したデータベースは存在しませんでした。
注1)Kozomara, A. & Griffiths-Jones, S. miRBase: annotating high confidence miRNAs using deep sequencing data. Nucleic Acids Res 42, D68–D73 (2014).
研究手法と成果
FANTOM5では、ゲノム上の遺伝子制御部位の活性を網羅的に測定するため、ヒトやマウスの、さまざまな細胞からRNAを抽出し、CAGE法[8]により正確な5'末端配列を決定・定量しました(図1)。これにより、そのRNAがゲノムDNA上のどのプロモーターから、どれくらい転写されたかを測定することができます。本研究ではさらに、CAGE解析を行った細胞と同じRNAサンプルに対して、miRNAを含む短いRNA(sRNA)に特化した配列解読(sRNAシーケンス、図1)を行いました。こうして、ヒトの100種以上の初代培養細胞やiPS細胞(人工多能性幹細胞)[9]などで発現するmiRNAについて、CAGEデータとsRNAデータを照らし合わせることが可能となりました。またマウスについても同様の作業を、初代培養細胞を中心に行いました。
得られたsRNA配列をmiRBaseと照会したところ、既知のmiRNAのほとんど(ヒトで98%、マウスで95%)が、今回作製したsRNAライブラリに含まれていました。また、sRNAの配列からmiRNAを予測するソフトウェア(miRDeep2注2))を用いて解析したところ、sRNAライブラリから、ヒトでは6,543個、マウスでは1,444個の新しいmiRNA候補を得ることができました。これらのmiRNA候補のほとんどは発現が低いため、これまでの解析では見落とされていたと考えられます。本研究では、個々のmiRNAがどの細胞でどれだけ発現しているかを定量化しており、発現パターンの類似度でmiRNAを分類するなど詳細な解析が可能です(図2)。
既知のmiRNAの細胞特異的な発現について調べたところ、特定の細胞のみに強く発現するものと、特定の細胞で発現が抑制されているものが存在しました(図3)。一方、新しいmiRNA候補については、特定の細胞に限定して発現するものがほとんどでした。
CAGE法は、Cap(キャップ)と呼ばれるRNAの5'末端に特徴的な構造を利用することで、転写開始点から始まるRNA配列を読み取る方法です。miRNAは複数の段階を経て成熟しますが(図1)、CAGE法で捉えられる最も顕著なシグナルは、pri-miRNAの転写開始点となります。一方、小型魚類ゼブラフィッシュのmiRNAを解析した先行研究から、pre-miRNAの生成時にRNA分解酵素Droshaによって切り出されるpri-miRNAの5'末端配列(pre-miRNAの3'側切断部位)もCAGE法で検出されることが示されていました注3)。
今回、FANTOM5のヒトとマウスを対象にしたCAGE解析でも、Droshaの切断配列が捉えられていることが確認できました。Drosha切断配列が確認できたものは、miRBaseに登録されているpre-miRNAのうち、実際に機能していると推定できる信頼度の高いものに集中していました(図4)。これは、新しいmiRNAの候補配列の検定に、CAGE法によるDrosha切断配列の解析が有効であることを示しています。
さらに、CAGE法で得られたpri-miRNAの転写開始点の情報とゲノム配列の情報を組み合わせ、ヒトでは1,357個、マウスでは804個のpri-miRNAプロモーターを同定しました。これらのプロモーター配列はヒトとマウスで保存されており、転写レベルでの発現制御が種を超えて重要であると推測されました。また、CAGE法で測定されたpri-miRNAの転写量と、sRNA解析で測定した成熟miRNAの発現量は相関していることが分かりました。これらの結果は、miRNAの発現は主としてpri-miRNAの転写で制御されていることを示しています。また、pri-miRNAの転写と成熟miRNAの発現量の相関を利用することで、CAGE解析のみで細胞内のmiRNA量の推定が可能になると考えられます。
miRNAには、異なるゲノム領域から転写されたpri-miRNAが同じ配列を持つ成熟miRNAを産生する場合があり、これらはパラログ(paralogous)miRNAと呼ばれます。miRNA配列からは区別ができないパラログmiRNAの発現も、CAGEデータがあればどちらのpri-miRNAがどれくらい転写されているかを測定できるため、異なる発現制御を受けるパラログmiRNAの解析が容易となります(図5)。
注2)Friedländer, M.R.et al. miRDeep2 accurately identifies known and hundreds of novel miRNA genes in seven animal clades. Nucleic Acids Res.40, 37–52 (2012).
注3)Nepal, C. et al.Transcriptional, post-transcriptional and chromatin-associated regulation of pri-miRNAs, pre-miRNAs and moRNAs..Nucleic Acids Res. 44, 3070–3081 (2015).
今後の期待
多数の新しいmiRNA候補配列を含むsRNAライブラリとそれらの発現データ、プロモーター配列を統合した「miRNA発現アトラス」は、インターネットを通じて誰でも自由に利用できます。
miRNA発現アトラス(英語)
これまで解析されていなかった細胞で発現しているmiRNAや、細胞種特異的に発現・抑制されているmiRNAを検索することが可能になるなど、miRNAに注目している研究者にとって必須のデータベースとなり、今後のmiRNA研究を加速させることが期待できます。
原論文情報
- Derek de Rie, Imad Abugessaisa, Tanvir Alam, Erik Arner, Peter Arner, Haitham Ashoor, Gaby Astrom, Magda Babina, Nicolas Bertin, A. Maxwell Burroughs, Carsten O. Daub, Michael Detmar, Ruslan Deviatiiarov, Alexandre Fort, Claudia Gebhard, Dan Goldowitz, Sven Guhl, Jayson Harshbarger, Akira Hasegawa, Kosuke Hashimoto, Peter Heutink, Chung Chau Hon, Edward Huang, Yuri Ishizu, Takeya Kasukawa, Peter Klinken, Timo Lassmann, Charles-Henri Lecellier, Weonju Lee, Marina Lizio, Vsevolod Makeev, Anthony Mathelier, Yulia A. Medvedeva, Christopher J. Mungall, Shohei Noma, Mitsuhiro Ohshima, Helena Persson, Filip Roudnicky, Pal Saetrom, Jessica Severin, Jay W. Shin, Hiroshi Tarui, Kristoffer Vitting-Seerup, Louise Winteringham, Yoko Yamaguchi, Kayoko Yasuzawa, Christine Wells, Kim M. Summers, Hideya Kawaji, Albin Sandelin, Michael Rehli, the FANTOM consortium, Yoshihide Hayashizaki, Piero Carninci, Alistair R. R. Forrest, Michiel J. L. de Hoon., "An integrated expression atlas of miRNAs and their promoters in human and mouse", Nature Biotechnology, doi: 10.1038/nbt.3947
発表者
理化学研究所
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 LSA要素技術研究グループ ゲノム情報解析チーム
実習生(研究当時) デレック・デ・リー(Derek de Rie)
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 LSA要素技術研究グループ ゲノムデータ解析アルゴリズム開発ユニット
ユニットリーダー ミヒル・デ・ホーン(Michiel J. L. de Hoon)
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 LSA要素技術研究グループ トランスクリプトーム研究チーム
チームリーダー ピエロ・カルニンチ(Piero Carninci)
予防医療・診断技術開発プログラム
プログラムディレクター 林崎 良英(はやしざき よしひで)
ハリー・パーキンス医療研究所 Systems biology and Genomics
教授 アリスター・フォレスト(Alistair Forrest)
(理研ライフサイエンス技術基盤研究センター機能性ゲノム解析部門
LSA要素技術研究グループ ゲノム情報解析チーム 客員主管研究員)
 デレック・デ・リー
デレック・デ・リー
 ミヒル・デ・ホーン
ミヒル・デ・ホーン
 ピエロ・カルニンチ
ピエロ・カルニンチ
 林崎良英
林崎良英
 アリスター・フォレスト
アリスター・フォレスト
お問い合わせ先
理化学研究所 ライフサイエンス技術基盤研究センター
広報・サイエンスコミュニケーション担当 山岸 敦(やまぎし あつし)
Tel: 078-304-7138 / Fax: 078-304-7112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
産業利用に関するお問い合わせ
理化学研究所 産業連携本部 連携推進部
お問い合わせフォーム
補足説明
- 1.マイクロRNA(miRNA)
細胞内に存在する長さ21~23塩基程度の1本鎖RNA。数百~数千の塩基の一次転写産物から段階的に切り出されて作られる。タンパク質へは翻訳されず、標的となるmRNAの分解、翻訳抑制を通して、遺伝子機能の抑制に働く。 - 2.ノンコーディングRNA(ncRNA)
non-coding RNA。メッセンジャーRNA(mRNA)と異なり、タンパク質の設計図として用いられないRNAの総称。エピジェネティクス(塩基配列に依存しない遺伝子の調節機構)や転写、翻訳といった生物の活動の中枢をなす反応、幹細胞性の維持など、さまざまな働きに関与するncRNAが次々に報告されており、その重要性に注目が集まってきている。 - 3.プロモーター
ゲノムDNA上でRNAに書き写される領域の近くにあり、遺伝子を発現させる機能を持つ部分をプロモーター領域(配列)という。 - 4.FANTOM5プロジェクト
FANTOM(Functional ANnoTation Of the Mammalian genome)は理化学研究所が主催する国際研究コンソーシアム。理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うことを目的に、理研ゲノム科学総合研究センターの林崎良英グループディレクター(現、予防医療・診断技術開発プログラム プログラムディレクター)が中心となり2000年に結成された。その成果は、iPS細胞(人工多能性幹細胞)の樹立研究など生命科学の広い分野に貢献している。5期目のプロジェクトとなるFANTOM5では、さまざまな哺乳類細胞のゲノム上の遺伝子制御部位の活性を測定し、転写状態やプロモーター活性の全容を明らかにする研究が進められた。現在のFANTOM6には20カ国、約60の研究機関が参加し、ノンコーディングRNAの網羅的な機能解析に取り組んでいる。 - 5.初代培養細胞
初代細胞とも呼ぶ。生体から採取した組織や細胞を最初に培養した段階の、原則として未分裂の細胞を指す。採取してからの時間が短いため、生体内と同様の挙動をすることが期待される。 - 6.ヘアピン構造
1本鎖のRNAは、分子内で塩基どうし(AとU、CとG)が水素結合することに局所的に2本鎖構造を形成する。2本鎖に挟まれたRNAが折れ曲がってループ状になる構造をヘアピン構造と呼ぶ。 - 7.Drosha、Dicer
2本鎖RNAを認識し、RNA分子の内部を切断する酵素の一種。標準的なmiRNA生成過程では、pri-miRNAからpre-miRNAを切り出すDroshaと、pre-miRNAを切断するDicerが主に働く。 - 8.CAGE法
理研が独自に開発した手法で、耐熱性逆転写酵素やmRNAのCap構造を捕捉する技術を組み合わせて転写産物の5'末端の塩基配列を決定する実験手法。この塩基配列を読み取ってゲノム配列と照らし合わせて、どこから転写が始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる。Cap Analysis Gene Expressionの略。 - 9.iPS細胞(人工多能性幹細胞)
体細胞に特定の遺伝子を導入することで得られる、多様な細胞に分化できる分化多能性と自己複製能を獲得した細胞。京都大学の山中伸弥教授らのグループにより最初に樹立された。
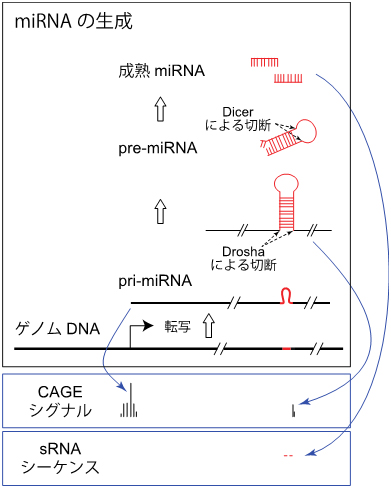
図1 miRNAの生成とCAGE法・sRNAシーケンス法
miRNAの一般的な生成過程。最終的にmiRNAとなるヘアピン領域を含む長いRNAがまず転写され、複数のプロセシングを受けて1本鎖の成熟miRNAが作られる。CAGE法による解析では、pri-miRNAの5'末端と、RNA分解酵素Droshaにより切断される3'側のpri-miRNA配列を同定・定量できる。またsRNAシーケンスは成熟したmiRNAの配列の解読に適した解析法である。CAGEデータとsRNAデータを合わせることで、miRNAの詳細な発現解析、プロモーター解析が可能となる。
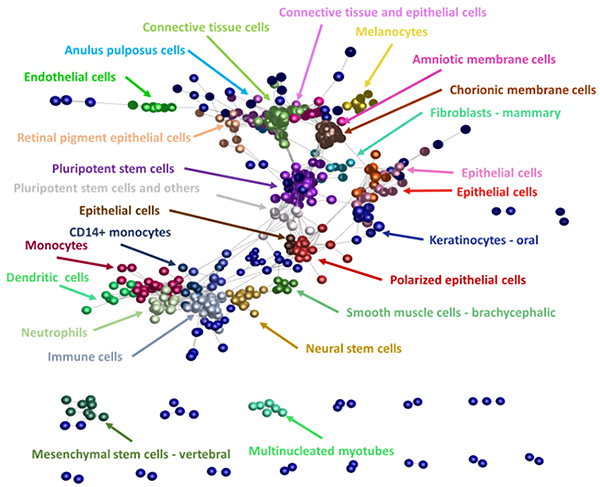
図2 ヒト初代培養細胞で発現するmiRNA
ヒト初代培養細胞で発現するmiRNAのうち、信頼度の高い(実際に機能している可能性が高い)ものを選んで網羅的な定量解析を行い、発現パターンの類似度をもとにmiRNA同士の関係をネットワークとして描画した。個々の点はmiRNAを示し、クラスターを指す矢印はそれらのmiRNAを強く発現する細胞種を示す。ネットワークの描画には、Miru(Freeman, T.C., et al. Construction, visualisation, and clustering of transcription networks from microarray expression data. PLoS Comput. Biol. 3, 2032–2042 (2007))を用いた。
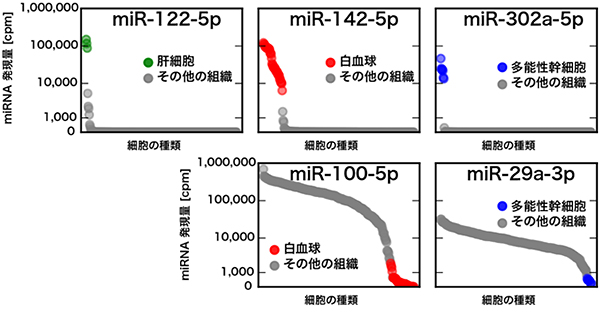
図3 細胞種特異的な発現パターンを示すmiRNA
5種の既知のmiRNAについて、ヒトのさまざまな初代培養細胞や多能性幹細胞での発現量を調べた。上段の三つのグラフで記載したmiRNAは、特定の細胞種で強く発現し、他の細胞ではほとんど発現していない。逆に下段の二つのmiRNAの場合、多くの細胞で広く発現しているが、特定の細胞種で発現が低下している。グラフの縦軸は発現量を示し、横軸は100種を超える初代培養細胞を発現量の高い順に並べている。
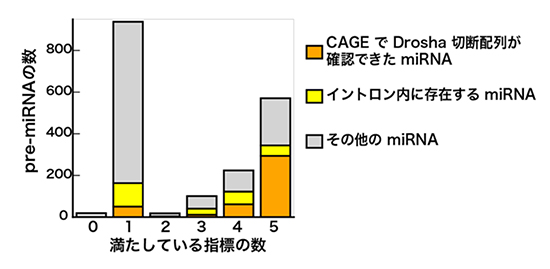
図4 Drosha切断配列が確認されたmiRNA
miRBaseは、構造予測や発現量をもとに五つの指標を設定し、登録されたmiRNAがそれらの指標をどれだけ満たしているかを検定している。グラフは、miRBaseに登録されたpre-miRNAについて、五つの指標を満たすものがどれだけあるかを示したもの。横軸の数字が大きいほど多くの指標を満たしており、信頼度が高いと推定できる。Drosha切断配列が確認できたmiRNA(オレンジ色)は、信頼度の高いものに多く含まれていることが分かる。なお黄色バーは、メッセンジャーRNA(mRNA)生成の際に切り出されるイントロン領域に由来するmiRNA(Mitrons)であり、Droshaの関与なしに成熟する。
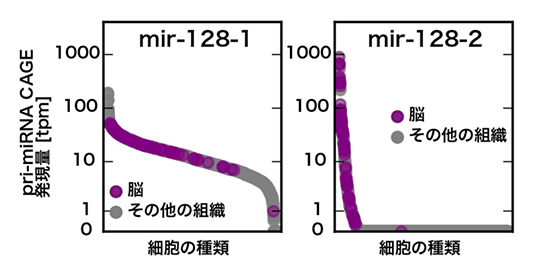
図5 パラログmiRNAの発現に寄与するpri-miRNAの判定
パラログmiRNAであるmir-128-1とmir-128-2は、同じ配列を持つ成熟miRNAが異なるゲノム領域に存在する二つのpri-miRNAから産生される。CAGE解析の結果から、mir-128-1を産生するpri-miRNAは多くの細胞で発現しているのに対し、mir-128-2を産生するpri-miRNAは脳細胞に特化した分布を示した。これらのパラログmiRNAが異なる発現制御を受けていることを示す。グラフの縦軸は発現量を示し、横軸は100種を超える初代培養細胞を発現量の高い順に並べている。
