2020年2月24日
理化学研究所
RNA-クロマチン相互作用を推定する新技術「RADICL-seq」
-長鎖ノンコーディングRNAの機能の推定が可能に-
理化学研究所(理研)生命医科学研究センタートランスクリプトーム研究チームのアレッサンドロ・ボネッティ客員研究員、ピエロ・カルニンチチームリーダーらの研究チームは、細胞核内のRNAとゲノムDNA(クロマチン[1])の相互作用をゲノム全体で調べることができる「RNA And DNA Interacting Complexes Ligated and sequence;RADICL-seq」法を新たに開発しました。
本研究成果は、「長鎖ノンコーディングRNA(lncRNA)[2]」の機能を推定することに役立ち、今後、核内のRNAを研究する技術として広く利用されると期待できます。
RNAには、lncRNAをはじめ機能が分かっていないものが多く存在します。その解明には、RNAと相互作用するクロマチン(DNAとヒストンタンパク質からなる)の領域を知ることが必要です。しかし、それらを包括的に調べる技術は限られており、新たな技術開発が必要とされていました。
今回、研究チームは、核内のRNAを網羅的に調べられるRADICL-seq法を開発しました。この方法により、ゲノムDNAの遺伝子発現調節やクロマチン構造の維持に重要な役割を果たすRNAなどさまざまな核内RNAと、それらが相互作用するゲノム領域の情報を同時に捉えることが可能になります。また、この方法によって得られたデータは、クロマチンがどのように構成・調節されるかの検証、またlncRNAの機能の網羅的な推定にも役立ちます。
本研究は、オンライン科学雑誌『Nature Communications』(2月24日付:日本時間2月24日)に掲載されます。
背景
DNAから転写されるRNAには、タンパク質を作る情報を持ったメッセンジャーRNA(mRNA)と、タンパク質を作る情報を持たないノンコーディングRNA(ncRNA、非コードRNA)があります。ncRNAのうち、約200塩基以上の長さを持つRNAは「長鎖ノンコーディングRNA(lncRNA)」と呼ばれます。lncRNAの一部は転写や翻訳、そしてDNAとヒストンタンパク質からなる「クロマチン」の構造変化による遺伝子発現の調節機構(クロマチンリモデリング)を含めた生体内の多様なプロセスに関わっています。
lncRNAの機能を知るためには、lncRNAと相互作用するクロマチンの領域を知ることが重要です。しかし、ChIRP-seq法[3]などの従来法では、標的とする1種類のlncRNAが相互作用するクロマチン領域を特定することしかできず、数多く存在する未知のlncRNAがどのように相互作用しているかを明らかにすることはできませんでした。そこで研究チームは、核内のRNA-クロマチンの相互作用をゲノム全体で検出する技術の開発を行いました。
研究手法と成果
研究チームが開発した「RNA And DNA Interacting Complexes Ligated and sequence;RADICL-seq)」法では、まず細胞核内に存在するRNA、タンパク質、DNAをホルムアルデヒドによって固定し、独自にデザインしたアダプター配列を介してRNAとDNAを連結させます(図1上段)。次に、固定されていたタンパク質を取り除き、アダプターに結合しているRNAとDNAだけを取り出します。得られるRNAやDNAの長さはさまざまなため、一定の長さになるように酵素で切断します。これらを次世代シークエンサー[4]で読み取り、ゲノムにマッピングします(図1下段)。これにより、DNAとその近傍に存在するRNAを網羅的に知ることができます。
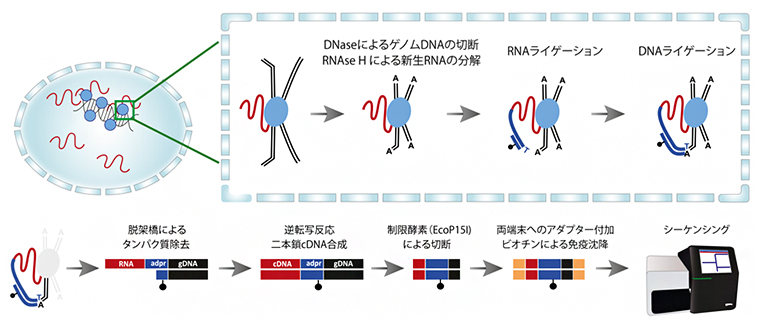
図1 RADICL-seqの概要
- 上段:左は、核内のDNA結合タンパク質(青丸)に架橋されたRNA(赤線)の様子。右枠内は、核内での反応を示す。RNAはタンパク質に固定され、クロマチンを構成するゲノムDNA(黒線)と結合する。リボヌクレアーゼH(RNAseH)は、ライブラリ内の新生RNA(RNAはゲノムのあらゆる場所に転写される)の量を減らし、デオキシリボヌクレアーゼ(DNAse)はゲノムDNAを切断化する。アダプター配列(青線)の一方はRNAと結合し(RNAライゲーション)、もう一方はDNAと結合する(DNAライゲーション)ことで、RNAとDNAを連結させる。これはのちにクロマチンに結合したRNAを分離するための重要なステップとなる。
- 下段:脱架橋によりタンパク質を除去すると、RNA(赤)、アダプター配列(青)、DNA(黒)が連結された分子が残る。逆転写反応によりRNAは相補的DNA(cDNA)に変換され、制限酵素によって両側のRNAとDNAが一定の長さに切断される。最後に、シーケンシングのためのアダプター配列(オレンジ)を両側に付加し、ビオチン(黒丸)により免疫沈降させ、塩基配列を読み取る。
開発したRADICL-seq法を実証するために、マウスのES細胞[5](mESC)とオリゴデンドロサイト前駆細胞[6](mOPC)を用いて実験を行いました。図2は、lncRNAであるNeat1[7]とmRNAであるFgfr2[8]が、ゲノムのどの部分と相互作用しているか示しています。19番染色体上にコードされているNeat1は、ES細胞とオリゴデンドロサイト前駆細胞では、異なるゲノム領域と相互作用しています(図2a,b)。また、7番染色体上にコードされているFgfr2は、ES細胞とオリゴデンドロサイト前駆細胞では、主に7番染色体と相互作用していることが分かります(図2c,d)。このように、RADICL-seq法では、さまざまなRNA-クロマチン間の相互作用に対して、細胞ごとにさまざまなパターンを明らかにすることができます。
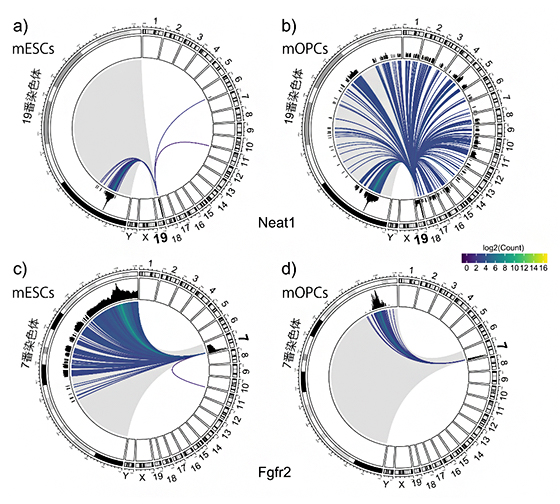
図2 Neat1とFgfr2のRNA-クロマチン相互作用を示すゲノムマップ
マウス染色体は19本の常染色体とX、Yの性染色体からなる。環状のおよそ右半分は、これら21本の染色体を表す。左半分はNeat1がある19番染色体、またはFgfr2がある7番染色体を拡大表示したもの。
- a-b:19番染色体から転写されるlncRNAであるNeat1のゲノムマップ。全ての線は、Neat1のRNAが転写される19番染色体を始点とし、相互作用するゲノム領域を終点とする。Neat1が19番染色体と相互作用する場合には、終点は拡大された19番染色体上になる。aのマウスES細胞(mESC)では、Neat1は19番、6番、11番染色体とだけ相互作用している。bのマウスオリゴデンドロサイト前駆細胞(mOPC)では、Neat1は全ての常染色体と相互作用している。
- c-d:脱架橋によりタンパク質を除去すると、RNA(赤)、アダプター配列(青)、DNA(黒)が連結された分子が残る。逆転写反応によりRNAは相補的DNA(cDNA)に変換され、制限酵素によって両側のRNAとDNAが一定の長さに切断される。最後に、シーケンシングのためのアダプター配列(オレンジ)を両側に付加し、ビオチン(黒丸)により免疫沈降させ、塩基配列を読み取る。
遺伝子の発現は、核内に存在するRNAの特性によって調節されています。ゲノムDNAの塩基配列がRNAに転写される際、写し取られる塩基が存在する転写部位の周辺で、RNAはクロマチンと相互作用をします。その際、RNAが転写された元の染色体のクロマチンと相互作用することを「シス相互作用」、RNAと由来が異なる染色体のクロマチンと相互作用することを「トランス相互作用」と呼びます。
RADICL-seq法により、既知のRNA-クロマチン構造を再現することや、新しい相互作用を特定することが可能になりました。その結果、トランス相互作用するRNAにはノンコーディングRNAが比較的多く、シス相互作用するRNAにはイントロン[9]領域のRNA(mRNAの一部かもしくは独立したノンコーディングRNAかは不明)が多い傾向が見られました。これにより、mRNAにあるイントロン領域が遺伝子発現の調節因子として働く可能性が新たに示されました。
また、RNAポリメラーゼII[10]の働きを阻害し、新たなRNAの転写を抑制した条件下においても、RNA-DNA相互作用が検出されたことから、クロマチンに安定に結合した転写産物が存在することが分かりました。さらに、タンパク質に媒介されないRNA-クロマチン相互作用が存在することが分かったことから、遺伝子発現の調節にRNAとDNAのハイブリッド構造であるRループ[11]や、RNA-DNAの三重らせん[12]が関与している可能性が示されました。
今後の期待
本研究により、ゲノム生理学におけるRNAの遺伝子発現に関わる調節的役割を明らかにするための、複数の細胞種におけるRNA-クロマチンインタラクトーム解析[13]の準備が整いました。RADICL-seq法は、FANTOM6プロジェクト[14]や他の関連プロジェクトにおいて、今後、真核生物ゲノムの遺伝子発現調節におけるRNAの役割を理解するための重要な技術として広く利用されると期待できます。
補足説明
- 1.クロマチン
ゲノムDNAがヒストンタンパク質に巻き付くことで形成されるDNA・タンパク質複合体。クロマチン構造は遺伝子の転写に関連し、転写の活性化した遺伝子座や、それに関係する転写制御領域では開いた状態となり、オープンクロマチン領域と呼ばれる。 - 2.長鎖ノンコーディングRNA(lncRNA)
タンパク質をコードしないRNA(ノンコーディングRNA)の一種。一般に、約200塩基以上のものを指す。反復配列やウイルス由来の配列断片を含むものが多く、配列の保存性はコーディング遺伝子より低い。転写、翻訳、エピジェネティクスなど生体内の多様なプロセスに関与するものが知られている。 - 3.ChIRP-seq法
Chromatin Isolation by RNA Purificationの略。特定のlncRNAに相補的に結合するDNAオリゴヌクレオチドを使い、目的のlncRNAを抽出して、それらに結合しているDNA断片を回収し、次世代シーケンサーを用いてそのlncRNAがゲノム上のどの場所に結合しているかを解析する方法。 - 4.次世代シークエンサー
2005年ごろに米国で開発された、遺伝子情報を桁違いに大量・高速に検出できる装置。 - 5.ES細胞
脊椎動物の初期胚が持つ、全ての種類の体細胞へ分化する能力を多能性という。多能性を持ち、試験管内で培養して無限に増やすことができる細胞を多能性幹細胞という。ES細胞は、哺乳類の着床前胚(胚盤胞)に存在する内部細胞塊から作製された多能性幹細胞。 - 6.オリゴデンドロサイト前駆細胞
オリゴデンドロサイトは神経細胞の軸索に巻き付いて、髄鞘(ミエリン鞘)を形成するグリア細胞。神経細胞の正常な機能維持に寄与している。オリゴデンドロサイト前駆細胞は、オリゴデンドロサイトに分化する前の状態にある細胞。 - 7.Neat1
lncRNAの一つで、複数のタンパク質と相互作用して核内構造体であるパラスペックル(遺伝子発現制御に関わるRNA-タンパク質複合体)を形成する。 - 8.Fgfr2
受容体型チロシンキナーゼの一つで、細胞外の分子を受け取り、チロシンのリン酸化を通して細胞内にそのシグナルを伝える。 - 9.イントロン
DNAにコードされている遺伝子はエキソンとイントロンで構成され、どちらもRNAとして転写されるが、イントロンの部分は後に切り取られる。残ったエキソンの部分が核の外に輸送されて、タンパク質に翻訳される。 - 10.RNAポリメラーゼII
DNAの塩基配列を読み取って相補的なRNAを合成する酵素の一つで、主に真核生物の核内でメッセンジャーRNA(mRNA)の転写を担う。 - 11.Rループ
二本鎖DNAが開いて片方が一本鎖となり、もう一方の鎖が転写物であるRNAとハイブリッドを形成してできる構造。 - 12.RNA-DNAの三重らせん
二本鎖DNAに一本鎖のRNAが加わり安定化した構造。 - 13.インタラクトーム解析
生体内分子間の相互作用を網羅的に解析すること。タンパク質が生物学的な機能を発揮する際に他の生体分子と相互作用するときなどの総称をいう。 - 14.FANTOM6プロジェクト
FANTOMは理化学研究所が主催する国際研究コンソーシアム。理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うことを目的に2000年に結成された。その成果は、iPS細胞(人工多能性幹細胞)の樹立研究など生命科学の広い分野に貢献している。6期目のプロジェクトとなるFANTOM6には20カ国、100以上の研究機関が参加し、ノンコーディングRNAの網羅的な機能解析に取り組んでいる。
研究チーム
理化学研究所 生命医科学研究センター トランスクリプトーム研究チーム
客員研究員 アレッサンドロ・ボネッティ(Alessandro Bonetti)
(現 カロリンスカ研究所 研究員)
専任研究員 橋本 浩介(はしもと こうすけ)
研究員 ジョバンニ・パスカレッラ(Giovanni Pascarella)
チームリーダー ピエロ・カルニンチ(Piero Carninci)
原論文情報
- Alessandro Bonetti, Federico Agostini, Ana Maria Suzuki, Kosuke Hashimoto, Giovanni Pascarella, Juliette Gimenez, Leonie Roos, Alex J. Nash, Marco Ghilotti, Christopher J. F. Cameron, Matthew Valentine, Yulia A. Medvedeva, Shuhei Noguchi, Eneritz Agirre, Kaori Kashi, Samudyata, Joachim Luginbuuühl, Riccardo Cazzoli, Saumya Agrawal, Nicholas M. Luscombe, Mathieu Blanchette, Takeya Kasukawa, Michiel de Hoon, Erik Arner, Boris Lenhard, Charles Plessy, Gonçalo Castelo-Branco, Valerio Orlando & Piero Carninci, "RADICL-seq identifies general and cell type-specific principles of genome-wide RNA-chromatin interactions", Nature Communications, 10.1038/s41467-020-14337-6
発表者
理化学研究所
生命医科学研究センター トランスクリプトーム研究チーム
客員研究員 アレッサンドロ・ボネッティ(Alessandro Bonetti)
(現 カロリンスカ研究所 研究員)
チームリーダー ピエロ・カルニンチ(Piero Carninci)
 Piero Carninci
Piero Carninci
 Alessandro Bonetti
Alessandro Bonetti
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
