理化学研究所(理研)環境資源科学研究センター 天然物生合成研究ユニットのテイ・ウ 特別研究員(研究当時、現 基礎科学特別研究員)、高橋 俊二 ユニットリーダー、国立感染症研究所 品質保証・管理部の石川 淳 主任研究官らの共同研究グループは、タンパク質立体構造予測人工知能(AI)プログラムAlphaFold2[1]を用いて、放線菌[2]「Streptomyces sp. 80H647」から単離されたヌクレオシド系抗生物質[3]アスカマイシン(ascamycin)のアミノアシル化反応[4]を触媒するアラニルtRNA合成酵素[5]様の未知酵素の機能を予測し、生化学的に新たな生合成機構および自己耐性機構の解明に成功しました。
本研究成果は、新たな生理活性を持つ天然化合物の酵素化学的創出につながると期待されます。
今回、共同研究グループは、アスカマイシン生産菌に転写制御因子AdpA[6]およびtRNALeu (UUA) BldA[7]を追加導入し、アスカマイシンの生産量を改善しました。遺伝子発現解析とタンパク質の構造機能予測を行い、アラニルtRNA合成酵素様の二つの酵素(AcmFとAcmD)を同定しました。AcmFはアスカマイシン生合成中間体(デアラニルアスカマイシン)のアミノアシル化反応を効率的に触媒すること、またこの反応はAcmDから供給されるアラニルtRNA(Ala-tRNAAla)を基質として利用することを明らかにしました。さらに、共同研究グループはAcmD-AcmF酵素ペアを用いて、グリシル化およびセリル化アスカマイシン誘導体を創出しました。
本研究は、科学雑誌『ACS Catalysis』オンライン版(2月20日付)に掲載されました。
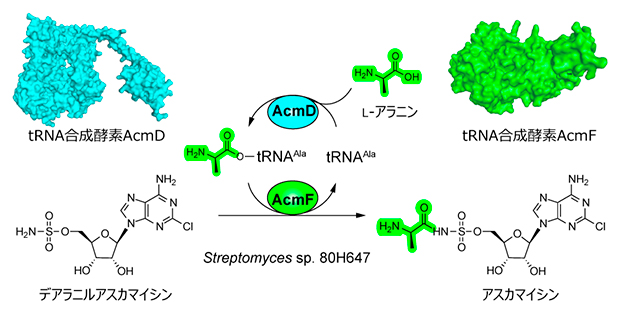
AcmDおよびAcmFによるヌクレオシド系抗生物質アスカマイシンのアミノアシル化反応
背景
ヌクレオシド系抗生物質は、細菌性の病原体やマラリア原虫のタンパク質合成を阻害する薬学的に重要な化合物の一種であり、アミノアシルtRNA合成酵素[4]や他のアデニル酸形成酵素の機能解明や結晶構造研究のために頻繁に化学合成されています。
アスカマイシンは、1984年に理研抗生物質研究室(当時)の磯野博士らにより放線菌「Streptomyces sp. 80H647」から単離されたユニークなヌクレオシド系抗生物質です注)。しかし、アスカマイシンの生産量は少なく、生合成に関わる酵素遺伝子および反応機構は長い間未解明でした。
- 注)Isono K, et.al. Ascamycin and dealanylascamycin, nucleoside antibiotics from Streptomyces sp., J. Antibiot. 37, 670-672 (1984)
研究手法と成果
共同研究グループは、アスカマイシンの生産を制御している可能性のあるadpA遺伝子とbldA遺伝子に着目しました。Streptomyces sp.80H647ゲノムから塩基配列情報を取得し、adpA遺伝子産物の転写制御因子AdpAおよびbldA遺伝子産物の転写制御因子tRNALeu (UUA)の構造を予測し、さらにadpAとbldAの遺伝子を生産菌に導入・発現させることによって、アスカマイシンの生産量を向上させました。
生合成反応に関与すると推定される遺伝子を特定するために、アスカマイシン生合成遺伝子クラスターの遺伝子発現解析を行いました。得られた候補酵素(AcmDとAcmF)のアミノ酸配列を基に、系統解析を行い、進化的関係を確認しました。AcmDと既知のアラニルtRNA合成酵素との間には密接な進化的関係が存在するのに対し、AcmFは進化の過程で分岐しており、新たな機能を持つ可能性が示唆されました。
また、AlphaFold2を用いてタンパク質構造を予測し、構造と機能に関する知見を獲得しました。AcmDはアラニルtRNA合成酵素の構造に似ていました。一方、AcmFはアミノアシル化ドメイン(領域)とtRNA認識ドメインのみで構成されていました。さらに、アミノアシル化ドメインの配列の比較により、AcmFにはATP(アデノシン三リン酸)結合に関与するアミノ酸残基のほとんどが欠如していました。分子ドッキング[8]の結果、AcmFは、AcmDに比べて基質を受け入れる活性部位が大きいことが分かりました。
これらの結果から、AcmFがアスカマイシンの生合成過程においてアミノアシル化反応を触媒し、AcmDがAla-tRNAAlaを供給している可能性が強く示唆されました。
予測した酵素の機能を検証するために、人工合成DNAを用いて発現ベクター(運び屋)を調製し、大腸菌を使って酵素を精製しました。精製酵素と基質を用いて反応を解析した結果、AcmD-AcmF酵素ペアがデアラニルアスカマイシンからアスカマイシンを効率的に生産することを検証できました。また、AcmDは、放線菌に普遍的に存在するアラニルtRNA合成酵素と比較して、アスカマイシンによる酵素阻害作用に耐性があることが判明しました。さらに、予測した構造から、AcmDのタンパク質構造中の活性部位が比較的狭いために阻害を受けにくいと推測しました(図)。
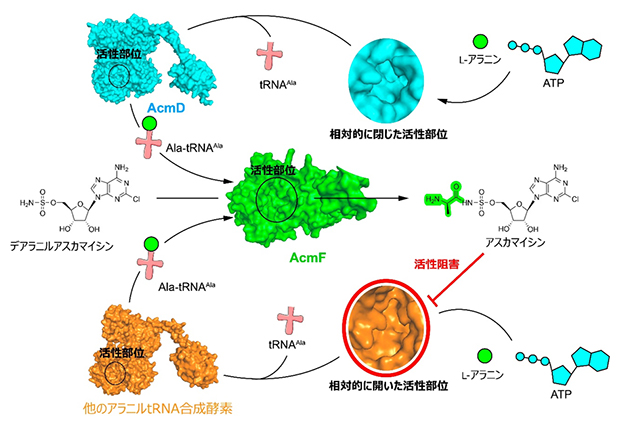
図 AcmD-AcmF酵素ペアはアスカマイシン生合成を効率化する触媒
AcmDはL-アラニンとATPからAla-tRNAAlaを生成し、Ala-tRNAAlaをAcmFの基質として供給する。AcmFはデアラニルアスカマイシンへアラニンを転移してアスカマイシンを生成する。AcmDは相対的に狭い活性部位を有するため、アスカマイシン阻害に対して自己耐性を示す。
AcmFの反応機構を理解するため、tRNAAla分子を試験管内(in vitro)で転写・精製し、L-アラニンを結合したAla-tRNAAla分子を基質として調製した後に、AcmF酵素反応を検証しました。次に、触媒活性を担う活性中心の必須アミノ酸領域を検証するために、部位特異的変異導入[9]した酵素を調製し、変異酵素反応を解析しました。その結果、Glu232などの重要な触媒アミノ酸残基を特定することに成功しました。
最後に、AcmD-AcmF酵素ペアの基質特異性を、L-アラニン以外の19種類のアミノ酸に対して調べた結果、アスカマイシンのグリシル誘導体とセリル誘導体が創製されていることを確認しました。このようなAcmD-AcmF酵素ペアによる天然化合物のアミノアシル化反応は、将来、新しいヌクレオシド系抗生物質を作るための重要な知見となります。
今後の期待
近年、AlphaFold2などのタンパク質立体構造予測AIプログラムの登場により、配列情報から生合成酵素の構造・機能を的確に予測できるようになり、合成生物学分野に大きなゲームチェンジが起きようとしています。本研究では、AIを活用して天然化合物の生合成酵素の機能を高い精度で予測し、単離から40年解明されていなかった新たな生合成機構と自己耐性機構を明らかにしました。酵素の機能を予知するとともに、生合成経路を自在に改変・拡張することによって、新たな生理活性化合物の生産ラインの開拓が期待されます。
本研究成果は、国際連合が定めた17項目の「持続可能な開発目標(SDGs)[10]」のうち「3.すべての人に健康と福祉を」に大きく貢献するものです。
補足説明
- 1.AlphaFold2
アミノ酸配列からタンパク質の立体構造を予測するAIプログラムであり、2020年11月にDeepMind社から発表された。さらに2021年7月には無償公開され、誰でも利用することが可能になった。 - 2.放線菌
土壌中など自然界に広く存在するグラム陽性の真正細菌であり、複雑な構造を持つ二次代謝産物を生産する。人類は、それらの中から、医薬、農薬、動物薬などの生理活性を持つ物質を利用してきた。現在も医薬探索源として重要視されている。 - 3.ヌクレオシド系抗生物質
核酸の構成成分である塩基と糖が結合した低分子化合物。タンパク質、核酸、細胞壁などの合成を阻害することによって抗菌活性、抗がん活性、抗ウイルス活性などの多様な生物活性を有する。 - 4.アミノアシル化反応、アミノアシルtRNA合成酵素
タンパク質を構成するアミノ酸は主に20種類存在する。20種類のアミノ酸のそれぞれに対してアミノアシルtRNA合成酵素が20種類存在し(アスパラギン酸に対応するAspRS、リシンに対応するLysRSなど)、アデノシン三リン酸(ATP)のエネルギーを利用してアミノ酸を活性化したのち、対応するtRNAのCCA末端に付加する反応。本研究では、Ala-tRNAAlaとデアラニルアスカマイシンからアスカマイシンを生成している。アミノアシルtRNA合成酵素は、タンパク質合成において、アミノ酸とそのアミノ酸に対応したアンチコドンを持つtRNAを結合させる、アミノアシルtRNAの生成反応を触媒する酵素。 - 5.アラニルtRNA合成酵素
アラニン(Ala)を対応するtRNAに結合させAla-tRNAAlaを合成する酵素。 - 6.AdpA
放線菌の一種であるStreptomyces属に存在する転写制御因子。特定のDNA配列に結合することによって、標的遺伝子の転写が制御され、抗生物質生産が誘導される。 - 7.BldA
Streptomyces属の放線菌において重要な転写制御因子の一つ。この制御因子は、特定のtRNALeu (UUA)を介して働き、遺伝子の転写を制御する。 - 8.分子ドッキング
薬品やタンパク質などの生体分子がどのように相互作用するかを予測するコンピュータシミュレーションのこと。 - 9.部位特異的変異導入
タンパク質を構成する特定のアミノ酸の改変を目的として、酵素遺伝子に変異を導入する実験手法。 - 10.持続可能な開発目標(SDGs)
2015年9月の国連サミットで採択された、持続可能でよりよい世界を目指す国際目標。17のゴールおよび169のターゲットで構成されており、国連加盟193か国が、2016年から2030年の15年間でこれらを達成することを目標としている。
共同研究グループ
理化学研究所 環境資源科学研究センター
天然物生合成研究ユニット
ユニットリーダー 高橋 俊二(タカハシ・シュンジ)
特別研究員(研究当時)テイ・ウ(ZHENG Yu)
(現 基礎科学特別研究員)
テクニカルスタッフⅠ 森田 直子(モリタ・ナオコ)
テクニカルスタッフⅠ 髙木 海(タカギ・ヒロシ)
テクニカルスタッフⅠ(研究当時)佐藤 裕美(サトウ・ユミ)
(現 化合物リソース開発研究ユニット テクニカルスタッフⅡ)
国立感染症研究所 品質保証・管理部
主任研究官 石川 淳(イシカワ・ジュン)
産業技術総合研究所 生命工学領域
総括研究主幹 新家 一男(シンヤ・カズオ)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業基盤研究(A)「天然化合物の多様性拡張を志向した生合成分子基盤の解明(研究代表者:高橋俊二)」、同学術変革領域研究(A)「AIを活用した未知の二次代謝生合成酵素の機能解明と分子間相互作用の精密解析(研究代表者:高橋俊二)」の助成を受けて行われました。
原論文情報
- Yu Zheng, Naoko Morita, Hiroshi Takagi, Yumi Shiozaki-Sato, Jun Ishikawa, Kazuo Shin-ya, Shunji Takahashi, "Alanyl-tRNA Synthetase-like Enzyme-Catalyzed Aminoacylation in Nucleoside Sulfamate Ascamycin Biosynthesis", ACS Catalysis, 10.1021/acscatal.3c05667
発表者
理化学研究所
環境資源科学研究センター 天然物生合成研究ユニット
ユニットリーダー 高橋 俊二(タカハシ・シュンジ)
特別研究員(研究当時)テイ・ウ(ZHENG Yu)
(現 基礎科学特別研究員)
国立感染症研究所 品質保証・管理部
主任研究官 石川 淳(イシカワ・ジュン)
 後列左から)髙木 海、高橋 俊二、テイ・ウ
後列左から)髙木 海、高橋 俊二、テイ・ウ(前列左から)森田 直子、佐藤 裕美
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
国立感染症研究所
Tel: 03-5285-1111
Email: info [at] nih.go.jp
※[at]は@に置き換えてください。
