2009年1月27日
理化学研究所
不良mRNAの品質管理機構が、mRNA様ノンコーディングRNAを抑制
-mRNA様ノンコーディングRNAの発現制御を網羅的に初めて解析-
ポイント
- 新たに97種のmRNA様ノンコーディングRNAを発見
- 抑制されたmRNA様ノンコーディングRNAの7~8割がアンチセンスRNA
- 未知のmRNA様ノンコーディングRNA機能解明に貢献
要旨
独立行政法人理化学研究所(野依良治理事長)は、シロイヌナズナ※1のさまざまな遺伝子のRNA発現量を検出するタイリングアレイ※2を用いて、不良mRNAを分解する機構「nonsense-mediated mRNA decay (NMD)」が、タンパク質をコードしないRNAであるmRNA様ノンコーディングRNA(mlncRNA)※3の多くを抑制していると、初めて明らかにしました。これは、理研植物科学研究センター(篠崎一雄センター長)植物ゲノム発現研究チームの関原明チームリーダー、栗原志夫特別研究員、生命情報基盤研究部門(豊田哲郎部門長)らによる研究成果です。
近年、ヒトをはじめとする動植物など真核生物のゲノムから、無数のmlncRNAが転写されていることが明らかになってきました。mlncRNAは、何らかのタンパク質をコードする遺伝子(センス)の逆鎖から転写されるアンチセンスRNA※4と、それ以外のものに分けられます。研究チームは、シロイヌナズナのmlncRNAの発現制御機構に関して分子レベルの解析を行ってきました。その過程で、mlncRNAの多くが、不良mRNAを分解するNMD機構の標的分子になりやすい構造をしていることに気づき、NMD機構によってmlncRNAが恒常的に抑制される可能性が高いと考えました。
そこで、NMD機構が働くための必須遺伝子であるUPF1、UPF3遺伝子※5の機能ノックダウン変異体※6upf1-1、upf3-1におけるトランスクリプトーム※7を、全ゲノムのRNA発現量を網羅的に検出するタイリングアレイを用いて調べました。得たデータを、スプライシング※8開始点を予測できるARTADE法※9により解析した結果、NMDによって抑制される候補mlncRNAとして36種の既知mlncRNAと97種の新規mlncRNAを同定しました。さらに、同定したmlncRNAのおよそ7~8割がアンチセンスRNAに分類されることが分かりました。
本研究は、タイリングアレイを用いてNMD機構によるmlncRNAの抑制を初めて網羅的に示したものです。しかし現在まで、植物だけでなく動物も含めて、mlncRNAの役割について明らかになっている例はほとんどありません。この成果が、mlncRNAの役割・機能の理解に大きく寄与することは間違いありません。
本研究成果は、米科学アカデミーの学術誌『Proceedings of the National Academy of Sciences of the United States of America』に1月26日の週に掲載されます。
背景
真核生物のタンパク質は、DNAの塩基情報をmRNAに写し取り、タンパク質の工場となっているリポソームでタンパク質に翻訳されることで合成されます。この時、点変異あるいは転写やスプライシング過程での間違いを引き起こし、mRNA上で、本来の終止コドンの上流に予期せぬ未成熟終止コドン(PTC)※10が現れてしまうことがあります。PTCを持つmRNAからC末端が欠失したタンパク質が作られてしまうと、生体に影響を与えかねません。そこで、真核生物には、そのようなPTCを持つ不良mRNA(または異常と判別されたmRNA)を選択的に分解するmRNA監視機構が備わっており、「nonsense-mediated mRNA decay(NMD)機構」と呼ばれています(図1)。このNMD機構がPTCを認識し、正常な機能を果たすための必須因子として、UPF1、UPF2、UPF3という3つのタンパク質からなるUPFタンパク複合体※11が知られています。UPFタンパク複合体は、mRNA上で形成され、そのmRNAの終止コドンの位置を認識すると考えられています。植物の場合、DNA情報が写し取られたmRNAに、およそ300塩基対以上の異常に長い3´ UTR(3´側のUnTranslated Region:非翻訳領域)が存在する場合、あるいは最終エクソン※8結合部から50塩基対以上上流に終止コドンが位置する場合に、UPFタンパク複合体によって不良mRNAとして認識され、分解されやすいことが示されています(図2)。
近年、動物、植物の両方において、数多くのmRNA様ノンコーディングRNA(mlncRNA)がゲノムから転写されていることが分かってきました。しかし、mlncRNAの細胞内での発現制御機構やその役割についての知見は、ほとんど得られていません。そのため、mlncRNAについての研究は、世界中で精力的に進められています。
研究手法と成果
ほとんどのmlncRNAには、短い翻訳可能領域(翻訳されているかどうかは未知)が存在します。シロイヌナズナの既知のmlncRNAの約87%は、300塩基対以上の3´ UTRを持ちます。さらに、タンパク質の設計に関与しないとされるイントロン※8を有するmlncRNAの約40%は、最終エクソン結合部から50塩基対以上上流に終止コドンが位置しています。このため、mlncRNAの多くが、NMD機構の標的分子になりやすい構造を保っていることになります(図2)。そこで、mlncRNAがNMD機構によって本当に抑制されているかどうかを調べるために、NMD機構が働くための必須遺伝子であるUPF1、UPF3の機能ノックダウン変異体upf1-1、upf3-1におけるトランスクリプトームを、全ゲノムタイリングアレイを用いて解析をしました。実験には、プレート培地で15日間生育させたシロイヌナズナを用いました。
- 1.各upf変異体で蓄積が増加している既知mlncRNAを同定
ランダムに変異を起こした変異体からupf1-1変異体、upf3-1変異体を選び、野生型のシロイヌナズナを比較すると、それぞれ237個、167個の既知遺伝子の転写産物が増加していました。それらのうち、31個、25個が既知mlncRNA(全部で36種類)、198個、138個がタンパク質をコードするmRNAでした(図3A)。蓄積が増加したタンパク質をコードするmRNAは、それぞれ、発現しているmRNAの中の1.2%、0.9%であったのに対して、蓄積が増加したmlncRNAは、それぞれ、発現しているmlncRNAの中の20.9%、16.8%でした。つまり、mlncRNAは、タンパク質をコードするmRNAよりも、NMD機構の標的になりやすいことが示唆されました。
また、upf1-1変異体、upf3-1変異体で蓄積が増加した31個と25個の既知mlncRNAのうち、それぞれ25個と20個のmlncRNAが、アンチセンスRNAであることがわかりました(図3B)。 - 2.各upf変異体で蓄積が増加している新規mlncRNAを同定
ARTADE法を用いて新規mlncRNA候補を予測したところ、野生型のシロイヌナズナ、upf1-1変異体およびupf3-1変異体でそれぞれ1,752個、1,707個、1,894個の新規mlncRNAが予測できました。その中から、upf1-1変異体、upf3-1-変異体において、それぞれ77個と59個の新規mlncRNA(全部で97種類)が、野生型と比較すると蓄積が増加していました。つまり、既知のmlncRNAに加えて、さらに多くの新規mlncRNAがNMD機構によって抑制されていることを示しています。また、upf1-1変異体、upf3-1変異体で蓄積が増加した77個と59個の新規mlncRNAのうち、それぞれ51個と42個のmlncRNAがアンチセンスRNAであることがわかりました(図4)。既知mlncRNAとあわせると、mlncRNAの約7~8割がアンチセンスRNAに分類されることになります。
このタイリングアレイデータの情報は、理研オミックブラウズのホームページで、公開しています。
今後の期待
本研究は、これまで未知であったmlncRNAの発現制御機構の一部を明らかにしたことになります。現在、mlncRNAの生体における役割や機能については、ほとんど明らかにされておらず、mlncRNAの生物学的意義の解明に貢献すると考えられます。
研究では植物のシロイヌナズナを用いて解析しましたが、このNMD機構は真核生物で高度に保存された機構で、UPF1、UPF2、UPF3のいずれか1つの機能を完全になくした場合、動植物とも死にいたります。従って、動物においてもmlncRNAがNMD機構によって抑制されていることが推測できます。今回得た成果は、ヒトをはじめとする動物でのメカニズムの解明に結びつくと期待されます。
発表者
理化学研究所
植物科学研究センター 植物ゲノム発現研究チーム
チームリーダー 関 原明(せき もとあき)
特別研究員 栗原 志夫(くりはら ゆきお)
Tel : 045-503-9587 / Fax : 045-503-9584
お問い合わせ先
横浜研究推進部 企画課Tel : 045-503-9117 / Fax : 045-503-9113
報道担当
理化学研究所 広報室 報道担当Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.シロイヌナズナ
学名は Arabidopsis thaliana (L.) Heynh. アブラナ科シロイヌナズナ属の一年草。モデル植物として植物で初めてゲノム解読が行われ、ゲノムサイズは約1.3億塩基対、全遺伝子数は約30,000個である。 - 2.タイリングアレイ
生物のゲノムDNA配列のほとんどを網羅するオリゴヌクレオチドにより構成される高密度マイクロアレイ。通常用いられているマイクロアレイは、スライドガラス上に1遺伝子あたり数箇所のプローブごとに発現量を計測し、それを平均化することで、各遺伝子のRNA発現量を検出する方法だが、タイリングアレイを用いて解析を行えば、遺伝子の存在が予測されていない領域に存在する新規の転写産物も同定することができる。 - 3.mRNA様ノンコーディングRNA(mlncRNA)
mRNAと同様に、RNAポリメラーゼIIにより転写されるが、タンパク質をコードしないRNA。真核生物において多数のmlncRNAが発見されているが、その役割や機能はほとんどわかっていない。 - 4.アンチセンスRNA
遺伝情報としてタンパク質などを合成する配列の方向性をセンスとする。センス配列に対して相補的で逆の方向性をアンチセンスという。そのセンス鎖から転写されるRNAをアンチセンスRNAと呼ぶ。 - 5.UPF1、 UPF3遺伝子
UP FrameshiftというNMD機構に必須の遺伝子。mRNA上にUPFタンパク複合体を形成し、不良mRNAを認識する。 - 6.ノックダウン変異体
ノックアウト変異体では遺伝子そのものが破壊され、機能を持たないのに対して、遺伝子の機能が大きく減衰しているが、完全になくなっていない場合にノックダウン変異体と呼ぶ。 - 7.トランスクリプトーム
ある生物種のゲノムDNAから転写されたRNAの総体を指す。 - 8.スプライシング、イントロン、エクソン
真核生物において、mRNA前駆体上には、タンパク質をコードする配列を含むエクソン領域とコードしない配列のイントロン領域が存在する。スプライシングとは、このイントロンを除き、残りのエクソン部分を結合して完全なmRNAを作ることをいう。 - 9.ARTADE法
Arabidopsis Tiling Array-based Detection of Exons法のこと。タイリングアレイを用いた発現データを用いて、構造未知遺伝子についてエクソン・イントロン構造を推定する方法。アレイデータと塩基配列データを、統計モデルを用いて統合的に解析することで、スプライシングの起こるポイントを正確に予測する。全ゲノムにわたり高い精度で、網羅的に遺伝子構造を推定することが可能な、世界初のバイオインフォマティクス技術。
(参考)Toyoda and Shinozaki (2005) Plant J. 43: 611-621. - 10.未成熟終止コドン(PTC)
タンパク質をコードするmRNA上に点変異あるいは転写やスプライシング過程での間違いにより、本来存在しないはずの終止コドンが出現する。このような終止コドンを未成熟終止コドンと呼ぶ。 - 11.UPFタンパク複合体
UPF1、UPF2、UPF3の3つのタンパク質からなる複合体。mRNA上に形成され、未成熟終止コドン(PTC)を持つ不良mRNAを分解に導く。
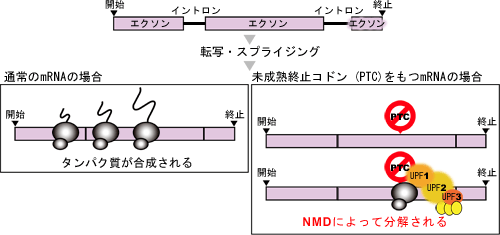
図1 nonsense-mediated mRNA decay (NMD) 機構 のモデル図
通常のmRNAからは、タンパク質が合成(翻訳)される。点変異あるいは転写やスプライシング過程での間違いによる未成熟終止コドン(PTC)を持つ不良mRNAは、UPFタンパク複合体によって認識され、分解される。
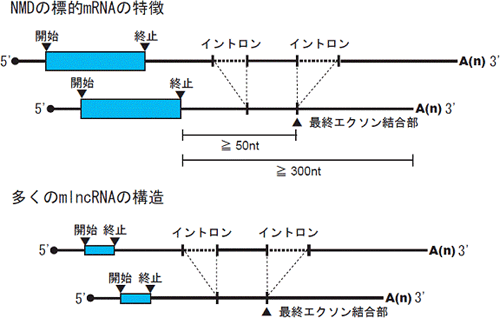
図2 NMD機構の標的になりやすいmRNAおよびmRNA様ノンコーディングRNA(mlncRNA)の模式図
一般的な植物の場合、およそ300塩基対以上の異常に長い3´UTRを持つmRNAや、最終エクソン結合部から50塩基対以上上流に終止コドンが位置するmRNAが、分解されやすいと示されている。多くのmlncRNAも、同様にNMD機構の標的になりやすい構造をとっている。
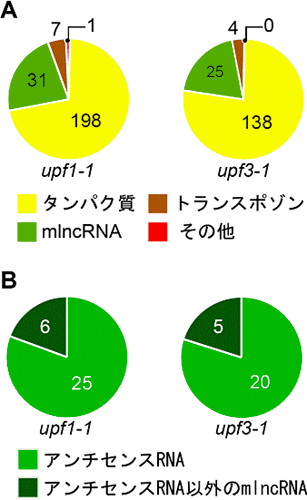
図3 upf1、upf3変異体で蓄積が増加した既知転写産物の分類
A:各変異体で蓄積が増加した既知転写産物を、タンパク質をコードするもの、トランスポゾンをコードするもの、mlncRNA、そのほかの転写産物の4種類に分類
B:各変異体で蓄積が増加したmlncRNAを、アンチセンスRNAとアンチセンスRNA以外のmlncRNAに分類。およそ8割がアンチセンスRNAであった。

図4 upf1、upf3変異体で蓄積が増加した新規転写産物の分類
蓄積が増加した新規転写産物をアンチセンスRNAとアンチセンスRNA以外のmlncRNAに分類。およそ7割がアンチセンスRNAであった。
