ポイント
- 人工染色体を用いた独創的な解析方法を開発
- ポリコム複合体は、異性型PRC1、PRC2、従来型PRC1の順でDNAに結合
- 正常な骨の形態形成には異性型PRC1が重要
要旨
理化学研究所(理研、野依良治理事長)は、遺伝子発現の抑制をつかさどるポリコム複合体[1]が、従来考えられていた順序とは異なる順序でDNAに結合することを発見しました。これは、理研統合生命医科学研究センター(小安重夫センター長代行)免疫器官形成グループの古関明彦グループディレクター、近藤隆研究生と、英国オックスフォード大学との共同研究グループによる成果です。
細胞が未分化の状態から特定の細胞へと分化する運命決定には、さまざまな遺伝子の発現(オン)・抑制(オフ)といった遺伝子発現の切り換えが深く関わっています。この遺伝子発現の切り換えを管理しているのがポリコムタンパク質群[2]です。ポリコムタンパク質群は、PRC1[1]、PRC2[1]という2種類のポリコム複合体を形成し、この複合体がDNAに結合することで、遺伝子の発現を抑制しています。また、最近、PRC1には、従来型PRC1の他に異性型PRC1[3]の存在が明らかになりましたが、異性型PRC1の機能は分からないままでした。
共同研究グループは、人工染色体(BAC)[4]を用いた新しい解析手法を開発し、ポリコムタンパク質がどのように複合体を形成し、遺伝子発現を制御するのかを詳細に調べました。その結果、ポリコム複合体のDNAへの結合の順序は、従来の概念とはまったく異なることが分かりました。これまでは、最初にPRC2がDNAに結合し、PRC1を呼び寄せ、遺伝子発現を抑制すると考えられてきました。しかし、最初に異性型PRC1がDNAに結合し、PRC2、従来型PRC1という順番で呼び寄せ、遺伝子発現を抑制していることが明らかとなりました。
遺伝子の発現調節メカニズムの解明は、形態形成だけでなく、細胞の正常な分化・脱分化の過程の解明、ES細胞(胚性幹細胞)やiPS細胞(人工多能性幹細胞)の分化のコントロールなど、発生に関わるさまざまな面で重要です。今後、さらにポリコム複合体による遺伝子発現のオン・オフの切り換えの核心に迫ることで、将来的には再生医療やがん治療に応用できると期待されます。
本研究成果は、米国の科学雑誌『Cell』(6月5日号)に掲載されるに先立ち、オンライン版(5月22日付け:日本時間5月23日)に掲載されます。
背景
細胞の運命は、幹細胞など多能性を維持した未分化細胞の時期に決定されます。その時期に、運命決定に関わるさまざまな遺伝子の発現(オン)・抑制(オフ)が切り換わります。この遺伝子発現のオン・オフの切り換えを管理しているのがポリコムタンパク質群です。
ポリコムタンパク質群は、その標的となるDNAの特定の部分で巨大な複合体であるポリコム複合体を形成し、標的遺伝子の発現を抑制します。これまで、ポリコム複合体は、PRC1、PRC2という2種類が存在するとされてきました。ところが最近、PRC1には、従来型PRC1の他に、異性型PRC1の存在が明らかになりました(図1)。しかし、異性型PRC1の機能は分からないままで、真にポリコムと呼べるかどうかも不明でした。そこで、共同研究グループは、ポリコムタンパク質がどのようにDNAに結合して複合体を形成し、遺伝子発現を制御するのかを詳細に調べることにしました。
研究手法と成果
共同研究グループは、ポリコムタンパク質群がどのようにDNAに結合していくかを調べるために、人工染色体(BAC)を用いた独創的な方法を開発しました(図2)。この方法は、特定のポリコムタンパク質だけをDNAに結合させて、その機能を培養ES細胞で解析することができます。
この方法を使って解析した結果、ポリコムタンパク質群のDNAへの結合の順序は、最初にPRC2がDNAに結合し、PRC1を呼び寄せるという従来の概念と異なっていることが分かりました。
まず、従来型PRC1を形成するポリコムタンパク質(PCGF2、PCGF4など)を最初にDNAに結合させたところ、予想に反して、従来型PRC1がDNAに結合してもPRC1特有の酵素活性(ヒストンH2Aの119番目のLysのユビキチン化[5])は観察されず、PRC2や異性型PRC1を呼び寄せないことが分かりました。次に、異性型PRC1を形成するポリコムタンパク質(PCGF1、KDM2Bなど)を最初にDNAに結合させたところ、ヒストンH2Aのユビキチン化活性が観察され、PRC2、従来型PRC1を呼び寄せることが分かりました。最後に、PRC2を形成するポリコムタンパク質を最初にDNAに結合させたところ、従来型PRC1を呼び寄せましたが、異性型PRC1は呼び寄せないことが分かりました。
これらの結果から、ポリコムタンパク質群のDNAへの結合の順序は、従来考えられていた順序ではなく、最初に異性型PRC1がDNAに結合して、PRC2、従来型PRC1という順番で呼び寄せ、遺伝子発現を抑制していることが分かりました(図3)。
また、共同研究グループは、異性型PRC1の機能を確認するため、異性型PRC1のDNA結合領域を抗がん剤の一種のタモキシフェンで欠失させ、異性型PRC1がDNAに結合できないようにしました。その結果、全てのポリコム複合体が結合できず、遺伝子発現の抑制ができなくなることが分かりました。
さらに、生体での異性型PRC1の機能を確認するため、異性型PRC1の遺伝子結合領域を欠失したマウスを作製しました。完全に遺伝子を欠失させたマウス(ホモの遺伝子ノックアウト)では、胚発生がうまくいかず、異性型PRC1が発生に必須であることが明らかとなりました。一方、異性型PRC1の遺伝子結合領域を半分だけ欠失させたマウス(ヘテロの遺伝子ノックアウト)は、胚発生はできるものの生後10日までに80%が死亡してしまいました。また、生き残ったマウスの骨格を調べたところ、頸骨と肋骨の境界が移動して首の頸骨が肋骨化したり、仙骨が前に移動して腹椎が減って腰椎が増えたりといった、骨の形態形成に異常が起きていました。これらの異常は、典型的なポリコムタンパク質欠失の表現型であり(図4)、異性型PRC1はポリコムの一員であることが明確になりました。以上のことから、正常な骨の形態形成には、ポリコム複合体の中でも異性型PRC1が重要であることが分かりました。
今後の期待
遺伝子の発現調節メカニズムの解明は、形態形成だけでなく、細胞の正常な分化・脱分化の過程の解明、ES細胞(胚性幹細胞)やiPS細胞(人工多能性幹細胞)の分化のコントロールなど、発生に関わるさまざまな面で重要です。
今後、さらにポリコム複合体による遺伝子発現制御の詳細な仕組みを明らかにし、遺伝子発現のオン・オフの切り換えの核心に迫ることで、将来的には再生医療やがん治療に応用が可能になるものと期待されます。
原論文情報
- Neil P Blackledge, Anca M Farcas, Takashi Kondo, Hamish W King, Joanna F McGouran, Lars LP Hanssen, Shinsuki Ito, Sarah Cooper, Kaori Kondo, Yoko Koseki, Tomoyuki Ishikura, Hannah K Long, Thomas W Sheahan, Neil Brockdorff, Benedikt M Kessler, Haruhiko Koseki, Robert J Klose,
"Variant PRC1 complex dependent H2A ubiquitylation drives PRC2 recruitment and polycomb domain formation" Cell,2014, doi: 10.1016/j.cell.2014.05.004
発表者
理化学研究所
統合生命医科学研究センター 免疫器官形成研究グループ
研究生 近藤 隆(こんどう たかし)
お問い合わせ先
統合生命医科学研究推進室
Tel: 045-503-9117 / Fax: 045-503-9113
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.ポリコム複合体、PRC1、PRC2
ポリコム複合体は、ポリコムタンパク質群の機能単位であり、複数のポリコムタンパク質が集合したもの。PRC1はCBX、PCGF2またはPCGF4、RING1、PHCを含み、巨大なポリコムボディと呼ばれるポリコムタンパク質群とDNAからなる巨大な複合体を形成する。PRC2はEED、EZH、SUZ12などを含みヒストンH3の27番目のアミノ酸残基であるリジンのメチル化を行い、H3K27me3を生成する。 - 2.ポリコムタンパク質群
ショウジョウバエで見いだされた一群のクロマチン因子。ホメオティック遺伝子群の調節因子として発見されたが、後にDNAが巻き付いているタンパク質「ヒストン」のメチル化、ユビキチン化の活性を有することが分かり、遺伝子の発現を抑制することが知られている。 - 3.異性型PRC1
BCoRとして知られていた抑制性クロマチン因子の複合体。PCGF1/3またはPCGF5、RING1、RYBP、KDM2Bなどを含む。本研究において元来ヒストンH2Aの119番目のアミノ酸残基であるリジンのユビキチン化はこれまで知られていたPRC1によるものではなく、異性型PRC1で行われることが強く示唆された。 - 4.人工染色体(BAC)
BACはbacterial artificial chromosomeの略。大腸菌の複製開始点を持ち、この技術により約200kbの大きさを持つDNA断片のクローニングが可能になった。本研究ではヒト由来のポリコム結合領域を持たない染色体領域をBACを用いてクローニングし、その中央に大腸菌由来のDNA結合タンパク質であるTetRの結合配列TetOを挿入したDNAを用いている。 - 5.ユビキチン化
ユビキチンは76個のアミノ酸からなる小さなタンパク質。このユビキチンが標的タンパク質に付加することをユビキチン化といい、タンパク質の翻訳後修飾の1つである。タンパク質のユビキチン化には、それぞれのタンパク質を特異的に認識するユビキチン化酵素が働いており、細胞周期特異的に機能するものも多い。
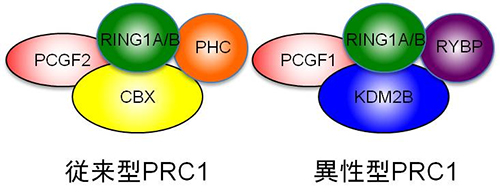
図1 従来型PRC1と異性型PRC1の複合体模式図
従来型PRC1はPCGF2、CBXなどのポリコムタンパク質で構成される。一方、異性型PRC1はPCGF1、KDM2Bなどで構成される。今回の実験により、異性型PRC1は骨の形態形成に重要な役割を果たすことが分かった。
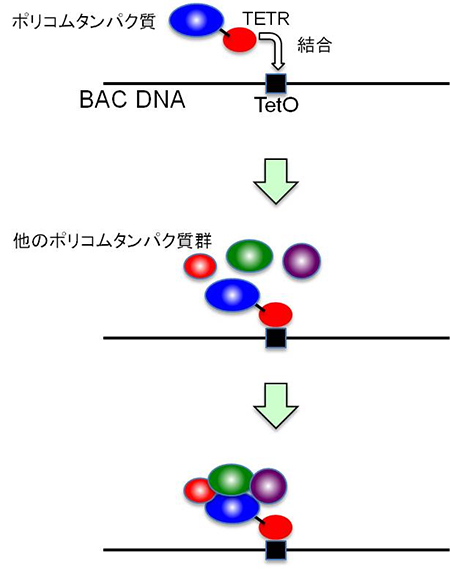
図2 今回開発したBACを用いた実験スキーム
今回の実験では、遺伝子もポリコムタンパク質の結合配列も含まれていない、ヒト由来の170kbに及ぶDNAを持つBACを用いる。このBACに、TetO、TETRというDNA結合タンパク質の結合配列をそれぞれ挿入し、ES細胞に導入する。そのES細胞内でTETRと特定のポリコムタンパク質を融合させた融合タンパク質を発現させることで、特定のポリコムタンパク質だけをDNAに結合させて、その機能を解析することができる。
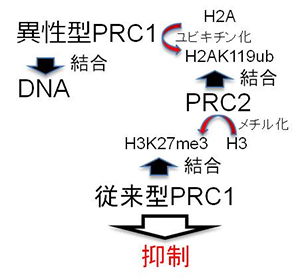
図3 ポリコム複合体がDNAを認識する順番
最初に異性型PRC1がDNAに結合し、H2Aをユビキチン化する。それをPRC2が認識しH3をメチル化し、それに従来型PRC1が結合し、遺伝子発現を抑制する。
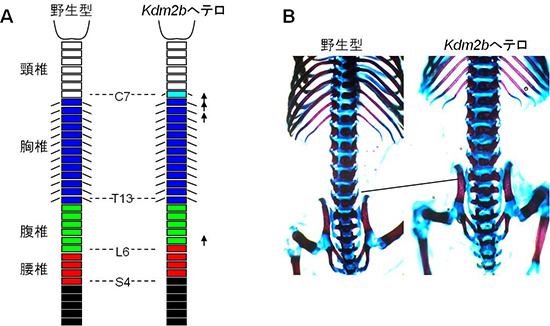
図4 異性型PRC1の遺伝子結合領域を欠損させたマウスの脊椎骨
- A.脊椎骨の表現型のスキーム
一般に野生型マウスの脊椎骨は、頸椎7、胸椎13、腹椎6、腰椎4という順で形成される。一方、異性型PRC1の遺伝子結合領域(Kdm2b)を半分だけ欠失させた、Kdm2bヘテロマウスは、全ての個体で、頸椎、胸椎、あるいは腰椎の前方へのシフトが観察される。これは典型的なポリコム遺伝子の変異による表現型を示している。 - B.骨パターンの一例
野生型では腹椎になる26番目の脊椎骨が腰椎の形状を持っており(図中黒線部分)、Kdm2bが生体内においてもポリコムタンパク質群の一員として機能していることを示している。
