要旨
理化学研究所(理研)ライフサイエンス技術基盤研究センター トランスクリプトーム研究チームのピエロ・カルニンチ チームリーダー(副センター長)、アレクサンダー・フォート客員研究員と、理研統合生命医科学研究センター免疫器官形成研究グループの古関明彦グループディレクターらの研究グループは、ES細胞(胚性幹細胞)とiPS細胞(人工多能性幹細胞)で発現する全てのRNAを比較解析し、ES細胞で発現しているノンコーディングRNA(ncRNA)[1]の多くが、iPS細胞では十分に発現していないことを発見しました。
iPS細胞の作製法が2006年に報告されて以来、分化した体細胞を効率良くリプログラミング(初期化)し、ES細胞と同等の性質を持つ高品質のiPS細胞を安定して得るための試みが続けられています。体細胞に由来するiPS細胞と受精卵に由来するES細胞は、幹細胞としての性質の多くが共通しています。しかし遺伝子発現の解析からは、両者で発現する遺伝子には違いがあり、iPS細胞はES細胞と同一の状態ではないことが指摘されています。ただし、これまでの解析は主にタンパク質の設計図となるメッセンジャーRNA (mRNA)について行われており、設計図として用いられないncRNAの詳細な比較は行われていませんでした。
研究グループは、マウス由来のES細胞とiPS細胞を用い、ncRNAを含めた全転写産物(全RNA)の網羅的な発現比較を行いました。その結果、ES細胞の核内で発現するncRNAの多くが、iPS細胞では十分に発現していないことが分かりました。これらのncRNAの中には、多能性の維持に関わる遺伝子の発現を促進する遺伝子制御部位(エンハンサー[2])や、多能性に関わるレトロトランスポゾン[3]由来のRNA配列が含まれていました。これは、既存のiPS細胞作製法では、ES細胞で機能している多くの遺伝子制御部位の活性化が十分に起きていないことを示しています。ncRNAを含む全てのRNAを対象とするトランスクリプトーム[4]解析はiPS細胞の状態を正確に捉えるために重要であり、今後、臨床応用に用いるiPS細胞の評価法としても役立つと期待できます。
本研究は、日本学術振興会「最先端・次世代研究開発支援プログラム」などの支援を受けて行われ、成果は米国の科学雑誌『Cell Cycle』オンライン版(2月12日付け:日本時間2月13日)に掲載されました。
※研究グループ
理化学研究所
ライフサイエンス技術基盤研究センター トランスクリプトーム研究チーム
チームリーダー Piero Carninci (ピエロ・カルニンチ)(副センター長)
客員研究員 Alexandre Fort (アレクサンダー・フォート)
研究員 橋本 浩介(はしもと こうすけ)
統合生命医科学研究センター 免疫器官形成研究グループ
グループディレクター 古関 明彦(こせき はるひこ)
研究員 山田 大輔(やまだ だいすけ)
背景
分化した体細胞を、ES細胞(胚性幹細胞)に近い多能性を持った状態に変換するiPS細胞(人工多能性幹細胞)の作製法が2006年に報告されました。iPS細胞は創薬・再生医療への応用が進められており、近年の細胞生物学における大きな成果です。一方、それぞれの細胞で発現している遺伝子を解析した研究から、iPS細胞はES細胞とは異なる状態にある可能性が報告されています。iPS細胞とES細胞の違いを正確に理解することは、iPS細胞作製技術の改良や、臨床応用に適したiPS細胞の評価選別法の確立にとって重要な意味があります。
これまで、ES細胞とiPS細胞では多くの遺伝子が異なる発現様式を示しているという報告がある一方、特定のiPS細胞はES細胞とほぼ区別がつかないという報告もあります。ただし、これらの解析は主にタンパク質の設計図となるメッセンジャーRNA (mRNA)や既知のノンコーディングRNA(ncRNA)だけを解析したもので、ES細胞とiPS細胞のncRNAを網羅的に解析する研究は行われていませんでした。研究グループは昨年、転写産物(RNA)の網羅的な解析を定量的かつ高感度に行うことができる理研の独自技術「CAGE法[5]」を用い、iPS細胞とES細胞の核内には、これまで知られていなかった数千種類のRNA(幹細胞特異的な転写産物:NASTs[6])が発現していることを見いだし、その多くがレトロトランスポゾンに由来するncRNAであることを明らかにしました注)。本研究では、同様の解析手法を用い、ES細胞とiPS細胞で異なる発現を示すRNAの解析を試みました。
注)2014年4月29日プレス発表「幹細胞の多能性に関わるレトロトランスポゾン由来のRNA」
研究手法と成果
実験では、マウスES細胞と、マウスリンパ球から樹立したiPS細胞を用い、 核と細胞質から別々に調製した全RNAをCAGE法で解析しました。ES細胞とiPS細胞で発現に差があるRNAを調べたところ、核内で4,526種、細胞質で1,488種、核と細胞質に共通するRNAでは1,364種が見つかりました(図1)。核で差が見られたRNAの大半は、ES細胞に比べてiPS細胞での転写量が少ないか、転写が見られませんでした。この結果は、ES細胞の核はiPS細胞の核に比べ多様なRNAを含んでいること、および、通常のiPS細胞作製法による細胞のリプログラミング(初期化)では、これらの転写誘導が十分に起きていないことを示しています。
iPS細胞では適切に誘導されていないES細胞核特異的なRNAは、その多くが、既知の塩基配列や機能との関連が不明な未知のRNAでした。そこで、ENCODE[7]プロジェクトで得られたヒストン修飾[8]のデータなどを用いて、これらのRNAの機能を予測したところ、エンハンサーやスーパーエンハンサー[9]など遺伝子発現制御に関わるncRNAが多く含まれることが示唆されました。またスーパーエンハンサーの約6割は、昨年の研究で同定したNASTsと配列が一致していました。
次に、これらのエンハンサーやスーパーエンハンサーが発現を制御している可能性のある約200遺伝子の発現を、ES細胞とiPS細胞で比較しました。その結果、ES細胞での発現量がiPS細胞の2倍以上となっている遺伝子が多く、エンハンサー量の違いが遺伝子発現に大きな影響を及ぼしていることが確認できました。発現量の差の大きい遺伝子には、多能性関連遺伝子(Klf2、Cited2)とともに、機能未知のロングノンコーディングRNA(lncRNA)[10]が含まれており、lncRNAがスーパーエンハンサーの制御を通して多能性に関与していることも強く示唆されました(図2)。
今後の期待
今回、ES細胞とiPS細胞の間には、ncRNAの転写量や種類に大きな違いがあることが分かりました。これは、ES細胞では活性化されているエンハンサーなどの遺伝子制御部位が、iPS細胞では適切に誘導されていないことを示します。このため幹細胞性の制御に関与する遺伝子やlncRNAの発現が十分に起こらず、iPS細胞がES細胞と同一の性質を持つに至っていない理由の1つと考えられます。iPS細胞で発現するncRNAの重要性を示した今回の成果は、さまざまなiPS細胞を適切に評価する方法の開発やiPS細胞作製技術の改良への貢献が期待できます。
原論文情報
- Alexandre Fort, Daisuke Yamada, Kosuke Hashimoto, Haruhiko Koseki & Piero Carninci, "Nuclear transcriptome profiling of induced pluripotent stem cells and embryonic stem cells identify non-coding loci resistant to reprogramming.", Cell Cycle, doi: 10.4161/15384101.2014.988031
発表者
理化学研究所
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 LSA要素技術研究グループ トランスクリプトーム研究チーム
チームリーダー CARNINCI Piero (ピエロ・カルニンチ)(副センター長)
客員研究員 Alexandre FORT (アレクサンダー・フォート)
研究員 橋本 浩介 (はしもと こうすけ)
統合生命医科学研究センター 免疫器官形成研究グループ
グループディレクター 古関 明彦 (こせき はるひこ)
研究員 山田 大輔 (やまだ だいすけ)
お問い合わせ先
理化学研究所 ライフサイエンス技術基盤研究センター
チーフ・サイエンスコミュニケーター 山岸 敦 (やまぎし あつし)
Tel: 078-304-7138 / Fax: 078-304-7112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.ノンコーディングRNA(ncRNA)
non-coding RNA。メッセンジャーRNA(mRNA)と異なり、タンパク質の設計図として用いられないRNAの総称。エピジェネティクス(塩基配列に依存しない遺伝子の調節機構)や転写、翻訳といった生物の活動の中枢をなす反応、幹細胞性の維持など、さまざまな働きに関与するncRNAが次々に報告されており、その重要性に注目が集まってきている。 - 2.エンハンサー
主に遺伝子の上流や下流に位置し、遺伝子の転写効率を変化させるDNAの特定の配列のうち、転写効率を著しく高める部分をエンハンサー領域(配列)という。 - 3.レトロトランスポゾン
HIVなどのレトロウィルスのように、DNA→RNAへの転写と、RNA→DNAへの逆転写によって増殖する性質を持つ遺伝因子。ゲノム上のレトロトランスポゾンが転写されてRNAになり、さらに逆転写酵素によりDNAに戻ることでゲノム中を移動しコピー数を増やす。ヒトゲノムの進化の過程で、ほとんどのレトロトランスポゾンは不活性化されたと考えられている。 - 4.トランスクリプトーム
細胞内の全DNAの塩基配列情報を指す「ゲノム」に対し、細胞内の全転写産物(全RNA)をまとめてトランスクリプトームと呼ぶ。タンパク質の設計図となるmRNAよりもそれ以外のncRNAが多く存在することが明らかになり、それらの機能を明らかにする解析が進められている。 - 5.CAGE法
CAGEは Cap Analysis Gene Expressionの略。理研が独自に開発した手法で、耐熱性逆転写酵素やmRNAのCap構造を捕捉する技術を組み合わせて転写産物の5'末端の塩基配列を決定する実験手法。この塩基配列を読み取ってゲノム配列と照らし合わせて、どこから転写が始まっているかを調べることができる。遺伝子の転写開始店をゲノムワイドに同定できる。 - 6.NASTs
Non- Annotated- Stem- Transcript sの略。幹細胞の核に特異的な転写産物で、ヒトやマウスで数千種が存在する。これらの1/3はレトロトランスポゾンというウィルス由来の配列を含んでおり、幹細胞において、クロマチン構造や細胞周期など、多能性維持に必須な転写制御ネットワークに関与していると考えられている。 - 7.ENCODE
The Encyclopedia of DNA Elementsの略。米国・国立ヒトゲノム研究所(NHGRI)が2003年に立ち上げたヒトゲノム解析プロジェクト。ヒトゲノムのすべての機能要素の解析を目指している。世界5か国(スペイン、米国、英国、日本、シンガポール)から32の研究機関が参加している。日本からは唯一理研が参加している。
The ENCODE Project(英語) - 8.ヒストン修飾
DNAは、ヒストンというタンパク質に巻きついて「クロマチン」という構造を作り核内に収納される。そのクロマチンの構造が変わることで遺伝子発現のオン・オフが調節されるが、その構造変化はDNAメチル化や、ヒストンタンパク質にアセチル基やメチル基などが付くヒストン修飾によって制御されている。 - 9.スーパーエンハンサー
最近提唱された概念で、エンハンサー領域のうち、幹細胞性に関与する3つの主要な転写因子(Nanog、Pou5f1、Sox2)と、転写メディエーターのサブユニット(Med1かMed12)が結合しているものを指す。普通のエンハンサーに比べて、長い配列を持ち、DNaseIによる切断を受けやすいほか、転写因子の種類や結合量、転写活性化能が異なる。幹細胞以外の分化した細胞でも見られ、細胞種に特異的な遺伝子群の発現に寄与していると考えられている。がん治療の有望な標的領域となることが報告されている。 - 10.ロングノンコーディングRNA(lncRNA)
long non-coding RNA。100~200塩基かそれ以上の塩基からなる、ncRNAの中でも長いものを指す。全長にわたる保存性は高くないが、反復配列やウィルス由来の配列などの保存性の高い断片が含まれる。転写、翻訳、エピジェネティクスなど生体内の多様なプロセスに寄与していることが示されてきている。
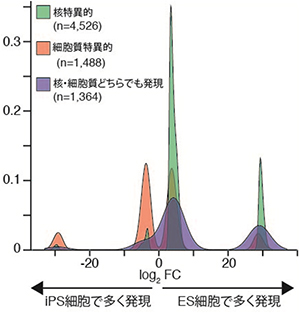
図1 ES細胞とiPS細胞で発現するRNAの比較
ES細胞で多く発現するRNAの割合が多く、特に核内で発現するRNAにその傾向が強い。
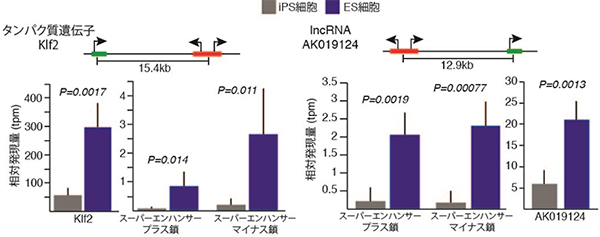
図2 スーパーエンハンサーと標的遺伝子の発現量の対応関係
タンパク質「Klf2」(左)、lncRNA「AK019124」(右)の発現量は、対応するスーパーエンハンサーのプラス鎖、マイナス鎖双方向の転写量と連動して、ES細胞では多く(青)、iPS細胞では少ない(灰色)。
