理化学研究所(理研)生命医科学研究センター遺伝子制御回路研究チームの河野掌リサーチアソシエイト、ジェイ・シンチームリーダー、ライフサイエンス技術基盤研究センターゲノミクス微量技術開発ユニットのシャルル・プレシユニットリーダー(研究当時)、生命医科学研究センターのピエロ・カルニンチ副センター長らの共同研究グループは、1細胞中に存在する転写[1]されたRNA[1]の5'末端[2]を網羅的に捉え、遺伝子発現量を計測する方法を新たに開発しました。
本研究成果は、現在盛んに研究されている1細胞生物学分野の発展や「ヒト細胞アトラス計画[3]」の推進に貢献すると期待できます。
ゲノム[1]から転写されたRNAにはさまざまな種類があり、器官や臓器を構成する各細胞種は、RNAの種類や量によって特徴づけられています。しかし、1細胞における遺伝子発現は、たとえ同じ細胞種であっても不均一です。したがって、それぞれの細胞種を正しく理解するには、RNAを1細胞単位で網羅的に検出、定量する必要があります。しかし、これまでの技術では、さまざまな種類のRNAの検出に偏りがあったり、ゲノム中のどの位置からRNAが転写されたかという情報が失われたりするという問題がありました。 今回、共同研究グループは、「CAGE法[4]」と全自動単細胞単離核酸調整機「C1システム[5]」を組み合わせた「C1 CAGE法」を開発し、ゲノムから転写されたRNAの5'末端を偏りなく定量的に検出することで、これまで見逃されていた1細胞中に存在する転写されたRNAの挙動を捉えることに成功しました。
本研究は、英国のオンライン科学雑誌『Nature Communications』(1月21日付け)に掲載されました。
※共同研究グループ
理化学研究所 生命医科学研究センター
遺伝子制御回路研究チーム
リサーチアソシエイト 河野 掌(こうの つかさ)
研究員 柴山 洋太郎(しばやま ようたろう)
チームリーダー ジェイ・シン(Jay Shin)
ゲノム情報解析チーム
特別研究員 ジョナサン・ムーディー(Jonathan Moody)
チームリーダー ヂョンチョウ・ホン(Chung-Chau Hon)
トランスクリプトーム研究チーム
技師 加藤 紗智(かとう さち)
大容量データ管理技術開発ユニット
研究員 イマド・アブケセーサ(Imad Abugessaisa)
ユニットリーダー 粕川 雄也(かすかわ たけや)
ゲノム制御ネットワーク研究チーム
研究員 グォン・テジュン(Kwon Tae Jun)
チームリーダー エリック・アーナー(Erik Arner)
ライフサイエンス技術基盤研究センター ゲノミクス微量技術開発ユニット(研究当時)
ユニットリーダー(研究当時)シャルル・プレシ(Charles Plessy)
理化学研究所生命医科学研究センター
副センター長 ピエロ・カルニンチ(Piero Carninci)
背景
ヒトの体は約37兆個の細胞で構成され、全ての細胞には同じゲノムが存在すると考えられています。器官や臓器を構成する各細胞種は、ゲノムから転写されたRNAの種類や量によって特徴づけられています。しかし、1細胞における遺伝子発現は、たとえ同じ細胞種であっても不均一です。したがって、それぞれの細胞種を正しく理解するには、RNAを1細胞単位で網羅的に検出、定量する必要があり、近年、1細胞を対象とした研究が盛んに行われています。
特に、「トランスクリプトーム解析[6]」は新しい細胞種の同定などに有用であり、さまざまな実験手法が開発されています。しかし、ほとんどの手法では、さまざまな種類のRNAの中で、ポリA[7]修飾を受けている成熟RNAの3'末端[2]側を対象として定量を行います。そのため、その他のポリA修飾を受けていないRNAや、遺伝子発現を制御するプロモーター[8]の下流に存在し転写開始点となる5'末端側、RNAの転写の方向性といった重要な情報は得られませんでした。
共同研究グループは、国際研究コンソーシアム「FANTOM[9]」において、RNAの5'末端を選択的に捉えて定量を行う「CAGE法」を用いて、これまでにヒト細胞中のプロモーター、エンハンサーRNA[10]を同定することに成功しています注1)。しかし、従来のCAGE法では、多量の細胞からキットを用いてRNAを抽出する必要があり、1細胞を対象にすることは困難でした。
米国のFluidigm社は、独自に開発した全自動単細胞単離核酸調整機「C1システム」を用いて、1細胞中に転写されているポリA修飾を受けている成熟RNAの末端領域以外を検出、定量することを可能にしました。しかし、調整過程でポリA修飾を受けていないRNAやRNAの5'末端側の情報が失われるという問題がありました。
そこで、共同研究グループは、偏りなくRNAの5'末端を選択的に検出・定量する方法の開発に取り組みました。
注1)2014年3月27日プレスリリース「ゲノム上の遺伝子制御部位の活性を測定し正常細胞の状態を定義」
研究手法と成果
共同研究チームは、Fluidigm社の全自動単細胞単離核酸調整機C1システムを用いて、1細胞中に転写されているごく微量のRNAから網羅的にRNAの5'末端を検出し、定量する「C1 CAGE法」を開発しました(図1)。C1 CAGE法では、逆転写プライマー[11]としてランダムプライマー[11]を使用することで、転写されていたRNAから偏りなくcDNA(相補的DNA)を合成します(逆転写反応)。また、cDNA合成時にTemplate Switching Oligo(TSO)[12]を用いて、RNAの5'末端に特異的な配列を付加させます。そしてcDNAの断片化後、RNAの5'末端となる領域をポリメラーゼ連鎖反応(PCR)[13]により増幅し、ライブラリーを調整します。 また、独自に開発したデータベースSCPortalen[14]上でC1システムのマイクロ流路上に捕捉されている1細胞の画像データと、実際に次世代シークエンサー[15]により定量されたRNAの転写量を比較することで、実験データの解釈の効率を向上させました。
開発したC1 CAGE法が、正しくRNAの5'末端を捕捉し検出しているかを確認するために、既知のマウス由来のES細胞[16]を用いて、C1 CAGE法とポリA修飾を受けているRNAを対象とした既存の方法を比較しました。その結果、C1 CAGE法では、ポリA修飾を受けていないRNAも含めたRNAの5'末端を捉えており、さらに遺伝子検出の数も既存の方法よりも改善されていることが分かりました(図2)。
次に、C1 CAGE法が正しくプロモーターやエンハンサーの活性を捉えているかどうかを評価するために、実際にヒト肺がん上皮細胞をTGFβ[17]により刺激し、0、6、24時間後の時間経過に伴う遺伝子ネットワークの変化を、細胞をラベル後、観測しました。その結果、1細胞が持つ不均一な遺伝子発現から、刺激応答の強さに応じた四つの細胞状態を確認することができました。さらに、五つの遺伝子制御ネットワーク(抑制型、脆弱性応答型1、脆弱性応答型2、早期応答型、遅延応答型)が刺激後の細胞状態を決定づけていることが分かりました(図3)。
さらに、遺伝子領域外に存在し、遺伝子の転写活性を調整するエンハンサーRNAに着目しました。これまで、エンハンサーRNAは正と負の双方向に転写されており、その発現も低いと考えられていました。しかし、C1 CAGE法によって1細胞単位で発現を観察したところ、双方向に転写されている細胞は稀に存在するだけで、多くの細胞では片方向にしか転写されていないことが分かりました。また、ごく一部の細胞のみが、プロモーターと同程度の高い発現を示していることが明らかになりました。これらの発見は、単一分子蛍光in situハイブリダイゼーション法[18]を用いて確認しました。
今後の期待
近年、1細胞中に存在する転写されたRNAを網羅的に定量する1細胞解析技術が盛んに行われていますが、多くの技術はポリAの修飾を受けた成熟RNAが対象であり、それ以外の転写されたRNAは見逃されがちです。しかし最近、これらのRNAが病気に関わっていることや、遺伝子の発現を制御していることが報告されています。本技術を用いて、1細胞中に転写された全てのRNAを検出、定量することは、各細胞の持つ不均一な発現を正確に理解する上で重要となります。
また、現在「ヒト細胞アトラス計画」が、世界中の研究者の協力の下で進行中です。集められたデータは、生命の理解や病気のメカニズムの解明に役立つと考えられており、本技術が新しい細胞種の発見などに貢献すると期待できます。
なお、関連する基礎情報は、論文発表と同時にWEBサイトZENBU[19]と、国立遺伝学研究所の日本DNAデータバンク(DNA Data Bank of Japan: DDBJ)のデータベース上で公開されます。また、C1 CAGE法はFluidigm社が管理するScript Hub上で公開されています注2)。
原論文情報
- Tsukasa Kouno*, Jonathan Moody*, Andrew Kwon*, Youtaro Shibayama, Sachi Kato, Yi Huang, Michael Böttcher, Efthymios Motakis, Mickaël Mendez, Jessica Severin, Joachim Luginbühl, Imad Abugessisa, Akira Hasegawa, Satoshi Takizawa, Takahiro Arakawa, Masaaki Furuno, Naveen Ramalingam, Jay West, Harukazu Suzuki, Takeya Kasukawa, Timo Lassmann, Chung-Chau Hon, Erik Arner, Piero Carninci, Charles Plessy# and Jay W Shin#, "C1 CAGE detects transcription start sites and enhancer activity at single-cell resolution", Nature Communications, 10.1038/s41467-018-08126-5
(*) Authors contributed equally
(#) Co-corresponding authors
発表者
理化学研究所
生命医科学研究センター 遺伝子制御回路研究チーム
リサーチアソシエイト 河野 掌(こうの つかさ)
チームリーダー ジェイ・シン(Jay Shin)
ライフサイエンス技術基盤研究センター ゲノミクス微量技術開発ユニット(研究当時)
ユニットリーダー(研究当時) シャルル・プレシ(Charles Plessy)
生命医科学研究センター
副センター長 ピエロ・カルニンチ(Piero Carninci)
 左よりグォン・テジュン、ジョナサン・ムーディー、河野 掌、ジェイ・シン
左よりグォン・テジュン、ジョナサン・ムーディー、河野 掌、ジェイ・シン
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
産業利用に関するお問い合わせ
補足説明
- 1.転写、RNA、ゲノム
ゲノムは細胞が持つ全遺伝情報であり、DNAに4種類の塩基(アデニン、チミン、グアニン、シトシン)の配列として記録されている。DNAからRNAを合成する段階を転写という。DNAの情報をコピーしたものがRNAである。 - 2.5'末端、3'末端
RNAは、ヌクレオシドの2'-リボースの5'位のヒドロキシ基と3'位のヒドロキシ基がリン酸ジエステル結合によって連結されてできている。RNAの5'位のヒドロキシ基側を5'末端と呼び、3'位のヒドロキシ基側を3'末端と呼ぶ。RNAの5'末端はDNA上の転写開始点に相当する。 - 3.ヒト細胞アトラス計画
ヒトの体内に存在する約37兆個を1細胞単位で解析して分類・カタログ化し、細胞の標準となるアトラス(地図)データを集める計画。2016から研究が始まり、疾患治療、再生医療の分野への貢献が期待されている。 - 4.CAGE法
理研が独自に開発した遺伝子解析技術で、耐熱性逆転写酵素やcap捕捉技術を組み合わせて、5'末端の塩基配列を決定する実験技法。この塩基配列を読み取ってゲノム配列と照らし合わせ、どこからどのくらい転写が始まっているかを調べることができる。遺伝子の転写開始点をゲノムワイドに同定できる世界唯一の解析技術。CAGEはCap Analysis of Gene Expressionの略。 - 5.C1 システム
マイクロ流路を用いて1細胞を単離し、RNA抽出後、逆転写プライマーにより効率良く1細胞由来のcDNAに変換するシステム。 - 6.トランスクリプトーム解析
DNAから転写されるRNAなどの全転写産物を網羅的に調べる手法の総称。 - 7.ポリA
タンパク質をコードしているRNA、メッセンジャーRNA(mRNA)の3'末端に50~200塩基ほどのアデニン(A)ヌクレオチドが付加されており、これをポリAテールと呼ぶ。ポリAは、mRNAに安定性を与え、翻訳を促進する働きがあると考えられている。 - 8.プロモーター
mRNA合成の開始に関与するDNA上の特定領域の短い塩基配列のこと。プロモーター領域にRNAを合成する酵素であるRNAポリメラーゼが結合し、転写が開始される。 - 9.FANTOM
理化学研究所が主催する国際研究コンソーシアム。理研のマウスゲノム百科事典プロジェクトで収集された完全長cDNAの機能注釈(アノテーション)を行うことを目的に、理研ゲノム科学総合研究センターの林崎良英グループディレクター(現:予防医療・診断技術開発プログラムプログラムディレクター)が中心となり、2000年に結成された。その成果は、iPS細胞(人工多能性幹細胞)の樹立研究など生命科学の広い分野に貢献している。5期目のプロジェクトとなるFANTOM5では、さまざまな哺乳類細胞のゲノム上の遺伝子制御部位の活性を測定し、転写状態やプロモーター活性の全容を明らかにする研究が進められた。現在のFANTOM6には20カ国、約60の研究機関が参加し、ノンコーディングRNAの網羅的な機能解析に取り組んでいる。FANTOMはFunctional ANnoTation Of the Mammalian genomeの略。 - 10.エンハンサーRNA
エンハンサー領域は、ゲノムDNA上で制御対象である遺伝子とは遠く離れた領域に位置し、遺伝子の転写効率を向上させる働きを持つ。エンハンサーRNAは、エンハンサー領域から転写されるノンコーディングRNA。 - 11.逆転写プライマー、ランダムプライマー
RNAを鋳型とし相補的なDNA(cDNA)を合成する反応を逆転写反応という。RNAウイルスが宿主細胞に進入し自己複製する際に働くRNA依存性DNA合成酵素である逆転写酵素を利用する。逆転写酵素がcDNA合成を行うには、RNA配列に相補的に結合した短いDNA鎖(逆転写プライマー)を出発点とする必要がある。ランダムプライマーを用いることで、偏りなくRNAからcDNAを合成できる。 - 12.Template Switching Oligo (TSO)
3'末端に連続したグアニン(G)を持つように設計したDNAオリゴのこと。 - 13.ポリメラーゼ連鎖反応(PCR)
熱耐性を持つDNA依存性DNA合成酵素を用いて、鋳型DNAとPCRプライマーの結合、相補DNAの合成、二本鎖DNAの解離の3ステップを繰り返すことで、DNAを連続的かつ指数関数的に増幅させることができる反応。鋳型DNAの長さや配列に依存した増幅バイアスが生じる。長いDNAは増えにくい。さまざまな種類のRNAを由来とするcDNAを増幅させるには、PCRプライマーが結合するための共通配列を予め逆転写プライマーに付加させておく必要がある。PCRはPolymerase Chain Reactionの略。 - 14.SCPortalen
1細胞の画像イメージと、次世代シークエンサーにより得られた情報を統合するデータベース。
SCPortalen: human and mouse single-cell centric database(英語) - 15.次世代シークエンサー
サンガー法を利用した蛍光キャピラリーシーケンサーである「第一世代シーケンサー」と対比させて使われている用語。多数のDNA断片を同時並行で解析し、大量の配列を読み取ることができるDNA配列解析装置。 - 16.ES細胞
胚性肝細胞。ほ乳類の着床前胚(胚盤胞)に存在する内部細胞塊から作製した細胞株で、体を構成する全ての種類の細胞に分化する能力(多能性)を持つもの。 - 17.TGFβ
細胞の増殖や分化を制御する因子であり、細胞外基質成分の産生を促進することが知られている。Transforming growth factor-βの略。 - 18.単一分子蛍光in situハイブリダイゼーション法
細胞内において転写されている特定のRA配列を検出する手法。目的となるRNA配列に相補的に結合する核酸を蛍光で印をつけ、それらと結合するRNAの細胞内の位置を1細胞レベルで、光学顕微鏡により観察する手法。 - 19.ZENBU
理研が開発したバイオインフォマティックス・ツール。次世代シークエンサーから得られたデータを、遺伝子発現情報の可視化や視覚化を可能とするツール。
2014年3月10日プレスリリース「 新しいバイオインフォマティクス・ツール「ZENBU」を開発」
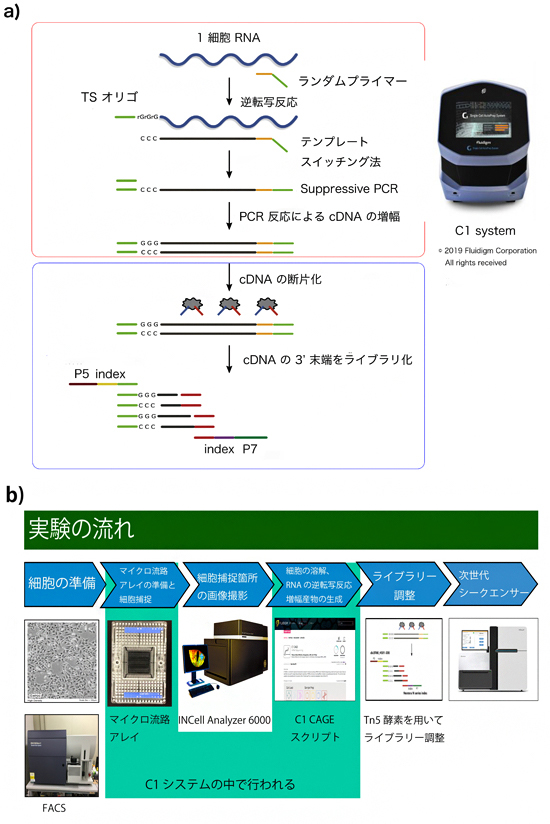
図1 今回開発したC1 CAGE法の反応と実験の流れ
- a) C1 CAGE法の反応図。C1 システムの中で、1細胞捕捉、ランダムプライマーによるRNAからcDNAへの逆転写反応、PCRによる増幅が行われる。生成された増幅産物は、市販のキットと独自の手法により、短く断片化され、RNAの5'末端を捉えることが可能である。
- b) 今回開発した実験の流れ。C1 アレイ上で捕捉された1細胞は、IN Cell Analyzer 6000という機械でイメージングを行い、シークエンスデータと比較することを可能とした。
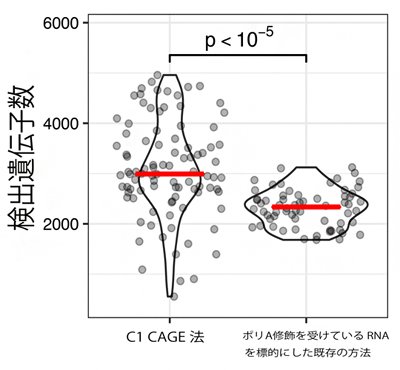
図2 C1 CAGE法と既存の方法による検出遺伝子数の比較
既知のマウス由来のES細胞を用いて、C1 CAGE法とポリA修飾を受けているRNAを対象とした既存の方法とで、検出された遺伝子数を比較した。赤線は中央値。
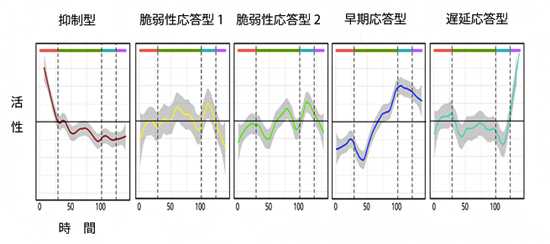
図3 C1 CAGE 法によって得られた遺伝子制御ネットワーク
定量データを共発現遺伝子ネットワーク解析にかけ、同定できたヒト肺がん上皮細胞の薬剤刺激後の細胞状態を決定づける五つのパターン。図中の上の色は、四つに分類された細胞状態を示す。横軸の数字は、解析により示された擬似的な時間を示す。
