理化学研究所(理研)革新知能統合研究センター遺伝統計学チームの矢野憲司特別研究員、田宮元チームリーダー、龍谷大学の吉田晋弥客員研究員、名古屋大学の松岡信教授らの共同研究グループは、機械学習[1]と従来の遺伝子同定法を組み合わせた手法により、イネの収量に関わる遺伝子を同定しました。
本研究成果は、イネの収量増加に貢献すると期待できます。また今後、本研究で使用した解析手法を、イネ以外の作物にも応用することが可能です。
イネの収量は、イネの丈や穂の数、穂の構造など、複数の要素により決定されます。このような複雑な要素を解析し、それに関わる遺伝子を同定するには、新しい解析手法が必要でした。
今回、共同研究グループは、日本で育成されたイネ169品種における収量に関わる遺伝子を同定するため、教師なし機械学習[1]の一つである主成分分析[2]とゲノムワイド関連解析(GWAS)[3]を組み合わせた手法を開発しました。その結果、イネの収量を制御する遺伝子を同定することに成功しました。
本研究は、米国の科学雑誌『Proceedings of the National Academy of Sciences of the United States of America(PNAS)』の掲載に先立ち、オンライン版(9月30日付け:日本時間10月1日)に掲載されました。
背景
有用な遺伝子の同定は、作物の育種において収量の増加やその遺伝的機構解明に極めて重要です。
イネの収量は、草型と呼ばれる形態と密接に関わっています。草型は、一般に穂の数と穂の大きさにより、大きく二つに分けられます(図1)。1株当りの穂の数が多く小さい穂をつけるタイプは「穂数型」、穂の数が少なく大きい穂をつけるタイプは「穂重型」と呼ばれています。さらに、イネの穂は、穂軸、一次枝梗(しこう)、二次枝梗、種子などの要素によって構成されており、これらの要素により穂の大きさが決められています(図2)。こうした形質は、発芽から花が咲くまでの日数やイネの丈にも影響します。
イネの収量は、複数の要素が複雑に関わり合って決まることから、総合的にこれらの要素を解析し、収量に関わる遺伝子の同定を行うことはこれまで困難でした。

図1 イネの草型の分類(左:穂数型、右:穂重型)
穂数型は1株当りの穂の数が多く小さい穂をつけるタイプ、穂重型は穂の数が少なく大きい穂をつけるタイプである。
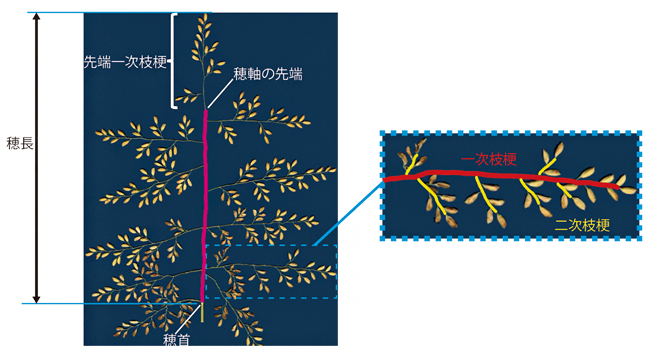
図2 イネの穂構造(左:穂の全体図、右:一次枝梗の拡大図)
穂軸は、穂首から先端一次枝梗の基部(左図の紫の部分)まで、一次枝梗は、穂軸から最初に分岐した枝梗(右図で赤色の部分)、二次枝梗は、一次枝梗から分岐した枝梗(右図で黄色の部分)である。
研究手法と成果
共同研究グループでは、日本で育成されたイネ169品種を用いて、草型の指標となる8形質(花が咲くまでの日数、イネの丈、穂の数、穂の長さ、穂軸の長さ、一穂当たりの一次枝梗の数、一穂当たりの二次枝梗の数、一穂当たりの種子の数)を計測し、教師なし機械学習と従来の遺伝子同定手法であるゲノムワイド関連解析(GWAS)を組み合わせることで、草型に関わる遺伝子の同定を試みました。
GWASとは、ある集団(個体の集まり)を設定し、集団に存在する個体の間の形質の違いとDNA配列の違いとの関連をゲノム全体にわたって調べることにより、対象となる形質と関連するDNA多型[4]を統計的に検出する手法です。作物の収量や病気への耐性といった性質は、おのおのの品種の持つゲノムの遺伝的な変異に由来します。作物の育種の過程において、ある遺伝的な変異が有益な形質をもたらした場合にはその変異は選抜されますが、選抜の目的や環境により有益な表現型は異なるため、育種された品種のゲノムは多様性を持つことになります。このような集団に対するGWASにより、ゲノム全体の中から標的とした形質に関わるDNA多型を見つけ出すことができます。本研究では、次世代シーケンサー[5]により全ゲノムシーケンス解析[6]を行い、169品種のゲノム情報を得ました。
一方、主成分分析とは、複数の因子を要約した値(特徴量)を抽出する教師なし機械学習手法の一つです。本研究の場合、草型の指標となる8形質を用いて主成分分析を行い、これらの形質の特徴量を抽出しました。この特徴量と関連しているDNA多型を見つけるためにGWASを行った結果、571多型が見いだされました。これらの多型について遺伝子機能への影響を調べたところ、34多型が14の候補遺伝子の機能に関わる可能性があることが分かりました。これらの遺伝子の中には、植物ホルモン「ジベレリン」のシグナル制御に関わるOsSPYが含まれていました(図3)。ジベレリンは、植物の丈に関わることが知られているホルモンです。そこで、この遺伝子に着目し、分子遺伝学的実験[7]により検証を行ったところ、イネの丈だけでなく、穂の数、穂の構造の制御に多面的に関わることが分かりました。つまり、この遺伝子が草型の穂重型と穂数型を決めている一因であることが明らかになりました。
これらの結果から、主成分分析はイネの草型評価に関わる形質を要約することを可能にするとともに、GWASとの統合は、複雑形質に関わる遺伝子の同定に有効であると結論付けられました。
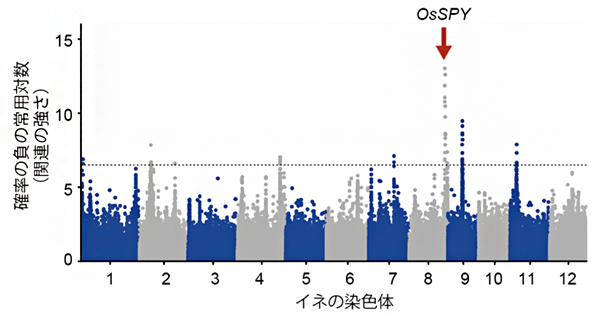
図3 ゲノムワイド関連解析の結果
赤矢印は、本研究で同定した収量に関わるゲノム領域を示す。この領域には、14の候補遺伝子が見つかり、その中に植物ホルモン「ジベレリン」のシグナル制御に関わるOsSPYが含まれていた。
イネは、古くから各地に存在する「在来品種」と比較的最近の在来種をもとに育成された「近代品種」に分類されます。草型との関連で、在来品種では穂重型、近代品種では穂数型が多い傾向があります。近代品種において丈の低い穂数型が多いのは、多肥栽培によって、イネの丈が伸びすぎて倒伏してしまうことを防ぐためだと考えられています。
そこで、在来品種と近代品種の遺伝距離[8](遺伝的分化の尺度)を全ゲノムにわたって調査しました。この調査により、在来品種から近代品種へ変移する過程で選抜された可能性のあるゲノム上の領域を統計学的に調べることができます。解析の結果、OsSPYを含むゲノム領域は、他の領域に比べ在来品種から近代品種の変移の過程で選抜された可能性が高いことが確認されました。また、イネの原産国である中国の品種を用いた解析においても同様の結果が確認されました。これらの結果は、日本だけでなく中国においても、イネの近代品種を育成する際にOsSPYが選抜の対象になったことを示唆しています。
今後の期待
今回、共同研究グループは、主成分分析とGWASを組み合わせることで、作物のイネにおける収量にかかわる遺伝子の同定を試み、一因となる遺伝子を同定することに成功しました。本手法は、本研究で解析対象としたイネだけでなく、他の植物種にも応用することが可能です。さらに、収量形質だけでなく、今後病気への耐性や環境応答などの複雑形質に関わる遺伝子の同定にも利用できると期待できます。
また、今後本研究で同定したOsSPYを育種に利用することも考えられます。近年、環境問題の観点から、より少ない化学肥料で作物を栽培することが求められています。低肥料の栽培環境では、穂数型より穂重型の方が収量に対して有利である可能性があり、OsSPYを利用して低肥料栽培に適したイネを育成できるかもしれません。このように、今後この遺伝子を草型の調節に利用することで、イネの収量増加に貢献すると期待できます。
補足説明
- 1.機械学習、教師なし機械学習
機械学習は、「分類」や「予測」を行うための統計学的な算法のことで、解析データから特徴量を抽出することができる。教師なし機械学習は、事前情報がない状態で特徴量を抽出する機械学習手法の一つ。 - 2.主成分分析
複数の量的な変数を、要約した指標(特徴量)にまとめることができるデータ解析手法。 - 3.ゲノムワイド関連解析(GWAS)
形質の違いとDNA配列の違いとの関連をゲノム全体にわたり調べることにより、対象となる形質と関連するDNA多型を統計的に検出する手法。GWASはgenome-wide association studyの略。 - 4.DNA多型
特定の集団のうち、個体ごとでDNA配列が違うこと、またはその異なったDNA配列のこと。集団における表現型の多様性を生む一つの原因となっている。 - 5.次世代シーケンサー
個体ごとにゲノムの塩基配列を高速に決定することができる装置。 - 6.全ゲノムシーケンス解析
次世代シーケンサーにより、生物の遺伝情報を包括的に取得する手法。 - 7.分子遺伝学的実験
遺伝現象の仕組みをDNAなどの分子を用いて調査する実験のこと。遺伝子への突然変異の誘発やDNA塩基配列の比較などを行うことで、遺伝子の機能を調べることができる。 - 8.遺伝距離
異なる集団の間で、遺伝的分化がどれほど近いか、遠いかを表す尺度。
研究支援
本研究は、文部科学省の次世代人工知能技術等研究開発拠点形成事業費補助金、日本学術振興会(JSPS)特別研究員奨励費「ゲノム情報利用による根機能を制御する有用遺伝子の単離と分子育種(領域代表者:矢野憲司)」、若手研究B「イネの高温登熟耐性を制御する分子メカニズムの解明(領域代表者:矢野憲司)」、基盤研究A「栽培環境に呼応して変動するQTL遺伝子の単離と同定(領域代表者:松岡信)」、特別研究員奨励費「イネ窒素利用に関するG×E GWASによる研究(領域代表者:松岡信)」、新学術領域研究(研究領域提案型)「植物新種誕生の原理-生殖過程の鍵と鍵穴の分子実態解明を通じて-(領域代表者:東山哲也)」、新学術領域研究(研究領域提案型)「生殖をモデルとした植物ホルモン機能拡張(領域代表者:上口美弥子(田中美弥子))」による支援を受けて行われました。
原論文情報
- Kenji Yano, Yoichi Morinaka, Fanmiao Wang, Peng Huang, Sayaka Takehara, Takaaki Hirai, Aya Ito, Eriko Koketsu, Mayuko Kawamura, Kunihiko Kotake, Shinya Yoshida, Masaki Endo, Gen Tamiya, Hidemi Kitano, Miyako Ueguchi-Tanaka, Ko Hirano, Makoto Matsuoka, "GWAS with principal component analysis identifies a gene comprehensively controlling rice architecture", Proceedings of the National Academy of Sciences of the United States of America(PNAS), 10.1073/pnas.1904964116
発表者
理化学研究所
革新知能統合研究センター 目的指向基盤技術研究グループ 遺伝統計学チーム
特別研究員 矢野 憲司(やの けんじ)
チームリーダー 田宮 元(たみや げん)
龍谷大学 食と農の総合研究所
客員研究員 吉田 晋弥(よしだ しんや)
(研究当時 兵庫県農林水産技術総合センター 研究主幹)
名古屋大学 生物機能開発利用研究センター
教授 松岡 信(まつおか まこと)
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
名古屋大学 総務部 総務課 広報室
Tel: 052-789-2699 / Fax: 052-789-2019
E-mail: nu_research [at] adm.nagoya-u.ac.jp
龍谷大学 学長室(広報)
Tel: 075-645-7882
E-mail: kouhou [at] ad.ryukoku.ac.jp
※上記の[at]は@に置き換えてください。
