理化学研究所(理研)環境資源科学研究センター技術基盤部門生命分子解析ユニットの野村勇太特別研究員と堂前直ユニットリーダーは、ヒトのがん抑制遺伝子[1]の発現に影響を与える新しい遺伝子を発見しました。
本研究成果は、今後のがん研究に加え、がんの性質や治療効果を評価する指標(バイオマーカー)の開発などに役立てられるものと期待できます。
これまでの遺伝子探索では、150個未満のアミノ酸からなる短いタンパク質をコードする遺伝子はその対象からほとんど除外されていました。
今回、野村特別研究員と堂前ユニットリーダーは公開データ・リポジトリ[2]から入手できるヒトのがん細胞株のトランスクリプトームデータ[3]とプロテオームデータ[4]を用いて、情報科学的解析(プロテオ・ゲノミクス解析)を行いました。その結果、120個のアミノ酸からなる短いタンパク質をコードする新しい遺伝子を発見し、その遺伝子がゲノム上でがん抑制遺伝子のSCRIB遺伝子と一部重なっていたことから、「oSCRIB」と命名しました。
最近の研究で、SCRIB遺伝子はがん原遺伝子[5]としても機能することが分かっており、細胞が正常に機能するには、SCRIB遺伝子が高発現・低発現せず、その発現量の適切な保持が重要とされています。そこで、生化学的手法を用いて、oSCRIB遺伝子がSCRIB遺伝子の発現を抑制していることを示しました。つまり、正常細胞では、oSCRIB遺伝子はSCRIB遺伝子の過剰発現を抑制する安全装置として機能していたと考えられます。また、ヒトの臨床プロテオームデータの再解析結果から、がん細胞ではoSCRIB遺伝子による安全装置の効果が弱められ、SCRIB遺伝子が高発現していることも示しました。
本成果は、科学雑誌『Communications Biology』(9月17日付)にオンライン掲載されます。
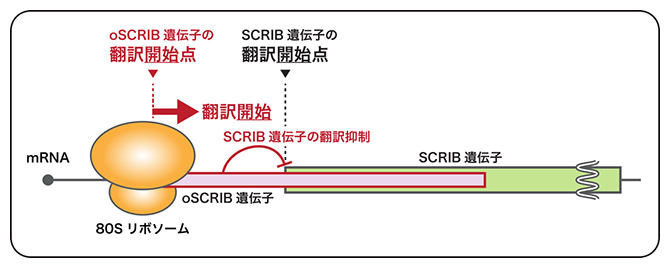
oSCRIB遺伝子によるSCRIB遺伝子の発現制御モデル
背景
2003年に完了した国際プロジェクト「ヒトゲノム計画」では、生命の設計図にあたるDNAの全塩基配列が解読されました。これによって、生命現象を担うタンパク質を構成するアミノ酸の配列情報が記されたDNA領域(遺伝子領域[6])が、ヒトでは約2万2000カ所以上存在することが分かりました。
ヒトゲノム計画の完了が宣言されてから18年が経過した今もなお、ヒトゲノムに隠された新しい遺伝子の探索は続けられています。特に、150個未満のアミノ酸からなる短いタンパク質やペプチド(Short ORF-encoded polypeptide; SEP)をコードする短い遺伝子の一群は、これまでの遺伝子探索の過程ではその対象からほとんど除外されていました。そのため、SEPファミリー遺伝子の探索を入念に進めることで、これまで知られていなかったタンパク質の一群(ダークプロテオーム)と、それらを中心とした新しい生命現象を明らかにできる可能性があります。
また、これまで長い間、私たちヒトを含む真核生物のメッセンジャーRNA(mRNA)には一つの遺伝子情報が含まれ、一つのタンパク質だけが合成されると信じられてきました。このmRNAは「モノシストロニックなmRNA[7]」と呼ばれます。一方、二つ以上の遺伝子情報が含まれ、二つ以上のタンパク質が合成されるmRNAは、「ポリシストロニックなmRNA[7]」と呼ばれます。SEPファミリー遺伝子は、これまでモノシストロニックなmRNAと考えられてきたmRNA上に重なって配置されていることも多く、ゲノムやmRNAの塩基配列情報だけからその存在を確定させることは困難です。そのため、SEPを発見するには、質量分析装置[8]を用いた直接的な検出が最も有効な手段の一つになっています。
研究手法と成果
野村特別研究員と堂前ユニットリーダーは、オンライン公開データ・リポジトリから入手できる、4種類のヒトがん細胞株(HeLa、MCF-7、A549、HCT-116)のトランスクリプトームデータを再解析し、従来の遺伝子探索では除外されることの多かったSEP候補のアミノ酸配列データベースを構築しました。続いて、このデータベースをSEP探索の指標としてコンピュータで照合すること(プロテオ・ゲノミクス解析)で、上記のヒトがん細胞株のプロテオームデータから新しいSEPを探索しました。その結果、さまざまなSEPの存在を見いだすことに成功しました。
そのうち、特に重要度が高いと考えられる120個のアミノ酸からなるSEPについて研究を進めました。まず、コンピュータ予測の真偽を検証するために、PUREシステムと呼ばれる試験管内タンパク質合成法(原核型の無細胞翻訳法[9]の一つ)を利用し、対象のSEPの標品タンパク質を調製しました。この標品タンパク質をトリプシンで消化したペプチド混合物を質量分析装置で解析し、取得した質量スペクトルおよびカラム保持時間の特性から、対象SEPが実際にヒト細胞で発現していることが明らかになりました。
このSEPの遺伝子は、細胞が正常に機能する上で必須な細胞極性を決定する足場タンパク質[10]SCRIBの遺伝子の上流部に位置しており、約80%の遺伝子領域がSCRIB遺伝子に重なるという特徴を持っていました(下図)。これにより、このSEP遺伝子を「oSCRIB(SEP-encoding overlapping ORF of SCRIB)」と命名しました。
SCRIB遺伝子は、さまざまな細胞内シグナル伝達経路(Hippo-YAP/TAZ、MAPK/ERK、PI3K/Akt/mTORなど)の重要な制御タンパク質として機能し、がん抑制遺伝子として広く認知されています。一方で、がん細胞では高発現し、がん原遺伝子としても機能することが近年明らかになっています。そのため、細胞が正常に機能するには、SCRIB遺伝子が高発現・低発現せず、その発現量が適切に保持されることが重要だと考えられます。
oSCRIB遺伝子はSCRIB遺伝子と一部重なっていたことから、oSCRIB遺伝子の発現がSCRIB遺伝子の発現に直接的な影響を及ぼす可能性が考えられました。そこで、真核型リボソームによる遺伝子発現を試験管内で再現する生化学的手法(真核型の無細胞翻訳法)を用いて、oSCRIBとSCRIBの二つの遺伝子をそれぞれコードしたポリシストロニックなmRNAを作製し、発現制御機構を調べました。その結果、oSCRIB遺伝子の翻訳ができないmRNAでは、SCRIB遺伝子の翻訳が過剰に生じることが分かりました。このことから、oSCRIB遺伝子は、SCRIB遺伝子の発現を抑制する(負に制御する)調節エレメント[11]であり、正常細胞においてSCRIB遺伝子の過剰発現を抑制する安全装置として働くと考えられます(下図)。
また、ヒトの臨床プロテオームデータを再解析し、ヒトの乳がんなどではSCRIB遺伝子の発現に加え、oSCRIB遺伝子の発現が調節不全となっていることを見いだしました。これは、がん細胞がoSCRIB遺伝子による安全装置の効果を弱め、SCRIB遺伝子の高発現を図っていることを示す興味深い結果です。
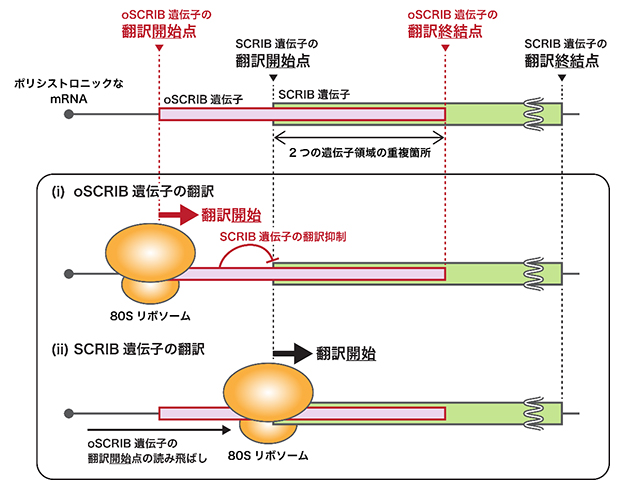
図 oSCRIB遺伝子によるSCRIB遺伝子の発現制御モデル
oSCRIB遺伝子はSCRIB遺伝子に重なって存在しているため、タンパク質合成装置「リボソーム」は、oSCRIB遺伝子の翻訳を始めると、SCRIB遺伝子の翻訳ができない(oSCRIB遺伝子の翻訳を始めなければ、SCRIB遺伝子の翻訳ができる)。oSCRIB遺伝子は、がん原遺伝子にもなるSCRIB遺伝子の過剰発現を抑制する安全装置として機能していると推定される。
今後の期待
網羅性の高いプロテオームデータは、いわば未知のタンパク質群(ダークプロテオーム)の情報をたくさん詰め込んだ「宝箱」のようなものです。今回新たに構築したSEPデータベースを手掛かりとしたプロテオ・ゲノミクス解析では、さらに多くの未知のSEPの存在が推定されたことから、試験管内タンパク質合成法による標品タンパク質の調製とそれを用いたコンピュータ予測結果の再検証により、より多くのSEPを同定できると考えられます。
また、今後のがん研究やがんの性質を調べる指標(バイオマーカー)としての応用につながるものと期待できます。
本成果は、国際連合が2016年に定めた17項目の「持続可能な開発目標(SDGs)[12]」のうち、「3. すべての人に健康と福祉を」への貢献が期待できます。
補足説明
- 1.がん抑制遺伝子
細胞のがん化を抑制する機能を持つタンパク質をコードする遺伝子。 - 2.公開データ・リポジトリ
世界中の多くの研究者によって取得された膨大なデータセットを保管するオンラインのデータ・ストレージ。 - 3.トランスクリプトームデータ
生細胞などから抽出したmRNAの全塩基配列を解読したデータセット。 - 4.プロテオームデータ
質量分析装置を用いて取得された生細胞などの網羅的なタンパク質の質量情報。 - 5.がん原遺伝子
遺伝子の過剰発現やそのタンパク質の細胞局在性の撹乱などで、結果として細胞のがん化を引き起こす原因となる遺伝子。 - 6.遺伝子領域
タンパク質をコードする遺伝子領域は、まずメッセンジャーRNA(mRNA)に転写される。続いて、リボソームと呼ばれる細胞内のタンパク質合成工場において、mRNAに写し取られたアミノ酸の配列情報(三つの塩基の配列が一つのアミノ酸情報に対応)を一つ一つ読み取りながら、アミノ酸を正しい順番でつなぎ合わせタンパク質を合成する。mRNAでは、三つの塩基を1組(コドン)として区切っていく方法(読み枠)は3通り存在するが、その中で翻訳の開始と終結を知らせるコドン(開始コドン、終止コドン)とそれに囲まれた部分(Open reading frame; ORF)が、タンパク質のアミノ酸配列情報(遺伝子情報)を記している可能性がある。 - 7.モノシストロニックなmRNA、ポリシストロニックなmRNA
一般的には、ヒトなど真核生物では、一つのmRNAに一つの遺伝子情報が含まれるように転写されるため、基本的には一つのmRNAから合成されるタンパク質は一つであると信じられてきた。このようなmRNAをモノシストロニックなmRNAと呼ぶ。しかし最近では、二つ以上の遺伝子情報を含んだmRNAの存在が徐々に明らかになってきており、これをポリシストロニックなmRNAという。これは、ヒトゲノムにより多くの未知な遺伝子が書き込まれていることを示唆していて、新しい遺伝子の探索が今後さらに期待される理由でもある。細菌やウイルスでは、生存戦略の一つとして遺伝情報をコンパクトにまとめたり、機能的に関連する複数の遺伝子群を効率良く同調的に発現させたりするために、むしろポリシストロニックなmRNAが主流だが、ヒトなど真核生物のポリシストロニックなmRNAの研究はまだ始まったばかりである。 - 8.質量分析装置
物質をイオン化して、その質量を測定する装置。 - 9.無細胞翻訳法
細胞抽出液などを用いて試験管内でタンパク質を合成する方法。 - 10.足場タンパク質
さまざまなタンパク質群と結合して複合体を形成するための足場となるタンパク質。 - 11.調節エレメント
遺伝子領域の発現を調節する領域。 - 12.持続可能な開発目標(SDGs)
2015年9月の国連サミットで採択された「持続可能な開発のための2030アジェンダ」にて記載された2016年から2030年までの国際目標。持続可能な世界を実現するための17のゴール、169のターゲットから構成され、発展途上国のみならず、先進国自身が取り組むユニバーサル(普遍的)なものであり、日本としても積極的に取り組んでいる(外務省ホームページから一部改変して転載)。
原論文情報
- Yuhta Nomura* and Naoshi Dohmae*, "Discovery of a small protein-encoding cis-regulatory overlapping gene of the tumor suppressor gene Scribble in humans", Communications Biology, 10.1038/s42003-021-02619-8 *Corresponding authors
発表者
理化学研究所
環境資源科学研究センター 技術基盤部門 生命分子解析ユニット
特別研究員 野村 勇太(のむら ゆうた)
チームリーダー 堂前 直(どうまえ なおし)
 野村 勇太
野村 勇太
 堂前 直
堂前 直
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
