理化学研究所(理研)生命機能科学研究センター高機能生体分子開発チームの八木創太基礎科学特別研究員、田上俊輔チームリーダー、構造バイオインフォマティクス研究チームのアディティア・クマール・パディ訪問研究員、ケム・ツァンチームリーダー、分子配列比較解析チームの中川れい子専門職研究員らの国際共同研究グループは、生命誕生の初期から存在したと考えられる原始的なタンパク質構造が、わずか7種類のアミノ酸だけで創れることを実証しました。
本研究成果は、生命の初期進化におけるタンパク質の誕生過程を再現するもので、生命誕生の謎を解くための重要な手掛かりとなると期待できます。
現生生物は、遺伝情報に従って20種類のアミノ酸を数珠状につなげることで、さまざまな機能を持つタンパク質を作ります。大きく複雑な立体構造を持つタンパク質は、より単純で短いものから徐々に進化したと考えられていますが、その過程は明らかになっていません。
今回、国際共同研究グループは、原始的なタンパク質構造と考えられているDouble-psi-beta-barrel(DPBB)[1]の誕生・進化過程を実験的に再現しました。現在の生物が持つDPBBの構造および配列情報を基に祖先の配列を推定し、古代に存在したと考えられるDPBBを再構成したところ、最も単純な古代DPBB構造は7種類のアミノ酸50個程度からなる短いペプチド[2]配列で成立することが分かりました。これら7種のアミノ酸はコドン表[3]においてまとまった位置に局在しており、太古の原始的なタンパク質合成[3]システムによってDPBBが生産されていた可能性が示されました。
本研究は、科学雑誌『Journal of the American Chemical Society』オンライン版(9月24日付)に掲載されました。
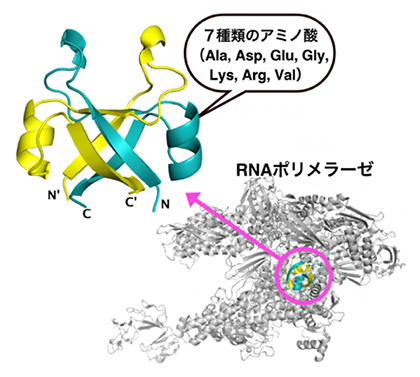
RNAポリメラーゼの中心部にあるDPBB(右下)と7種類のアミノ酸で復元された古代DPBB構造
背景
タンパク質は生命における最も重要な機能分子ですが、いつどのように地球上に誕生したのかは生命科学における大きな謎の一つです。現生生物は遺伝情報に従って20種類のアミノ酸を数珠状につなげることでタンパク質を作っています。その多くは数百~数千個のアミノ酸が連なり、非常に複雑な立体構造を持った巨大分子です。しかし、生物誕生の初期にはこのような複雑なタンパク質は存在せず、より単純で短いものから始まって、徐々に進化し複雑になったと考えられています。初期のタンパク質は、単純な立体構造の繰り返しや組み合わせで形作られ、生命の基本活動である代謝反応やセントラルドグマ[3]を担っていたと想像されます。しかし、古代のタンパク質がどれほど単純な分子であったかは未解明のままです。
現生生物のタンパク質を調べると、異なる機能を持つタンパク質において、部分的に非常によく似た立体構造が見られることがあります。その一つがDouble-psi-beta-barrel(ダブル・サイ・ベータ・バレル、以下DPBB)と呼ばれる約90個のアミノ酸からなる構造です。生命機能に不可欠な種々の酵素に見いだされることから、生物誕生の初期に登場した可能性が高いと考えられています(図1)。特に、全ての生物において遺伝子発現を担うRNAポリメラーゼ[4]の活性中心にも存在することから、DPBBの誕生は生命の中心原理であるセントラルドグマの起源にも密接に関係しているはずです。
そこで国際共同研究グループは、DPBB構造の誕生と進化を探るため、どれほど単純なアミノ酸配列でDPBB構造が成立できるかを実験的に検証しました。

図1 種々の酵素で保存されるDPBB構造
DPBB構造を持つ酵素の例。タンパク質の立体構造をリボンモデルで示し、DPBB構造をカラーで、他の部分を灰色で色分けした。RNAポリメラーゼは二つ、VCPシャペロンは六つ、ギ酸デヒドロゲナーゼは一つのDPBB構造を持つ。
研究手法と成果
本研究では、現生生物が持つDPBBタンパク質を出発材料として、段階的に古代DPBBの復元・解析を行いました。
古代DPBB構造を復元する上で重要な手掛かりとなるのが、DPBBが持つ内部対称性[5]です。DPBBは、約90個のアミノ酸が複数のαヘリックス[1]とβシート[1]を作り、それらがさまざまな向きに配置された複雑な構造をしています。しかし内部構造を詳しく見ると、N末端[2]側半分とC末端[2]側半分のアミノ酸配列が非常に似ており、二つのよく似た立体の組み合わせでできていることが分かります(図2)。そのため、DPBB遺伝子の起源は、半分の長さのおよそ45個のアミノ酸に対応する祖先遺伝子が重複・融合した結果、誕生したと考えられています。
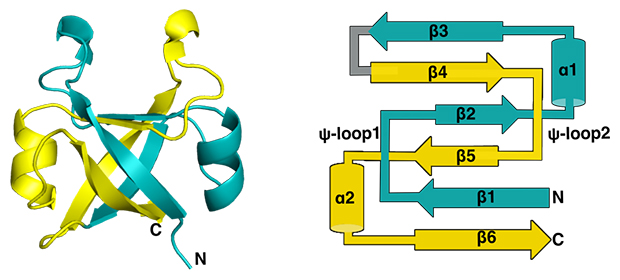
図2 DPBB構造が持つ内部対称性
超好熱性古細菌(Methanopyrus kandrerii)由来のVCPシャペロンが持つDPBBのX線結晶構造(左)と二次構造の模式図(右)。1本のタンパク質が複雑に折り畳まれているが、よく見ると二つのよく似た構造が向かい合った回転対称形になっていることが分かる。αはαヘリックス、βはβストランド、⍦-loopはDPBBに特徴的なループ構造を表し、それぞれ数字はN末端側からの順番を指す。DPPBをN末端半分(シアン)とC末端半分(黄)に分けると、β5、α2、β6(以上黄)とβ1、⍦-loop1(以上シアン)がX線結晶構造の向かって左側を構成し、β2、α1、β3(以上シアン)とβ4、⍦-loop2(以上黄)が向かって右側を構成する。N末端半分とC末端半分は配列の類似度が高く、⍦-loop1と⍦-loop2の立体構造も非常によく似たものとなる。
そこで、国際共同研究グループはこの仮説に基づき、初めに、全く同じアミノ酸配列の繰り返しから成る完全対称性のDPBBの復元を目指しました。まず、現生生物のDPBBの中でも比較的高い対称性を持つ超好熱性古細菌Methanopyrus kandreri[6]のVCPシャペロン[7]のDPBBドメインを出発材料として、理論設計法とコンピュータ計算手法の二つの手法で完全対称性DPBBの設計を目指しました(図3上・中段)。
理論設計法は、出発材料のDPBBにおいてN末端側とC末端側で異なるアミノ酸を、他の古細菌のDPBBドメインを参考に同じアミノ酸に交換していく作業を繰り返すことで、4個の完全対称性DPBBが得られました。一方コンピュータ計算手法では、さまざまな生物が持つ500種余りのVCPシャペロンの配列の比較などから祖先のDPBB構造を推定したり、DPBB構造を安定的に取り得るアミノ酸配列を計算で求める方法で、9個の完全対称性DPBBを設計できました。
以上の合計13個のうち、10個は溶液中できちんとDPBB構造を作ることが分かりました。また、そのうち6個はX線結晶構造解析[8]により詳細な分子構造を決定できました(図3中段)。さらに、一般的なタンパク質の至適温度をはるかに超える80℃付近でも、立体構造を保っているものもありました。この熱耐性が古代DPBBの特徴であるとすれば、全生物の共通祖先が熱水噴出孔[6]などの高温環境下で進化したという仮説と矛盾しない結果が得られたと考えられます。
次に、完全対称型としたDPBBを半分に切った約45個のアミノ酸から成る断片(断片化DPBBペプチド、図3下段)を作って解析したところ、2本のペプチドが組み合わさった二量体となり、2本で一つのDPBB構造を形成していました(図3下段)。つまり、DPBB構造が50個のアミノ酸にも満たない短いペプチドから誕生したことを本研究で初めて実験的に立証しました。
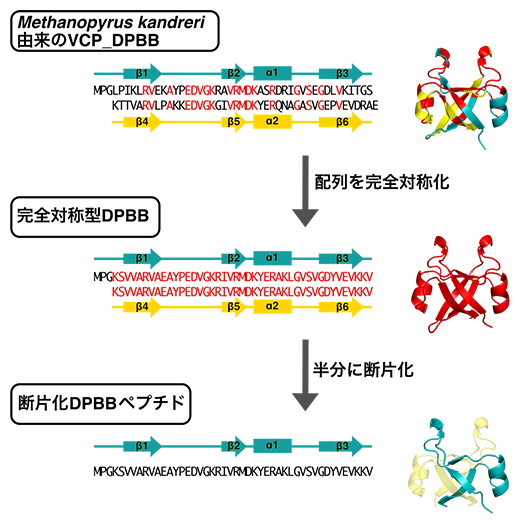
図3 現代のDPBBより古代断片化DPBBペプチドの復元
天然に存在するDPBB(上)、完全対称型DPBB(中)、断片化DPBBペプチド(下)の配列と結晶構造を示す。N末端とC末端それぞれで同じアミノ酸の場合は赤にしている。
興味深いことに、設計した断片化DPBBペプチドの中には、20種類のアミノ酸のうち7種(システイン、トリプトファン、フェニルアラニン、スレオニン、アスパラギン、グルタミン、ヒスチジン)を含まず、たった13種類のアミノ酸で構成されている配列もありました(図4左)。そこでさらに、断片化DPBBペプチドで使用頻度の少ない6種のアミノ酸(メチオニン、イソロイシン、ロイシン、プロリン、セリン、チロシン)を段階的に失くしたタンパク質を設計・解析しました(図4)。
最終的に設計した7種類のアミノ酸だけからなる断片化DPBBペプチドは、溶液中では折り畳まれませんでしたが、結晶を作ることは可能でした。この結晶をX線結晶構造解析で調べたところ、やはり2本のペプチドが組み合わさって一つのDPBB構造を維持していることが分かりました(図4右)。つまり、アラニン、アスパラギン酸、グルタミン酸、グリシン、バリン、リジン、アルギニンのたった7種類のアミノ酸から、DPBB構造が創れることを見いだしました。特に、ペプチド同士の相互作用に重要な疎水性アミノ酸は分子量の小さいバリンとアラニンだけであり、DPBB構造誕生においてロイシン、イソロイシン、メチオニン、フェニルアラニンなどの大きい疎水性アミノ酸[9]は必要ないことが分かりました。
また、使用した7種のアミノ酸はコドン表において右下の領域に分布します(図4右)。コドン表最下段の5種のアミノ酸は他のアミノ酸に比べて単純な構造を持ち、無生物環境下でも誕生しやすいアミノ酸であることが知られています。また、リジンやアルギニンといった正電荷アミノ酸は、負電荷を持つ核酸(RNAやDNA)との相互作用に不可欠であることから、このようなアミノ酸を含むタンパク質合成系とDPBB構造は、RNAやDNAなどの遺伝情報分子とともに、初期生命システムにおいて早い段階に誕生した可能性が高いといえます。これを裏付ける結果として、溶液中で立体構造を取る最も単純な断片化DPBBペプチド(上記7種に加えチロシンを含む8種のアミノ酸で構成)は、二本鎖DNAと特異的な相互作用が認められました。すなわち、古代DPBBは核酸と相互作用するDNA結合タンパク質[10]として機能していた可能性があります。
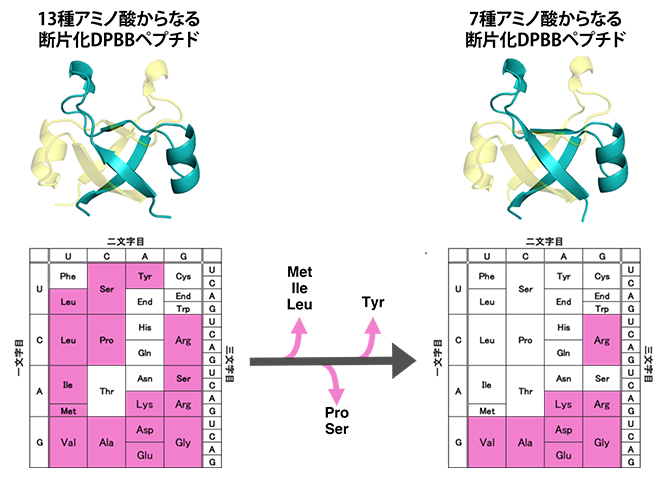
図4 現代のDPBBペプチドから古代断片化DPBBペプチドを復元
13種類のアミノ酸からなる断片化DPBBペプチドの配列から、メチオニン(Met)、イソロイシン(Ile)、ロイシン(Leu)、プロリン(Pro)、セリン(Ser)、チロシン(Tyr)を段階的に除くことで、7種のアミノ酸からなる断片化DPBBペプチドを構築した。残ったアラニン(Ala)、アスパラギン酸(Asp)、グルタミン酸(Glu)、グリシン(Gly)、バリン(Val)、リジン(Lys)、アルギニン(Arg)はコドン表において右下に集中して分布しており、これらのアミノ酸を指定するには、コドンの先頭がA(アデニン)もしくはG(グアニン)であれば十分であることが分かる。
今後の期待
本研究から、短く単純なペプチドであっても生命機能に必須なDPBB構造を作り出せることが分かりました。この結果は、構造・機能を持ったタンパク質の誕生が、これまで考えられてきたよりも容易だった可能性を示しています。
本研究の知見は、タンパク質進化および生命の起源研究において重要な手掛かりになると期待できます。さらに今後、古代の単純なDPBBがどのような機能を持ち得るかを検証することで、セントラルドグマを備えた初期生命がどのように誕生してきたかも解明できるかもしれません。
補足説明
- 1.Double-psi-beta-barrel(DPBB)、αヘリックス、βシート
DPBBは、βバレルと呼ばれる樽(バレル)状の立体構造を持つタンパク質に頻繁に見出される構造の一つ。ギリシャ文字の⍦に似た構造(サイ・ループ・モチーフ)が二つつながっていることからこの名が付けられた。αヘリックスとβシートは、タンパク質中の部分的な短い折り畳み構造を指し、それぞれ、らせん構造、平面構造をとったもの。なお、βシートは複数の直鎖状ペプチドが並んでできたものであり、それぞれのペプチドをβストランドと呼ぶ。前述のサイ・ループ・モチーフは、一つのαヘリックスと3本のβストランドの組み合わせで構成されている。 - 2.ペプチド、N末端、C末端
二つ以上のアミノ酸が、隣接するアミノ基とカルボキシ基で脱水結合(ペプチド結合)した分子をペプチドと呼ぶ。明確な定義はないが、アミノ酸が数個から数十個程度つながったものをペプチド、それ以上の長さの生体分子はタンパク質と呼ぶことが多い。ペプチドやタンパク質中の末端のフリーのアミノ基側をN末端、カルボキシ基側をC末端と呼ぶ。 - 3.コドン表、タンパク質合成、セントラルドグマ
タンパク質の中のアミノ酸の並び方は、そのタンパク質の遺伝子(DNA)の中の塩基配列に対応している。三つの塩基がひとまとまりになって一つのアミノ酸に対応しており、この3塩基の並びを「コドン」、コドンとアミノ酸などの対応関係を示した表をコドン表と呼ぶ。生物が行うタンパク質合成は、DNAの塩基配列をRNAにコピーする「転写」、RNAの配列を読み取りコドン表に従ってアミノ酸をつなげる「翻訳」から成り、この仕組みを「セントラルドグマ」と呼ぶ。 - 4.RNAポリメラーゼ
DNAの塩基配列を読み取って相補的なRNAを合成する酵素で、遺伝子の発現(セントラルドグマ)の第一段階をつかさどる。細胞内では、複数のタンパク質(サブユニット)が集合してできた巨大な複合体として存在する。 - 5.内部対称性
ここでは、一つのタンパク質分子の中に同じ構造が繰り返し存在することで、回転対称など対称性を持った構造が現れること。ただし、本文中にもある通り自然界では、まとまった立体構造を作る長さのアミノ酸配列が一つのタンパク質中に完全な繰り返しで存在することはないため、対称性は擬似的なものとなる。 - 6.超好熱性古細菌Methanopyrus kandreri、熱水噴出孔
80~90℃以上の温度を好んで生育する古細菌を超好熱性古細菌と呼び、中でもMethanopyrus kandreriは100℃以上の温水でも増殖できる。現在の地球上でこのような高熱環境が見られる場所は、地熱による熱水が噴出する海底の亀裂(熱水噴出孔)などがある。 - 7.VCPシャペロン
生体分子の折り畳みを補助して適切な立体構造の形成を促進したり、逆に立体構造を解くなどして生体分子の分解に関わる機能を持つタンパク質をシャペロンと総称する。VCPシャペロンは、タンパク質分解や細胞内輸送など多様な機能を持つシャペロン。VCPはvalosin-containing proteinの略。 - 8.X線結晶構造解析
結晶化させた試料にX線を照射したときの回折パターンから電子密度分布を取得し、分子の立体構造を解析する方法。 - 9.疎水性アミノ酸
生物が利用する20種類のアミノ酸のうち、水に溶けにくい性質(疎水性)持つもの。水溶液中で機能するタンパク質の表面は、水分子と接するため水に溶けやすい(親水性)アミノ酸が多く分布するが、タンパク質の内部は水分子から弾かれた疎水性アミノ酸が集まる(疏水結合)傾向にある。 - 10.DNA結合タンパク質
DNAに結合するタンパク質の総称。DNAの折り畳みに関わるヒストンタンパク質、DNA複製に関わるDNAポリメラーゼ、DNAを鋳型に転写反応を行うRNAポリメラーゼなど、さまざまな機能を担うものがある。
国際共同研究グループ
理化学研究所 生命機能科学研究センター
高機能生体分子開発チーム
チームリーダー 田上 俊輔(たがみ しゅんすけ)
基礎科学特別研究員 八木 創太(やぎ そうた)
構造バイオインフォマティクス研究チーム
チームリーダー ケム・ツァン(Kam Zhang)
訪問研究員 アディティア・クマール・パディ(Aditya Kumar Padhi)
分子配列比較解析チーム
専門職研究員 中川 れい子(なかがわ れいこ)
トゥールーズ連邦大学(仏国)
准教授 デビッド・シモンシーニ(David Simoncini)
博士研究員 イェレナ・ヴチニック(Jelena Vucinic)
トゥールーズ生物工学研究所(仏国)
研究部長 ソフィー・バルベ(Sophie Barbe)
国立農学研究センター(仏国)
研究部長 トーマ・シークス(Thomas Schiex)
研究支援
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、日本学術振興会(JSPS)科学研究費補助金若手研究「単純なペプチドから汎用的な祖先タンパク質フォールドへの進化(研究代表者:八木 創太)」、基盤研究(B)「タンパク質の構造設計の計算方法の研究開発および実験検証(研究代表者:ケム・ツァン)」、基盤研究(B)「初期生命におけるRNA・タンパク質共進化プロセスの再現(研究代表者:田上 俊輔)」およびL'Agence nationale de la recherche(フランス国立研究開発法人)による支援を受けて行われました。また、一部の実験は、高エネルギー加速器研究機構(KEK)およびSPring-8により行われました。
原論文情報
- Sota Yagi, Aditya K. Padhi, Jelena Vucinic, Sophie Barbe, Thomas Schiex, Reiko Nakagawa, David Simoncini*, Kam Y. J. Zhang, and Shunsuke Tagami, "Seven Amino Acid Types Suffice to Create the Core Fold of RNA Polymerase", Journal of the American Chemical Society, 10.1021/jacs.1c05367
発表者
理化学研究所
生命機能科学研究センター 高機能生体分子開発チーム
基礎科学特別研究員 八木 創太(やぎ そうた)
チームリーダー 田上 俊輔(たがみ しゅんすけ)
構造バイオインフォマティクス研究チーム
訪問研究員 アディティア・クマール・パディ(Aditya Kumar Padhi)
チームリーダー ケム・ツァン(Kam Zhang)
分子配列比較解析チーム
専門職研究員 中川 れい子(なかがわ れいこ)
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
