2014年12月17日
理化学研究所
バクテリアの抗生物質耐性を予測する新手法を開発
-少数遺伝子の発現量変化から高精度に抗生物質への耐性を予測-
要旨
理化学研究所(理研)生命システム研究センター多階層生命動態研究チームの古澤力チームリーダー、鈴木真吾研究員らの研究チームは、複数の抗生物質[1]に対して耐性を持つ大腸菌の解析を行い、少数遺伝子の発現量データだけで、抗生物質への耐性を定量的に予測できる新手法を開発しました。この手法によって、バクテリア[2]が抗生物質への耐性を獲得する際、鍵となるのはどの遺伝子の発現量変化なのか、膨大な数の遺伝子から抽出することが可能になります。
複数の抗生物質に耐性を持つ病原菌である「多剤耐性菌[3]」の出現は、世界的な問題となっています。多剤耐性菌の出現を抑えるには、病原菌の進化がどのように起こるかを理解し、耐性獲得をコントロールする手法の開発が必要です。しかし、病原菌の進化の過程は、ゲノムや細胞の状態変化が複雑に絡み合ったものであり、その全貌が理解されているとは言い難いのが現状です。
研究チームは、さまざまな抗生物質を添加した環境で、バクテリアの一種である大腸菌を長期に植え継いで培養することで、生体内で行われる抗生物質に対する耐性獲得の進化プロセスを生体外で再現できる実験システムを構築しました。実験では、どの遺伝子に突然変異が起きるのか、発現量に変化が生じるのかを調べると同時に、各種薬剤に対する耐性の変化を詳細に解析しました。その結果、1つの抗生物質への耐性獲得が、他の抗生物質に対する耐性獲得を引き起こす一方で、別の抗生物質に対しては、耐性の低下を引き起こすことが明らかになりました。また、耐性を獲得した大腸菌では、獲得前の状態と比較して、数千の遺伝子で発現量が変化するものの、どの抗生物質への耐性を獲得するのかは、数個の遺伝子の発現量によって高い精度で予測できることを、統計的解析によって示しました。さらに、耐性株でゲノムに生じた突然変異を調べた結果、全く種類の異なる遺伝子の変異が、類似の機能を持つ遺伝子の発現量の変化を引き起こし、それが抗生物質耐性の獲得につながっていることが示唆されました。
開発した手法により、どの遺伝子がどの抗生物質への耐性獲得に寄与しているかを定量的に解析することが可能となり、耐性獲得を抑制する手法の開発や新規抗生物質の開発への貢献が期待できます。
本研究は、科学研究費補助金新学術領域研究「複合適応形質進化の遺伝子基盤解明」、若手研究A、若手研究B、基盤研究A、などの支援を得て行われました。なお、研究の成果は、英国の科学雑誌『Nature Communications』のオンライン版(12月17日付け:日本時間12月17日)に掲載されます。
背景
抗生物質が効かない病原菌(耐性菌)、特に複数の抗生物質への耐性を持つ多剤耐性菌の出現は、世界的な問題となっています。新しい抗生物質を開発しても、投与を続けることにより、多くの場合においてその抗生物質への耐性菌が出現し、その効果が失われてしまいます。世界保健機関(WHO)は、こうした耐性菌の出現によって、ありふれた感染症や軽度のけがで命を落としかねない「ポスト抗生物質時代」が到来すると警告しています。
抗生物質に耐性を持つ病原菌は、突然変異などによる状態変化と選択の繰り返しである、いわゆるダーウィン進化の過程を経て出現すると考えられています。耐性菌の出現メカニズムを理解し、出現を抑制する手法を開発するためには、耐性菌へと進化する過程で何が起こっているかを明らかにすることが重要です。しかし、耐性獲得の進化過程は、ゲノム配列の変化に加え、細胞の状態変化といった多くの要素が絡み合う複雑な仕組みであるため、全貌が理解されているとはいえないのが現状です。
研究手法と成果
病原菌が抗生物質に対する耐性をどうやって獲得したのか知りたい場合に、病院などで発見された耐性菌が持つ性質(耐性を示す表現型やゲノム配列など)を調べることは広く行われています。しかし、この方法では、どのような過程を経て病原菌が耐性を獲得したのかを明確にすることが難しい場合がありました。そこで研究チームは、抗生物質を添加した環境で、バクテリアの一種である大腸菌を長期に植え継ぐことによって、生体内で起きる抗生物質耐性の進化プロセスを生体外で再現できる実験システムを構築しました。この手法により、どのような状態の大腸菌が、どのような変化を経て耐性を獲得するかを詳細に解析することが可能になります。この研究では、さまざまな作用機序[4]を持つ10種類の抗生物質をそれぞれ添加した環境で90日間の植え継ぎ培養による進化実験を行い、進化前の大腸菌より高い濃度の抗生物質を添加しても増殖が可能な抗生物質耐性株を取得しました(図1)。
これらの耐性株がどのような表現型を持つかを解析するために、1つの抗生物質を添加した環境で得られた耐性株が、他の抗生物質への耐性をどのように変化させるかを系統的に調べました(図2a)。その結果、1つの抗生物質への耐性獲得が、ある抗生物質に対しては耐性を上昇させ、ある抗生物質に対しては耐性の低下を引き起こすことが明らかになりました。この結果から、その耐性に正の相関がある抗生物質の組み合わせ(図2b)や、逆に負の相関がある組み合わせ(図2c)の同定に成功しました。特に後者は、ある抗生物質Aの耐性株は別の抗生物質Bへ感受性を示し、またその逆も成り立つというトレードオフの関係を意味しており、そうした抗生物質の組み合わせを投与することは、耐性株の出現抑制に効果があると期待できます。
次に、この耐性獲得のメカニズムを理解するために、DNAマイクロアレイ[5]を用いて耐性株にどのような遺伝子発現量の変化が生じているかを網羅的に定量しました。その結果、多くの遺伝子において発現量が変化をしていることが分かりましたが、その数が多すぎてどの遺伝子が耐性獲得に寄与しているかを見つけることは困難でした。そこで、回帰分析[6]とクロスバリデーション法[7]を適切に組み合わせて用いることにより、どれだけの数の遺伝子発現量から耐性の変化が予測できるかを解析しました。その結果、7~8個の遺伝子の発現量を組み合わせることによって、さまざまな抗生物質への耐性能を高い精度で予測することに成功しました(図3)。この手法によって、どの遺伝子がどの抗生物質の耐性に寄与しているかを、定量的に評価することが可能となり、耐性獲得メカニズムの抽出が容易となりました。例えば、図2cで示した抗生物質耐性のトレードオフに関しては、電子伝達系[8]に関与する遺伝子の発現量の変化の寄与が大きいことが示され、細胞内外の水素イオンの輸送量の変化が重要な要因になっていることが示唆されました。
少数の遺伝子の発現量から抗生物質への耐性予測が成功したことは、ゲノム、タンパク質、代謝反応といった膨大な要素が関わる抗生物質耐性の獲得について、比較的少数の要素(自由度)によって記述できることを意味します。その変化を引き起こした要因をさらに調べるために、得られた耐性株のゲノム配列がどのように変化しているかを超並列シーケンサー[9]を用いて解析しました。その結果、それぞれの耐性株で数個から十数個程度の突然変異がゲノムに生じていることが明らかになりました。一方で、遺伝子発現量とゲノムの変異の対応を解析したところ、類似した発現量変化が起こっている耐性株の間で、必ずしも類似した変異が生じているわけではなく、さまざまな変異が似通った遺伝子発現量の変化を引き起こし、それが抗生物質耐性の獲得につながっていることが示唆されました。
今後の期待
今回の成果は、遺伝子発現量の解析によって病原菌における抗生物質耐性の獲得過程を解析する際の新たなアプローチになります。今後、発現量の変化とゲノムの変異などを統合的に解析する手法を応用していくことにより、耐性獲得の進化プロセスを予測・制御する手法の開発や、新規抗生物質の発見に貢献すると期待できます。
原論文情報
- Shingo Suzuki, Takaaki Horinouchi, and Chikara Furusawa, "Prediction of antibiotic resistance by gene expression profiles", Nature Communications, 2014, doi: 10.1038/ncomms6792.
発表者
理化学研究所
生命システム研究センター 生命モデリングコア 多階層生命動態研究チーム
チームリーダー 古澤 力 (ふるさわ ちから)
お問い合わせ先
理化学研究所 生命システム研究センター
広報担当 川野武弘 (かわの たけひろ)
Tel: 06-6155-0113 / Fax: 06-6155-0112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.抗生物質
微生物などの増殖や生理機能を阻害する化学物質のこと。感染症の治療などに広く用いられる。 - 2.バクテリア
原核生物と呼ばれる微生物のこと。真核生物と異なり、核などの細胞内の複雑な構造を持たない。バクテリアの仲間には乳酸菌に代表される人に有益な善玉菌が含まれる一方で、食中毒やその他の重篤な病状を引き起すボツリヌス菌や破傷風菌をはじめ、さまざまな病原菌が含まれる。大腸菌は人や動物の腸内に共生し、ほとんどは無害だが、中にはO-157など強い病原性を有する大腸菌もあり、病原大腸菌と呼ばれる。本研究では病原性のない大腸菌を用いている。 - 3.多剤耐性菌
異なる作用を持つ複数の抗生物質に耐性を持つ微生物のこと。抗生物質による治療が難しい場合があるため、その出現が医学上の大きな問題となっている。 - 4.作用機序
抗生物質が効果を発揮するために必要な相互作用。今回の研究で用いた抗生物質の作用機序には、細胞壁の合成阻害、タンパク質の合成阻害、DNA複製の阻害などがある。 - 5.DNAマイクロアレイ
細胞内の遺伝子発現量などを定量するために、基板上にプローブと呼ばれる多数の短いDNA断片を固定した分析機器。蛍光標識したサンプルDNAあるいはRNAをプローブと相互作用させることにより、プローブと相補的な配列を持つDNAあるいはRNAの量を測定する。高密度に配列の異なるプローブを配置することにより、膨大な遺伝子の発現量などを同時に定量できる。 - 6.回帰分析
ある1つの量(目的変数)を別の量(説明変数)を用いて予測するための関係式を導く統計的手法。今回の研究では、抗生物質への耐性能を目的変数、遺伝子発現量を説明変数として予測のための関係式を構築した。 - 7.クロスバリデーション法
統計学において標本データを分割し、その一部のデータを用いて統計的解析を行い、残りのデータを用いてその解析の妥当性の検証や確認を行う手法のこと。今回の研究では、一部の発現量データを用いて耐性を予測するための関係式を構築し、残りのデータを用いてその予測精度の検証を行った。 - 8.電子伝達系
生物が酸素を用いた好気呼吸を行うときに用いる一連の代謝反応の総称。ニコチンアミドアデニンジヌクレオチド(NADH)などの還元力を利用して、細胞内外の水素イオンの濃度勾配を作り出す。この水素イオンの濃度勾配は、ATP合成酵素によるATP合成に利用される他、多剤排出ポンプによる抗生物質の排出にも利用される。 - 9.超並列シーケンサー
DNAポリメラーゼなどの酵素によるDNAの伸張反応を、蛍光や発光などによって検出して膨大な数の塩基配列を超並列的に決定できるシーケンサーの総称。次世代シーケンサーとも呼ばれる。
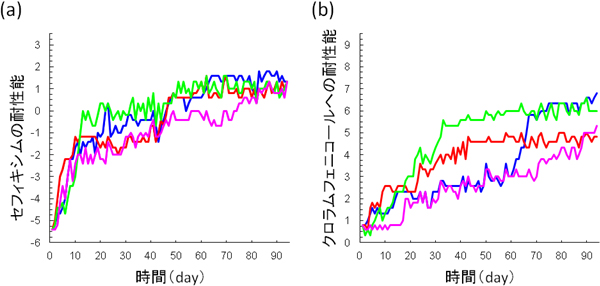
図1 抗生物質添加環境での進化実験結果の例
(a)はセフィキシム(細胞壁合成阻害剤)、(b)はクロラムフェニコール(タンパク質合成阻害剤)を添加した環境での進化実験結果を示す。縦軸は、菌の増殖が阻害される最小の抗生物質濃度(mg/L)を対数変換したもので、この値が大きいほど高い耐性能を持つ。進化過程の再現性や多様性を解析するために、それぞれの抗生物質について4つの独立系列での進化実験を行った。
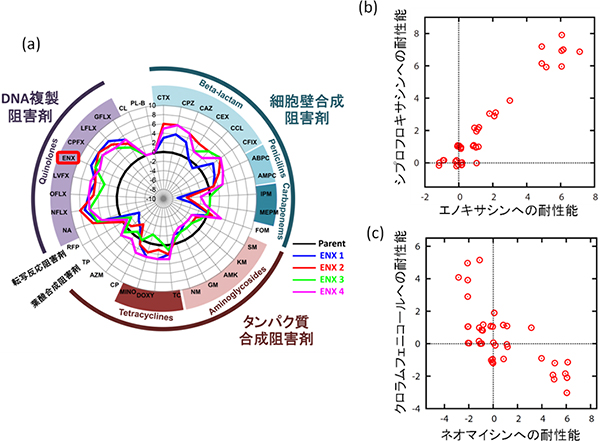
図2 さまざまな抗生物質への耐性能の解析
- (a) エノキサシン(ENX)耐性株が、他のさまざまな抗生物質に対してどのように耐性の上昇・低下を変化させたかを定量した例。放射線軸が進化前の株に対する耐性能の対数比を示す。太い黒線で示した円は、進化前の株の耐性能を表し、アルファベット2~4文字(CTX, CPZなど)は抗生物質の略号を表している。各色の線が黒い円よりも外側にある場合は、エノキサシン耐性を獲得することによって対応する抗生物質への耐性が上昇したことを表し、内側に入ったときは、逆に耐性が低下したことを意味する。こうした解析を、10種類の抗生物質の耐性株についてそれぞれ行った。
- (b) シプロフロキサシン(CPFX)とエノキサシン(ENX)(ともにDNA複製阻害剤)の耐性能の関係。ここでの正の相関は、シプロフロキサシンの耐性を獲得した株はエノキサシンへの耐性が上昇し、その逆も成り立つことを意味する。
- (c) クロラムフェニコール(CP)とネオマイシン(NM)(ともにタンパク質合成阻害剤)の耐性能の関係。ここでの負の相関は、クロラムフェニコールの耐性を獲得した株はネオマイシンに対して耐性の低下を示し、その逆も成り立つというトレードオフの関係を示す。
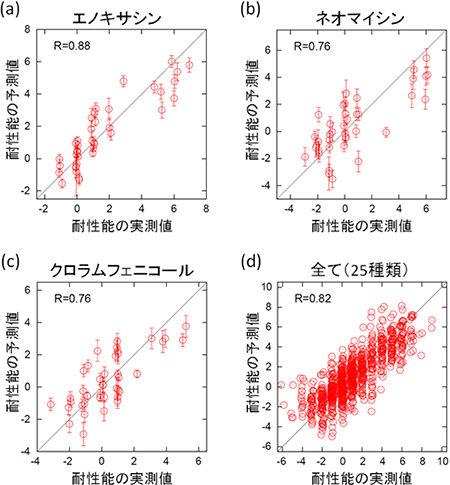
図3 遺伝子発現量による耐性能の予測
それぞれの軸は進化前の株に対する耐性能の対数比を表し、横軸が実測値、縦軸が予測値を示す。発現量から耐性能を予測するモデル式を構築し、その予測精度が最大となる遺伝子数を探索したところ、7~8個の遺伝子の組み合わせが最も高い予測力を持つことが示された。図では、acrB, ompF, cyoC, pps, tsx, oppA, folA, pntBの組み合わせによる予測例を示している。
