2019年2月8日
理化学研究所
東京大学
日本医療研究開発機構
コンパクトなDNAをスムーズに転写する仕組み
-ヌクレオソームを乗り越える転写伸長複合体の構造解析-
理化学研究所(理研)生命機能科学研究センター転写制御構造生物学研究チームの関根俊一チームリーダー、江原晴彦研究員、東京大学定量生命科学研究所の胡桃坂仁志教授、鯨井智也助教らの共同研究チーム※は、真核細胞の遺伝子発現を担う「RNAポリメラーゼ[1]II(RNAPII)」が、ヒストン[2]に巻きついたDNA(ヌクレオソーム[2]DNA)をスムーズにほどきながら塩基配列を読み取り、RNAに転写する仕組みを解明しました。
本研究成果は、DNAを核内にコンパクトに収納しつつ、DNAをスムーズに転写することが真核細胞でどのように両立しているのかという生物学上の大きな謎に応えるものであり、今後、転写伸長の制御やその破綻による疾患メカニズムについての研究が発展するものと期待できます。
真核生物のDNAは、ヒストンタンパク質と結合して「ヌクレオソーム構造」を形成し、さらに複数のヌクレオソームが数珠状に連なった「クロマチン[2]」と呼ばれる高次構造をとって、細胞核内に収納されています。メッセンジャーRNA(mRNA)の転写を担う酵素RNAPIIにとって、ヌクレオソーム構造は転写伸長の大きな障壁となることが分かっています。
今回、共同研究チームは、RNAPIIとともに働く複数のタンパク質(転写伸長因子)が協調してヌクレオソーム障壁を解除し、RNAPIIがヌクレオソームDNAをスムーズに転写する仕組みを「クライオ電子顕微鏡[3]」観察により世界に先駆けて明らかにしました。
本研究は、米国の科学雑誌『Science』(2月15日号)の掲載に先立ち、オンライン版(2月7日付け:日本時間2月8日)に掲載されます。
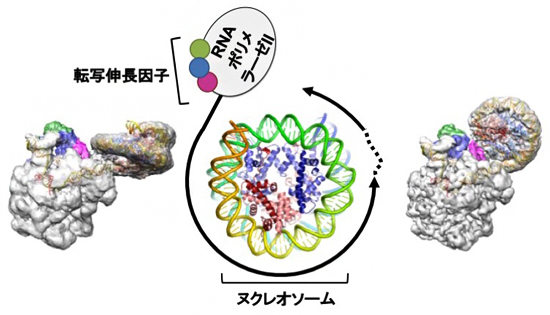
図 ヌクレオソームDNAの転写(中央の模式図)を捉えたクライオ電子顕微鏡像(左と右)
※共同研究チーム
理化学研究所 生命機能科学研究センター
転写制御構造生物学研究チーム
チームリーダー 関根 俊一(せきね しゅんいち)
研究員 江原 晴彦(えはら はるひこ)
タンパク質機能・構造研究チーム
チームリーダー 白水 美香子(しろうず みかこ)
東京大学 定量生命科学研究所 クロマチン構造機能研究分野
教授 胡桃坂 仁志(くるみざか ひとし)
助教 鯨井 智也(くじらい ともや)
大学院生 藤野 優佳(ふじの ゆうか)
※研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金 新学術領域研究(研究領域提案型)「ヌクレオソーム高次構造とダイナミクスの解析によるクロマチン潜在能の解明(代表:胡桃坂仁志)」「再構成ヌクレオソームを用いた動的クロマチン構造の解明(代表:同)」、同基盤研究(B)「転写因子によるRNAポリメラーゼの構造変化と転写制御のメカニズム(代表:関根俊一)」、日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業 創薬等先端技術支援基盤プラットフォーム(BINDS)「エピジェネティクス研究と創薬のための再構成クロマチンの生産と性状解析(代表:胡桃坂仁志)」および「統合的構造解析に向けた高難度複合体の生産支援と高度化(代表:白水美香子)」の支援を受けました。
背景
生命の設計図である遺伝情報は、細胞の核内にあるDNAに塩基の配列として保存されています。生物は遺伝子、つまりDNA上で遺伝情報が記された領域を、適切なときに適切な器官や組織、細胞で働かせることにより、個体を形成し、生命機能を維持しています。
遺伝子を働かせるためには、遺伝子のDNA配列がまずRNAにコピー(転写)される必要があります。DNAからRNAへの転写をつかさどるRNAポリメラーゼは、全ての生物にとって必須の酵素です。ヒトを含め真核生物には3種類のRNAポリメラーゼが存在し、そのうち「RNAポリメラーゼII(RNAPII)」がタンパク質をコードするメッセンジャーRNA(mRNA)の転写を担当しています。RNAPIIによる転写は、1秒間に数十塩基のRNAを正確に合成する極めてダイナミックな反応です。
真核生物のDNAは、核内では「クロマチン」と呼ばれる高次構造をとって収納されています。クロマチンはDNAとタンパク質の複合体であり、その基本単位は、ヒストンタンパク質の周りにDNAが約1.7回(145~147塩基対)巻きついた「ヌクレオソーム」です。クロマチンの高次構造の変化は、転写制御に関わっていることが知られていますが、ヌクレオソーム構造そのものは転写の進行に対する大きな障壁となっていると考えられてきました。事実、共同研究チームが試験管内で再構成したヌクレオソームとRNAPIIを反応させたところ、DNAを転写する途中でRNAPIIがヌクレオソーム上の複数の位置で停止する様子が観察されました注1)。
一方、実際の細胞核内にはRNAPIIの転写伸長を補助するさまざまなタンパク質(転写伸長因子)が存在し、ヌクレオソームDNAのスムーズな転写を実現していると考えられています。しかし、転写伸長因子がどのようにヌクレオソーム障壁を解除し、ヌクレオソームDNAの転写を可能にしているかは、よく分かっていませんでした。
関根チームリーダーらは先行研究において、RNAPIIとDNA、RNA、および3種類の転写伸長因子を組み合わせた「転写伸長複合体」を試験管内で再構成し、その構造を解明しました注2)。さらに2018年には、胡桃坂教授らと共同で、RNAPIIがヌクレオソームDNAを転写する仕組みを世界に先駆けて発表しました注1)。今回はそれらを組み合わせ、再構成したヌクレオソームにRNAPIIと転写伸長因子を反応させ、細胞核内での転写伸長反応を正確に再現することを試みました。
- 注1) 2018年10月5日プレスリリース「真核生物での遺伝子読み取りの仕組みを解明」
- 注2) 2017年8月4日プレスリリース「転写中のRNAポリメラーゼIIの構造を解明」
研究手法と成果
共同研究チームはまず、上記の再構成系におけるRNAの合成効率を解析した結果、転写伸長因子であるSpt4[4]とSpt5[4]の複合体(Spt4/5)とElf1[5]が共存したときに、ヌクレオソーム上でのRNAPIIの停止頻度が低下し、ヌクレオソームDNAを最後まで転写したRNAの量が劇的に増大することが分かりました(図1)。
次に、ヌクレオソームDNAの転写におけるSpt4/5とElf1の役割をさらに詳しく調べるため、転写伸長因子を結合したRNAPIIがDNAを転写する際の構造を「クライオ電子顕微鏡」で観察しました。その結果、SHL(–5)とSHL(–1)の2カ所において一時停止した状態の複合体の構造を捉えることができました。このとき、Spt4/5とElf1は一直線に並んだ状態で、RNAPIIとヌクレオソームの間に割り込むように位置していました(図2)。
詳細な構造解析の結果、Spt4/5とElf1の存在により、①RNAPIIとヌクレオソームが直接接触し、安定な停止状態に陥るのを防ぐ、②DNAとヒストンの間の結合を弱め、DNAをヒストンから引き剥がしやすくするとともに再結合を抑制する、③ヌクレオソームがRNAPIIに対して角度を変える動きを促進し、RNAPIIが転写の停止箇所を乗り越えて前進するのを助けるなど、ヌクレオソーム障壁を低下させ、RNAPIIの進行を促進する仕組みが明らかになりました。
今後の期待
本研究では、細胞核内での転写伸長反応を正確に再現した再構成実験を行い、クライオ電子顕微鏡で解析することに成功しました。RNAPIIはヌクレオソームの複数箇所で停止しやすいことが分かっていましたが、本研究から、転写伸長因子が各停止箇所でのヌクレオソーム障壁を低減し、RNAPIIの進行を促進する仕組みが明らかとなりました。
細胞内では、転写伸長因子に加えてヒストンシャペロン[6]やリモデリング因子[7]、ヒストン修飾[8]などが複雑に連携して巧妙に制御された転写が実現されています。今回確認されたヌクレオソームDNAが途中までほどかれた状態(SHL(–5)とSHL(–1))は、これらの因子の作用に重要な中間体として機能している可能性があり、今後のエピジェネティクス[8]や転写制御の研究を飛躍させる重要な基礎となることが期待できます。
RNAPII転写伸長複合体やヌクレオソームのようなDNAとタンパク質からなる巨大複合体は、細胞内の自然な環境を反映した構造解析の難度が非常に高く、これまで取り残されていた創薬対象の一つです。本研究で得られた知見から、今後、転写制御やクロマチン構造の破綻による疾患や老化のメカニズムについての研究が発展するものと期待できます。
原論文情報
- Haruhiko Ehara, Tomoya Kujirai, Yuka Fujino, Mikako Shirouzu, Hitoshi Kurumizaka and Shun-ichi Sekine, "Structural insight into nucleosome transcription by RNA polymerase II with elongation factors", Science, 10.1126/science.aav8912
発表者
理化学研究所
生命機能科学研究センター 転写制御構造生物学研究チーム
チームリーダー 関根 俊一(せきね しゅんいち)
研究員 江原 晴彦(えはら はるひこ)
東京大学 定量生命科学研究所 クロマチン構造機能研究分野
教授 胡桃坂 仁志(くるみざか ひとし)
助教 鯨井 智也(くじらい ともや)
お問い合わせ先
理化学研究所 生命機能科学研究センター センター長室 報道担当
山岸 敦(やまぎし あつし)
Tel: 078-304-7138 / Fax: 078-304-7112
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
東京大学 定量生命科学研究所 総務チーム
Tel: 03-5841-7813 / Fax: 03-5841-8465
E-mail:soumu [at] iam.u-tokyo.ac.jp
※上記の[at]は@に置き換えてください。
産業利用に関するお問い合わせ
AMED事業に関すること
日本医療研究開発機構 創薬戦略部 医薬品研究課
Tel: 03-6870-2219 / Fax: 03-6870-2244
E-mail:20-DDLSG-16 [at] amed.go.jp
※上記の[at]は@に置き換えてください。
補足説明
- 1.RNAポリメラーゼ
DNAの塩基配列を読み取って、相補的なRNAを合成する酵素で、遺伝子発現(セントラルドグマ)の第一段階をつかさどる生命活動に必須の酵素。複数のタンパク質(サブユニット)が集合してできた巨大な複合体で、細菌からヒトまで共通した“カニのはさみ”のような形をしている。中央にできた溝にDNAを挟み込み、およそ10塩基対のDNAをほどいて「転写バブル」を形成し、一方の鎖を鋳型にしてRNAを合成する。 - 2.ヒストン、ヌクレオソーム、クロマチン
DNAを巻き付けることで、長大なDNAを核内に納めるタンパク質をヒストンという。代表的なヒストンはH1、H2A、H2B、H3、H4の5種類があり、H2A、H2B、H3、H4の4種類(コアヒストン)が二つずつ集まってヒストン8量体を形成する。ヒストン8量体の周りにDNAが巻き付いた構造をヌクレオソームと呼び、真核生物においてヌクレオソームを基本単位とするゲノムDNAとタンパク質の高次複合体をクロマチンと呼ぶ。 - 3.クライオ電子顕微鏡
タンパク質などの生体試料を観察するために開発された電子顕微鏡。タンパク質などの試料を含んだ溶液を薄く展開し、液体エタン(-183℃~-160℃)中で急速凍結して試料をごく薄い氷の層に閉じ込めた上、液体窒素温度(-196℃)で電子顕微鏡により観察する。タンパク質試料を生理的な(細胞の状態に近い)条件で観察できる、低温のため試料の電子線による損傷が軽減される、といった利点がある。近年著しい発展を見せている構造解析技術であり、2017年のノーベル化学賞の対象ともなった。
- 4.Spt4、Spt5
Spt4とSpt5は、複合体(Spt4/Spt5)として機能する転写伸長因子。Spt4は真核生物と古細菌に、Spt5は真核生物と古細菌だけでなくバクテリアにも保存されている。転写中に起こるRNAPIIの休止を抑制し、プロセッシブな転写的伸長を保証する。高等真核生物では、DSIFという名で知られており、転写のON/OFF制御において中心的な役割を果たしている。また、CTRと呼ばれるC末端のフレキシブルな領域を持っており、これを介して複数の他のタンパク質をRNAPIIに呼び込む役割も果たす。
- 5.Elf1
真核生物と古細菌の一部に保存された転写伸長因子。Spt4やSpt5、TFIISなどの転写伸長因子と合成致死性を示す因子として同定された。細胞内で転写中のRNAPIIに共存していることが知られているほか、クロマチン構造の維持への関与も見いだされている。
- 6.ヒストンシャペロン
「シャペロン」は、新生タンパク質が正常な立体構造をとるのを補助したり、変性したタンパク質を認識して正常な構造に戻す、あるいは除去する機能を持つ因子の総称。ヒストンシャペロンは、特にヒストンとDNAの結合や解離に関わる因子をさす。 - 7.リモデリング因子
クロマチンの構造変化を引き起こす因子の総称。遺伝子発現の制御機構。 - 8.ヒストン修飾、エピジェネティクス
ヒストンタンパク質は、アセチル基転移酵素と脱アセチル化酵素による可逆的なアセチル化修飾と、メチル基転移酵素と脱メチル化酵素による可逆的なメチル化修飾を受ける。ヒストンのアセチル化/メチル化の状態は、DNAの可逆的なメチル化修飾とともに遺伝子発現の制御に大きな役割を果たしており、これらの化学修飾が関わる生命現象や、それを対象とする研究分野をエピジェネティクスと呼ぶ。
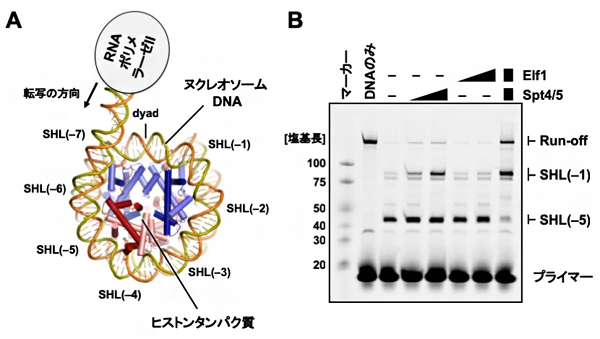
図1 RNAポリメラーゼII(RNAPII)、転写伸長因子、ヌクレオソームの再構成
- (A) 本研究での再構成実験の模式図。RNAPIIと転写伸長因子は酵母由来、ヌクレオソームはヒト由来のものを用いた。SHL(superhelical location)はヌクレオソーム上の位置に番号を振ったものである。SHL(-7)からSHL(-5)までRNAPIIが進むと、DNAが約20塩基対ほどけた状態、SHL(-1)までなら約60塩基対ほどけた状態となる。dyadはヌクレオソームの中間点を示す。本実験では、ヌクレオソームの外でRNAPIIが結合し転写を開始するDNA領域(53塩基対)に、ヒストンに巻きつくヌクレオソームDNA(145塩基対)をつなげた人工DNAを用いた。
- (B) 再構成実験で得られた転写産物(RNA)の長さを電気泳動により解析した。Elf1、Spt4/5をそれぞれ単独に加えた場合(左から4番目~7番目のレーン)に比べて、両者が共存すると(右端のレーン)、RNAPIIのSHL(-5)での停滞が抑えられ、SHL(-1)を通過してヌクレオソームDNA全長を転写したRNA産物(Run-off)が著しく増加することが分かった。
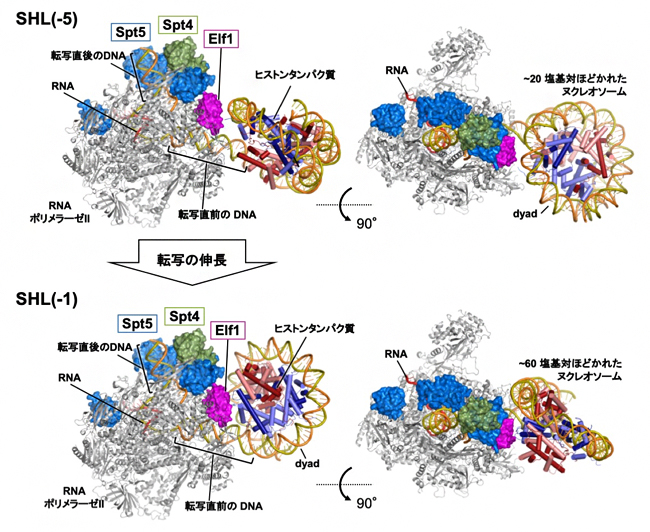
図2 転写伸長中のRNAPII、転写伸長因子、ヌクレオソームの立体配置
クライオ電子顕微鏡により観察された立体構造。上段はDNAがSHL(-5)まで(約20塩基対)、下段はSHL(-1)まで(約60塩基対)ほどけた状態。左図はRNAPIIの位置を基準にそれぞれの立体配置を示しており、右図はその90°回転図で転写伸長因子の側から見た場合。転写の伸長に伴い、RNAPIIに対するヌクレオソームの角度が変化している。またSpt4/5とElf1は一直線に並んだ状態で、RNAPIIとヌクレオソームの間に割り込むように位置しているのが分かる。この構造により、スムーズな転写が可能になる。
