理化学研究所(理研)生命医科学研究センタートランスクリプトーム研究チームのピエロ・カルニンチチームリーダー、ポリン・ロッブ特別研究員らの国際共同研究グループは、血液がんである慢性リンパ性白血病(CLL)[1]患者の臨床経過リスクを層別化する方法を提示しました。
本研究成果は、CLL患者のより個別化された治療につながると期待できます。
CLLはBリンパ球[1]が異常に増える血液のがんであり、中高年に好発します。これまでの研究で、CLLの臨床経過は患者ごとに異なり、特定の治療法に対する患者の反応も異なることが分かっています。
今回、国際共同研究グループは全ゲノムシークエンス解析[2]を用いて、CLLゲノム全体におけるDNA変異を系統的に特徴付けることにより、異なるDNA変異パターンをもつ五つの新しいCLLサブグループを発見しました。これらのCLLサブグループは患者の臨床経過とも関連していました。慢性リンパ性白血病に関するゲノム解析としては世界最大規模であり、全ゲノムシーケンス解析をがんの分類に適用する重要性を示したものといえます。
本研究は、科学雑誌『Nature Genetics』オンライン版(11月4日付)に掲載されました。
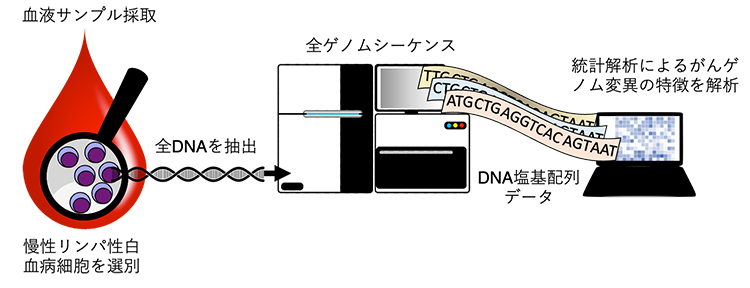
本研究の概要図
背景
慢性リンパ性白血病(CLL)は、Bリンパ球が異常に増える血液のがんで、中高年に好発します。CLLに対する治療は近年進歩してきていますが、依然として治癒は困難です。これまでの研究で、CLLの臨床経過は患者ごとに異なり、特定の治療法に対する効果もそれぞれの患者で異なることが分かっています。
基本的に、がんはDNA変異によって引き起こされ、その変異は治癒しなければ一生涯にわたって発生します。CLL患者ごとに臨床経過が異なる理由の一つは、それぞれの患者で異なる白血病細胞のDNA変異のためだと考えられています。しかしこれまで、白血病細胞のDNA変異は十分に解明されていませんでした。
現在、特定の治療法に対する患者の治療効果を予測する手法は、がん抑制遺伝子TP53などがんDNAの単一な変異に焦点を当てているため、個々の患者における治療効果を正確に予測することはできません。そこで国際共同研究グループは、「がんの後天的なDNA変異を一度に全て調べることで、治療効果予測の精度を高めることはできないか」と考え、研究を進めました。
研究手法と成果
国際共同研究グループは、英国ゲノミクス・イングランドの10万ゲノムプロジェクト[3]に参加するCLL患者485人からがん組織(血液)と正常組織(唾液)を採取し、全ゲノムシーケンス解析を行いました。DNAの配列アラインメント[4]とDNA変異同定のための統計的アルゴリズムを用いて、得られた全ゲノムシーケンスデータのがん組織と正常組織の塩基配列を比較しました。その結果、既知および新規のDNA変異、染色体の構造異常、変異シグネチャー[5]など、全ゲノムにわたりCLLに関連するDNA変異を特定することに成功しました(図1a)。
次に、がんを促進する既知のDNA変異を教師データとして学習させたアルゴリズムを用いて、がんを進行させる可能性のあるDNA変異を予測した結果、既知のCLLのドライバー[6]を含む186個のCLLドライバーを同定しました(図1b)。このドライバーの中にはタンパク質をコードする領域だけではなく、遺伝子の調節に関わる非コード制御因子におけるDNA変異、総変異数、染色体テロメア[7]の長さなど、これまで発見されていなかったCLLドライバーが含まれます。
さらに、これら186個のCLLドライバーに機械学習[8]アルゴリズムを適用し、CLLに関連するDNA変異の類似性により患者ゲノムデータを五つのCLLサブグループに分類しました。これらのCLLサブグループが、特定の化学療法および免疫療法に対する臨床経過に対応しているか調べたところ、実際の臨床経過と関連していることが確認できました。患者の18%が含まれる二つのサブグループ(高リスク)は治療完了後早期に再発するリスクが高く、また患者の26%が含まれる一つのサブグループ(低リスク)は治療後に長期寛解を示すことが明らかになりました(図1c)。
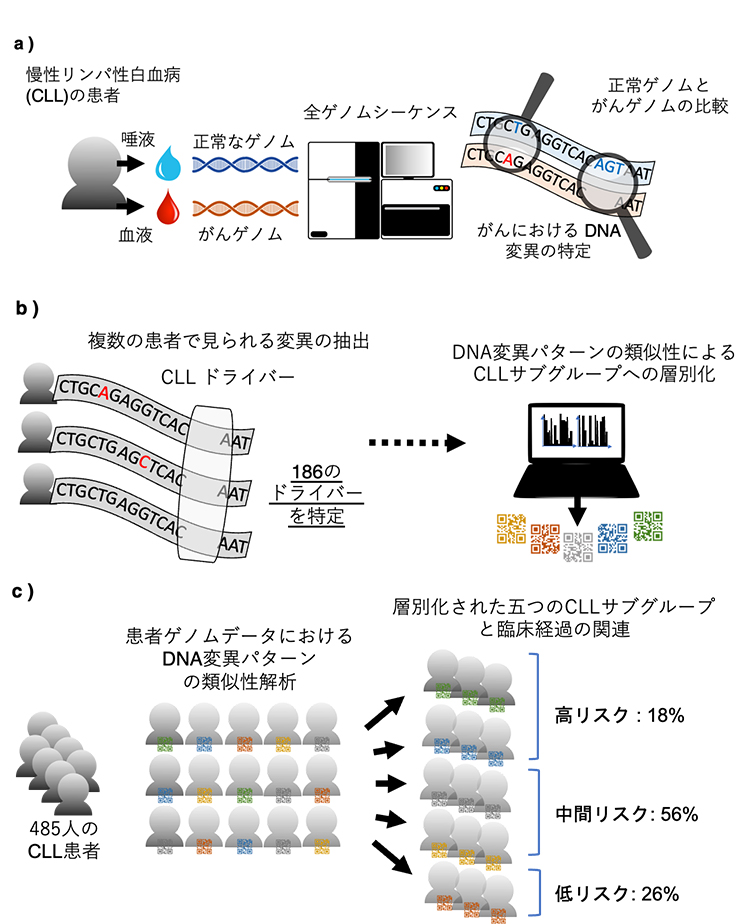
図1 全ゲノムシーケンス解析による患者の新しい層別化手法
- a)全ゲノムシーケンス解析によるDNA変異の特定。
- b)CLLに関連するDNA変異(186のCLLドライバー)の特定および変異パターンの解析により患者ゲノムデータを五つのCLLサブグループへ層別化。
- c)CLL患者をDNA変異パターンの類似性解析により層別化、五つのCLLサブグループと実際の臨床経過区分との関連。
今後の期待
本研究を用いれば、CLL患者を五つのCLLサブグループに層別化して、この層別化に対応する最適な治療を最初に受けることができ、副作用の可能性がある不必要な治療を省くことができます。また、新しい標的療法の臨床試験に最も適した患者の選択にも役立ちます。今後、本研究において解析しなかった新たな標的療法を受けた患者のデータについて追加解析をすることで、効果的な治療法の予測精度を上げる可能性もあります。
また、本研究により、CLLの原因となる可能性がある新たなドライバーが発見されました。今後、これらのドライバーに関する研究が進むことにより、CLLの発症・進行のメカニズムが明らかになり、新しい治療薬の開発につながることが期待されます。
さらに、この研究は、CLLだけではなく、他の多くの種類のがんに応用できる可能性があります。
補足説明
- 1.慢性リンパ性白血病(CLL)、Bリンパ球
慢性リンパ性白血病は中高年に好発する血液のがんで、骨髄のBリンパ球で発生し、血液中に広がる。通常はゆっくりと進行し重度の症状を示すことはないが、一部の患者では、深刻で進行の速い症状を示し、治療を要する。Bリンパ球は白血球の20~40%を占める免疫細胞で、主に抗体を作ってウイルスなどを排除する働きをする。CLLはchronic lymphocytic leukemiaの略。 - 2.全ゲノムシークエンス解析
個人やがん細胞の全ゲノム情報を解読し、塩基配列の違いや変化を同定することを全ゲノムシークエンス解析という。がん細胞の場合は、がんのDNAと同一患者由来の正常DNAの全ゲノムシーケンス解析を行い、その差分を調べる。 - 3.10万ゲノムプロジェクト
希少疾患、がん、感染症の患者および家族の10万全ゲノムシーケンス解析を行うゲノミクス・イングランド社が運営する英国政府のプロジェクト。病気の起源を理解し、患者の予防、診断、治療を改善するのに役立つよう、大規模な集団でゲノムおよび臨床データの豊富な情報を収集することを目的とする。 - 4.配列アラインメント
DNAやRNA、タンパク質の配列(一次構造)の類似した領域を特定できるように、複数の塩基やアミノ酸の配列の中から共通部分を抽出し、お互いを整列させる作業を指す。 - 5.変異シグネチャー
がん細胞に発生するさまざまな変異は、その要因によって異なるパターンを示す。そのパターンを変異シグネチャーと呼ぶ。 - 6.ドライバー
さまざまながんの発生過程において、DNA変異によって細胞機能や発現が変化することで、発がんやがんの進展と直接的に関連する要素のこと。 - 7.テロメア
6塩基の繰り返し配列からなる染色体の末端部にある構造。ゲノム複製や老化に重要であると考えられている。 - 8.機械学習
膨大なデータをコンピュータに入力し、その中にある既知の特徴を繰り返しコンピュータに学習させるか、もしくはデータそのものからコンピュータに規則性を発見させることで、未知のデータに対する解答を自動で得る手法。
国際共同研究グループ
理化学研究所 生命医科学研究センター
トランスクリプトーム研究チーム
特別研究員 ポリン・ロッブ(Pauline Robbe)
チームリーダー ピエロ・カルニンチ(Piero Carninci)
遺伝子制御回路研究チーム
客員研究員 アニカ・プラブ(Anika V Prabhu)
オックスフォード大学(英国)
チームリーダー Anna Schuh
博士研究員 Kate E Ridout
研究員 Dimitrios V Vavoulis
ロンドン大学癌研究所(英国)
チームリーダー Richard S Houlston
博士研究員 Ben Kinnersley
リバプール大学(英国)
チームリーダー Andrew R Pettitt
イルミナUK(英国)
チームリーダー Mark Ross
バルセロナ大学(スペイン)
チームリーダー José I Martin-Subero
研究員 Marti Duran-Ferrer
セント・ジェームズ大学病院(英国)
チームリーダー Peter Hillmen
ゲノミクス・イングランド(英国)
所長 Mark J Caulfield
その他、40名の主著者と二つの研究コンソーシアム、100名近くの研究者による巨大プロジェクトによる成果です。
研究支援
本研究は、日本学術振興会(JSPS)外国人特別研究員制度の助成を受けて行われました。
原論文情報
- Pauline Robbe, Kate E Ridout, Dimitrios V Vavoulis, Helene Dréau, Ben Kinnersley, Nicholas Denny, Daniel Chubb, Niamh Appleby, Anthony Cutts, Alex J Cornish, Laura Lopez Pascua, Ruth Clifford, Adam Burns, Basile Stamatopoulos, Maite Cabes, Reem Alsolami, Pavlos Antoniou, Melanie Oates, Doriane Cavalieri, Jane Gibson, Anika V Prabhu, Ron Schwessinger, Daisy Jennings, Terena James, Uma Maheswari, Martí Duran-Ferrer, Piero Carninci, Samantha JL Knight, Robert Månsson, Jim Hughes, James Davies, Mark Ross, David Bentley, Jonathan C Strefford, Stephen Devereux, Andrew R Pettitt, Peter Hillmen, Mark J Caulfield, Richard S Houlston, José I Martín-Subero, Anna Schuh, "Whole-genome sequencing of chronic lymphocytic leukaemia identifies subgroups with distinct biological and clinical features" Nature Genetics, 10.1038/s41588-022-01211-y
発表者
理化学研究所
生命医科学研究センター トランスクリプトーム研究チーム
特別研究員 ポリン・ロッブ(Pauline Robbe)
チームリーダー ピエロ・カルニンチ(Piero Carninci)
 ポリン・ロッブ
ポリン・ロッブ
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
※上記の[at]は@に置き換えてください。
