理化学研究所(理研)バイオリソース研究センター iPS細胞高次特性解析開発チームの林 洋平 チームリーダーらの研究チームは、ヒトiPS細胞[1]に対する高効率な蛍光タンパク質導入法を開発しました。
本研究成果は、ヒトiPS細胞を用いた細胞・発生生物学的研究、創薬におけるタンパク質発現を指標とした薬剤候補評価系の構築、再生医療に向けた細胞製造法の確立に貢献すると期待できます。
生きた細胞の状態を可視化するために、指標(マーカー)となる重要な遺伝子発現を、蛍光または発光タンパク質(レポーター)を用いて可視化する研究開発が広く行われています。特にゲノムに直接蛍光タンパク質を組み込むこと(ノックイン)は非常に有効ですが、その効率は低くとどまっています。今回、研究チームは、うまく組み込まれなかった細胞を除去するシステムを開発することにより、ヒトiPS細胞の高効率なノックインを達成するとともに、数々のマーカー遺伝子の蛍光レポーターiPS細胞株群を作製しました。
本研究は、科学雑誌『Cell Reports Methods』オンライン版(12月11日付:日本時間12月12日)に掲載されました。
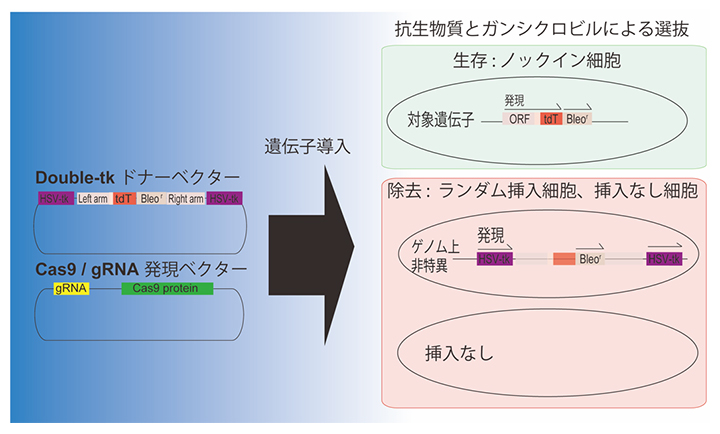
本研究で開発されたDouble-tkドナーシステムの概要
背景
ヒトiPS細胞などの多能性幹細胞を特定の細胞種に分化させるさまざまな方法が開発されています。開発された分化細胞は、基礎生物学だけでなく、再生医療のモデルや薬剤候補の評価に広く用いられています。多能性幹細胞が期待する細胞種に分化したかどうかを確認するためには、指標(マーカー)となる遺伝子・タンパク質の発現を検出する実験を行いますが、通常、これらの実験手法は細胞の破壊を伴います。このような細胞破壊を回避し、生きたまま細胞の状態を調べるためには、目的の遺伝子に対して、その発現状態を知らせるための蛍光や発光タンパク質(レポーター)を導入し、遺伝子発現を可視化することが一般的に行われています。
生きた細胞の遺伝子発現状態を可視化するために、ゲノム編集技術[2]を用いて、さまざまなマーカー遺伝子の末端に対して、蛍光タンパク質を共発現させる遺伝子をゲノムへと挿入(ノックイン)する方法があります。これにより、遺伝子発現の正確な監視(モニタリング)や特定の細胞タイプの選択(セレクション)が可能となります。このゲノム編集技術を用いたノックインは、一般的にCas9タンパク質・ガイドRNA(gRNA)発現ベクターとドナーベクターを共に導入して、対象とするゲノム配列に対して相同組換えを引き起こすことで行われます。
この相同組換えに基づくノックインの効率は、特に幹細胞では低くとどまっています。それは、細胞への遺伝子導入後にゲノムDNAの非特異的な位置にベクターが挿入されたり、ベクターが細胞質にとどまったりしてしまうためです。本研究では、ノックインされなかった細胞のみを薬剤で除去・選抜し、ノックインされた細胞を高効率に濃縮する手法を開発することを目指しました。
研究手法と成果
研究チームは、細胞のネガティブセレクション[3]に広く用いられている、単純ヘルペスウイルス由来のチミジンキナーゼ(HSV-tk)とそれに対応する抗ウイルス剤であるガンシクロビル(GCV)のシステムを用いました。この方法では、GCVによってHSV-tkを発現している細胞のみが除去されます。この方法は広く用いられていますが、単一のHSV-tk発現カセットを搭載したベクターに対するネガティブセレクションの効率は不十分であることが報告されていました。そこで、相同組換えを起こす領域の両外側にHSV-tk発現カセットを搭載するドナーベクター(「Double-tkドナーベクター」)を開発し、ドナーベクターがランダムに組み込まれた細胞がGCVによって確実に除去されるようにしました。この手法でヒトiPS細胞の多能性マーカーとして知られるOCT3/4(POU5F1)遺伝子[4]に対して、赤色蛍光タンパク質の一種であるtdTomatoをレポーターとして発現させるノックインシステムを構築しました(図1)。
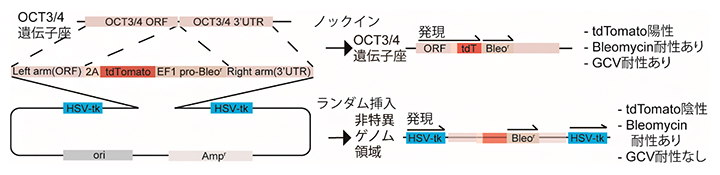
図1 OCT3/4(POU5F1)遺伝子に対するDouble-tkドナーベクターの設計とその概要
このDouble-tkドナーベクターは、OCT3/4(POU5F1)遺伝子の終止コドン前に発現タンパク質切断用の2Aペプチドと赤色蛍光タンパク質であるtdTomatoを発現させる。その後ろにEF1プロモーターで発現されるBleomycin耐性遺伝子(商品名としてZeocinなど)を搭載して、tdTomatoとBleomycin耐性遺伝子の組み合わせを作製する。相同組み換えを起こさせるためLeft armとRight armの配列で前記組み合わせの両側を挟み込む。さらにその両側にHSV-tk遺伝子を搭載したベクターを用いる。ori:プラスミド複製開始起点、Ampr:アンピシリン耐性遺伝子。
このDouble-tkドナーベクターとOCT3/4タンパク質のC末端に対するガイドRNA(gRNA)Cas9タンパク質の発現ベクターを一緒にiPS細胞に導入しました。その後、まずは抗生物質のBleomycinファミリーの一種であるZeocin(別名Phleomycin D1)で耐性細胞を選別し、その後に各種の濃度のGCVで処理したところ、tdTomato陽性細胞の割合が増加しました(図2)。このtdTomato陽性細胞を単離して解析したところ、tdTomatoの蛍光はiPS細胞が分化すると消失したため、未分化iPS細胞特異的なOCT3/4タンパク質の発現を可視化できていることが分かりました。さらに、細胞内で恒常的に発現しているEEF1A1(Elongation factor 1-alpha 1)[5]遺伝子とH2BC21(H2B clustered histone 21)[6]遺伝子に対しても同様に、Double-tkドナーベクターとGCVを用いることで、レポーターとなる蛍光タンパク質をノックインしたヒトiPS細胞を効率的に濃縮できることが分かりました。
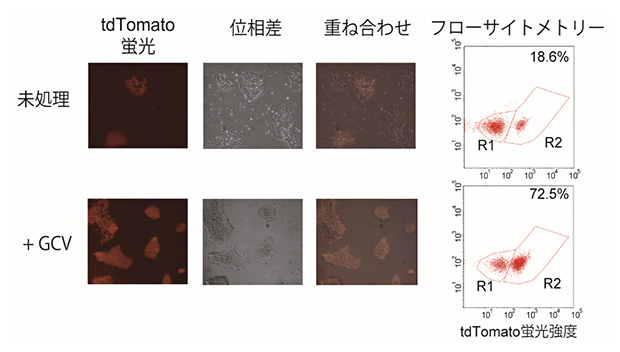
図2 OCT3/4(POU5F1)遺伝子に対するノックイン効率におけるGCVの影響
GCV(30 μg/mL)「あり」、「なし」それぞれのOCT3/4-tdTomatoレポーター発現を比較解析した結果。蛍光・位相差顕微鏡像(左図)とフローサイトメトリーデータ(右図)を示す。
以上は、ヒトiPS細胞で発現している遺伝子の例で、ゲノム編集でノックインすればすぐに蛍光タンパク質の発現が可視化できるものでした。一方、未分化iPS細胞では、発現していない遺伝子または特定の細胞種のみで発現が見られる分化マーカー遺伝子に対しては、すぐに発現を可視化できません。そのノックイン細胞の取得と確認には手間がかかり、未分化iPS細胞で発現している遺伝子に対してノックインするよりも、難易度が高くなります。
本研究では、運動神経細胞、膵臓前駆細胞、一部の心臓前駆細胞に発現しているISL1(Islet 1)遺伝子[7]に対して、蛍光タンパク質レポーターをノックインしました。GCVで処理していない細胞集団には約1%のノックインされた遺伝子が含まれていたのに対して、GCVで処理した細胞集団には約39%のノックインされた遺伝子が含まれていました(図3)。GCVで処理した細胞集団から一細胞に起因する23クローンを単離しました。これらのクローン株の遺伝子を調べると、23クローン中16クローンがノックインされた遺伝子を有していました。
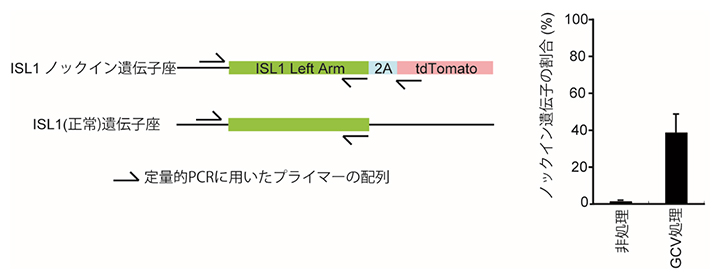
図3 ISL1遺伝子に対するノックイン効率の測定(左)とGCVの影響を調べた結果(右)
ISL1遺伝子の遺伝子座に対して、元々の遺伝子とノックインされた遺伝子配列のそれぞれに特異的に相補的なプライマーを設計した(左)。ISL1に対するtdTomatoレポーターiPS細胞作製のための遺伝子導入後、Zeocinで耐性細胞を選抜した後に、GCV処理群と非処理群に分けて、それぞれのゲノムDNAサンプルを抽出・精製した。これらのサンプルに対して、上記のプライマーを用いて定量的PCRで増幅し、存在量を測定する。それぞれのDNA産物の量比からノックイン遺伝子の割合を推定した結果のグラフを右側に示す。
以上の結果から、GCVでノックインされたiPS細胞を濃縮できること、単一細胞に由来するノックインiPS細胞クローン株の取得が容易になることが判明しました。これらのiPS細胞クローン株を運動神経細胞へと分化させると、tdTomatoの蛍光発現が観察できました。心筋細胞で特異的に発現するMYH7遺伝子[8]に対しても、同様にDouble-tkドナーベクターとGCVを用いることで、レポーターとなる蛍光タンパク質をノックインしたヒトiPS細胞を濃縮でき、効率的に単一細胞に由来するクローン株を取得できることが分かりました。
今後の期待
本研究では、相同組換えを起こす領域の両外側にHSV-tkの発現ユニットを配置した「Double-tkドナーベクターシステム」を開発し、このシステムを用いて、想定外のゲノム配列にランダムに挿入されたiPS細胞を除去し、正しくノックインされたヒトiPS細胞のみがGCV処理によって効率的に濃縮されることを示しました。蛍光タンパク質などのレポーターがノックインされた細胞を利用することで、細胞状態・細胞種を可視化することは、細胞・発生生物学的研究、創薬におけるタンパク質発現を指標とした薬剤候補評価系の構築、再生医療に向けた細胞製造法の確立に貢献します。
本研究などで開発した蛍光タンパク質レポーターiPS細胞株は順次、理研バイオリソース研究センター細胞材料開発室(理研細胞バンク)に寄託され、ウェブサイトにてその情報を公開しています注1)。
- 注1)理研 バイオリソース研究センター 細胞材料開発室(理研細胞バンク)「マーカー発現iPS細胞一覧」
補足説明
- 1.ヒトiPS細胞
ヒトを含む哺乳類の体細胞に、初期化する能力を持つ因子を導入し、培養すると、その細胞はさまざまな組織や臓器の細胞に分化する能力を持つ多能性幹細胞に変化する。この細胞をiPS細胞(誘導性多能性幹細胞)と呼ぶ。iPSはinduced pluripotent stemの略。 - 2.ゲノム編集技術
ゲノム内のDNA配列を意図的に切断し、切断されたDNAが損傷したり、修復されたりする過程で外来遺伝子などの配列に組換えが起こることを狙った技術。いくつかの手法があるが、短い塩基配列から成るガイドRNA(gRNA)と結合し、複合体を作って、それと相同なDNA配列に結合、切断するCas9タンパク質を用いて、特定のゲノムDNA配列を切断させる。ノックインの場合には、さらに切断部位と相同な(ホモロジードナー)配列を持ったDNAと共に導入することで、相同組換えを引き起こす。 - 3.細胞のネガティブセレクション
特定の反応性を持つ細胞のみを除去する過程のこと。特に「自殺遺伝子」とそれに対応する薬剤を用いることで、その遺伝子が発現している細胞のみを除去することができる。今回は、「自殺遺伝子」として、単純ヘルペスウイルスのチミジンキナーゼ(HSV-tk)とその基質であるガンシクロビル(GCV)を用いている。ガンシクロビルは、HSV-tkによってリン酸化されると、DNA複製阻害に起因する強い細胞毒性を示す。 - 4.OCT3/4(POU5F1)遺伝子
iPS細胞などの多能性幹細胞に特異的に発現している遺伝子の一つ。多能性の維持に重要な転写因子となるタンパク質をコードしている。OCT3/4は「リプログラミング因子」として、iPS細胞の誘導にも用いられている。 - 5.EEF1A1(Elongation factor 1-alpha 1)遺伝子
翻訳伸長タンパク質の一種として、タンパク質合成に重要である。基本的に全ての体細胞で発現して、その機能を果たしていると考えられるため、「ハウスキーピング」遺伝子として知られている。また、その発現を誘導するプロモーター領域の配列は非常に強い転写活性があるため、遺伝子工学で広く用いられている。 - 6.H2BC21(H2B clustered histone 21)遺伝子
細胞内でゲノムDNAにおけるクロマチン構造を形成する四つの主要なヒストンタンパク質のうちのH2Bタンパク質をコードする遺伝子の一つである。H2Bタンパク質に蛍光タンパク質を融合することで、通常の蛍光顕微鏡での観察において染色体の様子を可視化することができ、細胞周期などの指標となる。 - 7.ISL1(Islet 1)遺伝子
LIMホメオドメインを持つ転写因子で、膵臓のβ細胞において、インスリン遺伝子の発現調節領域に結合して、その発現を制御する因子として発見された。現在では、膵臓の他、心臓や神経系の発生において重要な役割を担っていることが知られている。 - 8.MYH7遺伝子
ミオシン重鎖β(MHC-β)アイソフォームタンパク質をコードする遺伝子で、主に心臓に発現する。MHC-βは心筋の太いフィラメントを構成する主要なタンパク質であり、心筋収縮に大きな役割を果たしている。
研究チーム
理化学研究所 バイオリソース研究センター
iPS細胞高次特性解析開発チーム
チームリーダー 林 洋平(ハヤシ・ヨウヘイ)
(筑波大学 医学医療系 教授[連携大学院]、筑波大学グローバル教育院 ライフイノベーション学位プログラム 教授[協働大学院])
開発研究員 髙﨑 真美(タカサキ・マミ)
テクニカルスタッフⅡ(研究当時)塚本 聡美(ツカモト・サトミ)
テクニカルスタッフⅡ(研究当時)安 瑜利(アン・ユリ)
テクニカルスタッフⅡ 辺見 康子(ヘンミ・ヤスコ)
テクニカルスタッフⅡ 佐藤 伊織(サトウ・イオリ)
研修生 下田 柚須乃(シモダ・ユズノ)
(筑波大学大学院 博士課程、日本学術振興会特別研究員DC1)
研修生(研究当時)Jingyue Li(リ・ケイゲツ)
(筑波大学大学院 修士課程)
遺伝子材料開発室
室長 三輪 佳宏(ミワ・ヨシヒロ)
開発研究員(研究当時)中出 浩司(ナカデ・コウジ)
テクニカルスタッフⅡ 中島 謙一(ナカシマ・ケンイチ)
研究支援
本研究は、日本医療研究開発機構(AMED)再生・細胞医療・遺伝子治療プロジェクト「疾患特異的iPS細胞の樹立・特性解析・加工の高度化・効率化・情報公開(研究開発代表者:林洋平(23bm1423010h0001)」、ナショナルバイオリソースプロジェクト「遺伝子材料の収集・保存・整備・提供(課題管理者:三輪佳宏(21ae0201004s0504)」、科学技術振興機構(JST)つくば型デジタルバイオエコノミー社会形成の国際拠点「生活習慣病を含む多因子疾患を原因とする腎機能低下の病態解明および進行予測マーカーの同定に向けた次世代医療基盤の構築(研究開発課題リーダー:西山博之、(JPMJPF2017)」、日本学術振興会(JSPS)科学研究費助成事業若手研究(A)「染色体異常を伴った疾患特異的iPS細胞を修復する「染色体編集法」の開発(研究代表者:林洋平、課題番号:17H05063)」、同基盤研究(C)「立体構築・組織維持・線維症におけるコラーゲン解析基盤モデル動物の構築(研究代表者:三輪佳宏、課題番号:21K05994)」による助成を受けて行われました。
原論文情報
- Koji Nakade, Satomi Tsukamoto, Kenichi Nakashima, Yuri An, Iori Sato, Jingyue Li, Yuzuno Shimoda, Yasuko Hemmi, Yoshihiro Miwa and Yohei Hayashi, "Efficient selection of knocked-in pluripotent stem cells using a dual cassette cellular elimination system", Cell Reports Methods, 10.1016/j.crmeth.2023.100662
発表者
理化学研究所
バイオリソース研究センター iPS細胞高次特性解析開発チーム
チームリーダー 林 洋平(ハヤシ・ヨウヘイ)
 林 洋平
林 洋平
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
