2013年5月14日
理化学研究所
モデル実験植物と作物遺伝子をつなぐデータベース「SABRE2」を公開
-シロイヌナズナの豊富な遺伝子情報を活用して作物研究を加速する基盤を整備-
ポイント
- 既存のデータベースにコムギなど6種の作物遺伝子の情報を追加、より実用的に
- 蓄積されたシロイヌナズナの研究成果が有用な作物作出に貢献
- 『From bench to bountiful harvest』(実験台から豊かな収穫へ)を目指す
概要
理化学研究所(理研、野依良治理事長)は、植物遺伝子情報データベース「SABRE(セイバー)[1]」にナショナルバイオリソースプロジェクト(NBRP)[2]から提供されている植物6種の遺伝子クローン情報を加えて、他種の植物間で横断的に検索できる「SABRE2(セイバー2)」を開発しました。理研バイオリソースセンター(理研BRC、小幡 裕一センター長)のホームページに5月15日から公開します。
これは、理研BRC情報解析技術室の深海 薫(ふかみ かおる)室長と同センター実験植物開発室の小林 正智室長らによる成果です。
SABRE2の前身となるSABRE(セイバー)は、国際的なモデル実験植物として広く利用されているシロイヌナズナ[3]の遺伝子を基にして、他の植物の遺伝子クローンを横断的に検索できるデータベースです。SABREの大きな特徴は、シロイヌナズナの研究で豊富に得られたそれぞれの遺伝子の機能に関する情報を、他の植物が持つ類似遺伝子と関連付けて活用できることです。ただ、さらなる応用研究を展開するためにはSABREへ新たな植物遺伝子の情報を追加し、機能を拡充する必要がありました。
そこで今回、NBRPから提供されているモデル植物6種(コムギ、オオムギ、アサガオ、ミヤコグサ、ダイズ、トマト)の遺伝子クローンの情報を追加し、合計14種の植物遺伝子クローン情報を一度に検索できるSABRE2を開発しました。
SABRE2は、SABREに比べて約3倍となる150万を超える植物遺伝子クローンの情報を持ちます。質・量ともに大きく変化したデータの検索を容易に行えるよう、検索画面や結果の表示の仕方に大幅な変更を加えました。例えば、検索キーワードの入力画面は、クローン名や遺伝子番号など、どのような言葉をキーワードに使えばよいかが一目で分かるようなデザインにしました。また、検索・表示対象とする植物種を利用者が取捨選択して設定できるようにし、必要な検索結果だけ得られるようにしました。SABRE2には、インターネットが利用できる環境であれば、PCやスマートフォンを使って誰もが簡単にアクセスできます。さらに、API(エーピーアイ)[4]を用いることでSABRE2の機能を他のウェブサイトやデータベースからも利用できるようにしました。
このデータベースが多く活用されることにより、幅広い研究分野でシロイヌナズナに関する情報を活用した研究が進み、有用作物の創出など食料や環境に貢献する成果につながることが期待できます。
なお、本研究は科学技術振興機構 ライフサイエンスデータベース統合推進事業「ゲノム情報に基づく植物データベースの統合」(代表者 田畑哲之かずさDNA研究所副所長)の支援によって行われました。
背景
シロイヌナズナは、草丈40センチほどの小型な高等植物で、蛍光灯の光だけで栽培可能なため実験室内でも育ち、3カ月程度で次世代の種子を採取できます。このように研究用として扱いやすい特徴を持つことから、世界標準のモデル実験植物として植物のさまざまな現象や機能を理解する基礎研究で盛んに利用されています。2000年12月には高等植物として初めて全ゲノムの塩基配列が解読されました。
シロイヌナズナの全ゲノム解読に続き、2001年から約2万7000個のシロイヌナズナの全遺伝子の機能を調べることを目標とした国際連携プロジェクト、“Arabidopsis 2010 Project”が開始されました。現在、多くの遺伝子の機能に関する情報が得られています。
プロジェクト最終年の2010年6月に横浜で開催された国際シロイヌナズナ研究推進委員会は、今後のシロイヌナズナ研究の方向性を議論するなかで、新たな目標として『From bench to bountiful harvest』(実験台から豊かな収穫へ)が掲げられました。それを受けて各大学や研究機関では、蓄積されたシロイヌナズナの研究成果を生かした有用な作物に関する研究開発に取り組んでいます。
日米欧のリソースセンターでは、完全長cDNA[5]をはじめとする膨大なシロイヌナズナの遺伝子クローンを保存・提供し、得られた研究成果を米国の情報センターに集めるという国際的な仕組みが構築され、研究推進に貢献してきました。理研BRCはシロイヌナズナの世界的な拠点として、完全長cDNAや変異体種子、培養細胞などの多様な実験材料を国内外に提供しています。このシロイヌナズナの研究基盤が多くの研究者により活用されれば、作物研究を加速することができます。
そこで2007年には理研BRCが保有する植物4種(シロイヌナズナ、ヒメツリガネゴケ、タバコの培養細胞、ポプラ)の遺伝子を横断的に検索できるデータベース「SABRE(セイバー)」を開発し、その後もキャッサバ、塩生植物ハロフィラ、寄生植物ストライガ、ハクサイと植物種を追加し、さまざまな植物種の遺伝子情報をシロイヌナズナのゲノム情報や完全長cDNAに連結してその活用を図ってきました。
一方、わが国では2002年から開始されたナショナルバイオリソースプロジェクト(NBRP)により多くの植物種の遺伝子情報が収集され、それぞれの植物種の中核機関より提供されています。しかし、これらの植物種の遺伝子情報全体をシロイヌナズナのゲノム情報に連結する機能を持ったデータベースはこれまで存在しませんでした。
そこで理研BRCは、シロイヌナズナに関する情報を作物研究に活用し、国内外の植物研究を活性化することを目的として、SABREの改良に着手しました。
研究手法と成果
SABREの大きな特徴は、TAIR(テア)[6]と呼ばれるシロイヌナズナのデータベースを「串」にして、類似の配列を持つ他の植物の遺伝子クローンを検索できることです。遺伝子クローンとTAIRの遺伝子モデル、その遺伝子モデルに付加された生物学的情報が串刺しになって検索されることで、シロイヌナズナの遺伝子機能解析の成果を、他の植物が持つ類似遺伝子に関連付けて活用できるようになっています(図1)。つまり、ある植物種では初めて見つかった遺伝子でも、シロイヌナズナに類似遺伝子があれば、機能を推測することができます。
今回、NBRP情報中核機関(国立遺伝学研究所)から提供を受けた情報をもとに、モデル植物6種(コムギ、オオムギ、アサガオ、ミヤコグサ、ダイズ、トマト)の遺伝子クローンの情報を追加し、合計14種の遺伝子クローンを一度に検索できるSABRE2を開発しました(図2)。その結果、従来のSABREに比べて約3倍となる150万を超えるクローンが検索対象となり、NBRPから入手可能なすべての植物遺伝子クローンが検索可能となりました。検索された遺伝子クローンからは各植物種の中核機関のデータベースへアクセスできるようにリンクが張られており、より詳細な情報が入手できて提供の申込みも行えます。
SABRE2では、データの質や量がともに大きく変化したため、それに対応して検索画面や表示方法にさまざまな工夫を施しました。例えば、検索キーワードの入力画面は、クローン名や遺伝子番号など、どのような言葉をキーワードに使えばよいかが一目で分かるようなデザインにし、キーワード入力時には候補を表示するようにしました。取り扱う植物種が増えたため、検索や表示対象とする植物種を利用者が取捨選択して設定できるようにし、必要な結果だけ得られるようにしました。このような工夫により、作物の育種研究者などのシロイヌナズナ以外の植物研究者にも使いやすくしました。また、PCからのアクセスに加え、スマートフォンからのアクセスに対しての画面デザインも用意しました。さらに、API(エーピーアイ)を用いることでSABRE2の機能を他のウェブサイトやデータベースからも利用できるようにしました。
今後の期待
SABRE2は実験植物と作物、基礎研究と応用研究を連結するための、国際的にもユニークなツールです。搭載する遺伝子情報が全て入手可能なcDNAクローンに由来することも大きな特徴です。このデータベースが多くの研究者に活用されることにより、幅広い研究分野でシロイヌナズナのリソースや情報を活用した研究が進み、食料や環境に貢献する成果につながることが期待できます。
発表者
独立行政法人理化学研究所
バイオリソースセンター 情報解析技術室
室長 深海 薫(ふかみ かおる)
バイオリソースセンター 実験植物開発室
室長 小林 正智(こばやし まさとも)
お問い合わせ先
バイオリソース推進室
Tel: 029-836-9058 / Fax: 029-836-9100
報道担当
独立行政法人理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.
- SABRE(セイバー)
-
正式名称はSystematic consolidation of Arabidopsis and other Botanical REsource。
SABREでは、TAIRと遺伝子クローンのデータベースを、両者が持つ遺伝子配列の類似度をもとに関連づけている。配列の類似度は、各遺伝子クローンが持つ配列情報を用いて、TAIRの遺伝子モデルの塩基配列にblastn、遺伝子モデルの翻訳アミノ酸配列にblastx検索を行うことで調べられる。blastn、blastxはどちらも、バイオインフォマティクスで配列の相同性検索を行う際に、世界的に最もよく用いられているコンピュータプログラムである。SABREにはblastn、blastxによる検索の結果が格納されている。そして指定された条件に従い、検索結果から条件にかなう遺伝子クローンとそれに関連付けられているTAIRの遺伝子モデル、その遺伝子モデルに付加された生物学的情報を串刺しにして抽出し、表示する。
- 2.
- ナショナルバイオリソースプロジェクト(NBRP)
-
NBRPは、文部科学省によるライフサイエンス研究の基盤を整備するプロジェクト。2002年度からバイオリソース(実験動植物やES細胞などのライフサイエンス研究開発のための実験材料)について収集・保存・提供を実施する拠点の整備を行っている。理研は、中核的拠点整備プログラムの代表的な機関として、シロイヌナズナのほか、実験動物マウス、ヒト・動物細胞、遺伝子材料、一般微生物などのバイオリソースの整備を担っている。このほか20を超える生物種について、大学などの中核機関が選定され、整備を行っている。
- 3.
- シロイヌナズナ
-
アブラナ科に属する一年草で、モデル実験植物として広く用いられている。2000年12月に高等植物として初めて全ゲノムの塩基配列が解読された。理研BRCは遺伝子を破壊した系統と完全長cDNAを中心に、30万を超える数のシロイヌナズナの実験材料を提供している。また理研BRCも参加する国際研究推進委員会では2020年までのロードマップに『From bench to bountiful harvest』(実験台から豊かな収穫へ)を設定しており、研究成果の社会への還元が研究コミュニティの目標の1つとなっている。
- 4.
- API(エーピーアイ)
-
正式名称はApplication Program Interface。個々のソフトウェアの開発者が全ての機能をプログラミングするのは困難で無駄が多いため、多くのソフトウェアが共通して利用する機能を、命令や関数の集合の形にまとめて使用できるようにしたもの。または、それらを利用するためのプログラム上の手続きを定めた規約の集合。個々の開発者は規約に従ってその機能を「呼び出す」だけで、自分でプログラミングすることなくその機能を利用したソフトウェアを作成することができる。
今回のSABRE2の開発では、機能拡張の実装にはAPIを利用した。それによりSABRE2の機能が他サイト、データベースからも利用可能となっており、データベース統合の基盤を提供することにも貢献している。
- 5.
- 完全長cDNA
-
cDNAは、mRNA(メッセンジャーRNA)から人工的に合成したDNAのこと。mRNAは細胞の中でゲノムのDNAを鋳型として作られ、タンパク質の設計図として使われる。中でも完全長cDNAはmRNAの全長配列をもとに作成したDNAを指し、完全なタンパク質の合成に必要となる情報全てを持っているため利用価値が高い。理研BRCはシロイヌナズナをはじめさまざまな生物種の完全長cDNAクローンを提供している。
- 6.
- TAIR(テア)
-
正式名称はThe Arabidopsis Information Resource。シロイヌナズナのゲノム、遺伝子、分子生物学的データを収集したデータベース。注釈付けされたゲノム、遺伝子産物、代謝、遺伝子発現、マーカー、リソース情報、文献情報を提供している。豊富な情報量と情報の正確さ、情報整備の質の高さが特色で、世界中のシロイヌナズナ研究の情報基盤として利用されている。SABRE2では、TAIRに記載されているシロイヌナズナの遺伝子モデルならびにそれらに付加された生物学的情報を、植物遺伝子リソースの横断検索に利用している。
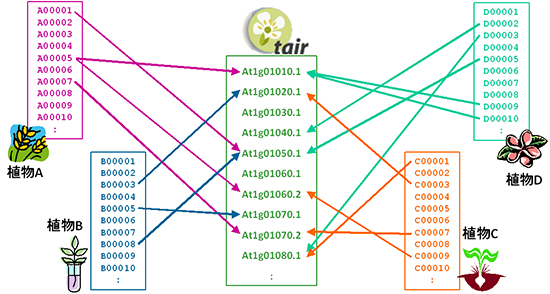
図1 相同性検索で遺伝子クローンとTAIR遺伝子モデルを関連付ける
これによりTAIRを介して、異なる植物の類似遺伝子を横断的に検索することが可能になる。
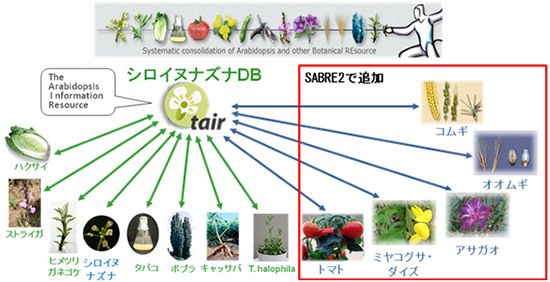
図2 SABRE2が検索対象としている植物種
四角で囲った部分が、SABRE2で新たに追加された。
