2014年6月6日
理化学研究所
神経細胞で働くmRNAを網羅的に同定する新しい手法を確立
-小脳の「プルキンエ細胞」の部位特異的な転写物全体の解析を実現-
ポイント
- プルキンエ細胞で働く数千種類のmRNAを網羅的に同定
- プルキンエ細胞の各部位特異的に発現するmRNAを分類
- 運動の学習などを担うプルキンエ細胞の働きの理解に大きく貢献
要旨
理化学研究所(理研、野依良治理事長)は、ラット小脳[1]のプルキンエ細胞[2]で翻訳中のmRNA[3]を、網羅的かつ細胞内部位特異的に同定する手法を確立しました。これは、理研脳科学総合研究センター(利根川進センター長)Launey研究ユニットのパスカル・ベガン研究員、トーマス・ローニー ユニットリーダーと、理研ライフサイエンス技術基盤研究センター(渡辺恭良センター長)ゲノミクス微量技術開発ユニットのアントン・クラッツ訪問研究員、シャルル・プレシ ユニットリーダーらの研究グループによる成果です。
小脳は運動学習に不可欠な脳の部位です。小脳ネットワーク内で唯一の出力神経細胞として中心的な役割を果たすのがプルキンエ細胞ですが、その機能を支える分子機構の詳細は分かっていません。そのため、プルキンエ細胞で作られるタンパク質を完全に網羅したカタログの作成が待たれてきました。また、運動学習を支える重要なプロセスの多くが樹状突起[4]で作られるタンパク質を必要としていることから、このカタログは細胞内部位特異的な精度であることが望まれます。
特定の種類の細胞で作られているタンパク質をリストアップするには、その細胞内でリボソーム[5]に結合し、まさにタンパク質へと翻訳されているmRNAを選択的に取り出す「TRAP法[6]」が有効ですが、費用と時間のかかる遺伝子改変マウスの作製が必要です。今回研究グループは、特定の神経細胞に感染するウイルスを利用し、より迅速・安価で、遺伝子改変マウス以外の動物にも広く適用の可能性のある手法を確立しました。そしてこの「改良版TRAP法」でラットのプルキンエ細胞の細胞内部位ごとにmRNAを回収し、理研が独自に開発した「CAGEscan法[7]」で、数千ものmRNA配列を高感度で定量的に読み出しました。配列データの解析の結果、世界で初めて、ラットプルキンエ細胞で作られているタンパク質を細胞内部位特異的にほぼ網羅したカタログの作成に成功しました。
今回研究グループが開発した手法は、より迅速かつ安価なだけでなく、疾患の動物モデル研究に不可欠な霊長類への応用も期待できます。小脳のプルキンエ細胞の異常はスムーズな動きができなくなる運動失調症を引き起こし、またある種の自閉症患者の脳ではプルキンエ細胞の減少や異常が確認されています。本研究の成果がこうした疾患の研究に貢献し、将来的には治療法の開発につながっていく可能性もあります。
本研究成果は、米国の科学雑誌『Genome Research』に近日掲載されます。
背景
私たちの脳には1,000億以上もの神経細胞があると推定されています。これらの神経細胞は、互いにつながり合ってコミュニケーションをとることで、反射のような反応や、記憶や学習などの高次機能を可能にしています。神経細胞において、他の細胞から入力を受け取るのに特化した部位を樹状突起といいます(図1)。核を含む部位は細胞体で、そこから伸びる軸索という構造を通じて、信号は次の神経細胞に伝えられます。この樹状突起、細胞体、軸索の形は神経細胞の種類によってさまざまです。
この膨大な数の脳神経細胞のうち半分は、体積では脳全体の10パーセントしかない小脳にあるといわれています。私たちは一度覚えた自転車の乗り方を忘れませんし、スポーツで学んだ技などは「体が覚えている」という表現をしますが、こうした運動学習にはこの小脳が関与しています。小脳のネットワーク内で、唯一の出力神経細胞として中心的な役割を果たしているとされるのが、小脳に特異的に存在する「プルキンエ細胞」です。プルキンエ細胞では樹状突起が非常によく発達し、その入力量は平均的な神経細胞の約10倍といわれています。
しかし、このようなプルキンエ細胞の機能や形態がどのような分子機構によって支えられているのか、詳細は分かっていません。そのため、小脳研究の領域では、プルキンエ細胞で作られるタンパク質を完全に網羅したカタログの作成が待たれてきました。また、運動学習は、プルキンエ細胞とそこに入力する神経細胞とのつながりの強弱が変化することで起こるとされていますが、この変化に関与するタンパク質の多くは樹状突起で作られていることが分かってきています。このため、樹状突起で作られているタンパク質を全て同定できれば、運動学習を可能にする分子機構の理解が大きく進むと考えられます。そこで研究グループは、プルキンエ細胞とその樹状突起で作られているタンパク質をそれぞれ網羅的に同定しようと試みました。
研究手法と成果
タンパク質は、遺伝子の情報がmRNAを介して読み取られ、それがリボソームという細胞小器官で翻訳されることにより作られます。特定の種類の細胞で作られているタンパク質を全てリストアップするためには、その細胞内でリボソームに結合しているmRNAを選択的に取り出す技術が有効です。このような技術の1つがTRAP法です。しかし、従来のTRAP法では、特定の種類の細胞に由来するリボソームを標識するプローブタンパク質を発現させた遺伝子改変動物が必要で、時間と費用がかかり、マウス以外の動物に適用することは困難でした。そこで今回、研究グループは、より迅速かつ安価で、遺伝子改変マウス以外の動物にも広く適用の可能性のある手法を確立しました(図2)。
この「改良版TRAP法」は、翻訳中のmRNAをラットのプルキンエ細胞から選択的に抽出するために、リボソームタンパク質(RPL10A)と蛍光タンパク質(YFP)を融合させたプローブタンパク質(YFP-RPL10A)を、プルキンエ細胞特異的に発現させる手法です。まず、プローブタンパク質をコードする遺伝子を、ラットのプルキンエ細胞を標的とするよう設計したアデノ随伴ウイルス[8]に挿入し、生後間もないラットの小脳に注入します。数週間後、極小メスを用いて、プルキンエ細胞の樹状突起部分と細胞体部分を分離し、それぞれの試料からプローブタンパク質で標識されたリボソームを抽出します。これらのリボソームには、プルキンエ細胞内のそれぞれの部位でタンパク質に翻訳されていたmRNAが結合しています。研究グループは、理研が独自に開発した「CAGEscan法」で、数千ものこのようなmRNAの配列を高感度かつ定量的に読み出しました(図2)。配列データの解析の結果、世界で初めて、ラットのプルキンエ細胞で作られているタンパク質をほぼ網羅的に記述するカタログの作成に成功しました。
今後の期待
プルキンエ細胞内で作られるタンパク質のカタログは、小脳が関与するさまざまな現象を支える分子機構の解明に広く貢献するものと考えられます。特に、細胞内部位特異的なカタログを作成したことで、運動学習のようにタンパク質の作られる場所が重要な現象の研究への寄与が期待できます。
また、今回開発した「改良版TRAP法」は、遺伝子改変動物の作成を必要としない画期的な手法で、疾患の動物モデル研究に不可欠な霊長類への応用も期待されます。
プルキンエ細胞の異常は、スムーズな動きができなくなる運動失調症を引き起こし、ある種の自閉症の患者の脳ではプルキンエ細胞の減少や異常が見られます。今回の研究成果には、これらの疾患の原因の究明に貢献し、さらにはその治療法の発見につながっていく可能性も期待されます。
原論文情報
- Anton Kratz, Pascal Beguin, Megumi Kaneko, Takahiko Chimura, Ana Maria Suzuki, Atsuko Matsunaga, Sachi Kato, Nicolas Bertin, Timo Lassmann, Rejan Vigot, Piero Carninci, Charles Plessy and Thomas Launey. "Digital expression profiling of the compartmentalized translatome of Purkinje neurons". Genome Research, 2014, doi:10.1101/gr.164095.113
発表者
理化学研究所
脳科学総合研究センター Launey研究ユニット
ユニットリーダー トーマス・ローニー (Thomas Launey)
ライフサイエンス技術基盤研究センター 機能性ゲノム解析部門 LSA要素技術研究グループ ゲノミクス微量技術開発ユニット
ユニットリーダー シャルル・プレシ (Charles Plessy)
お問い合わせ先
脳科学研究推進室
Tel: 048-467-9757 / Fax: 048-462-4914
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
補足説明
- 1.小脳
脳幹の背側に位置し、カリフラワーのような外観をしている脳の部位。脳全体の体積・重さの約10%程度を占める。運動に関与している。 - 2.プルキンエ細胞
小脳皮質にある、GABA性抑制性の投射神経細胞。平たく非常に発達した樹状突起を持つ。 - 3.mRNA
生物の体内では多くのタンパク質が働いているが、タンパク質は核内のDNA配列の遺伝子にあたる部分がRNAという形でコピーのように写し取られ(転写)、それが読み出される(翻訳)ことで作られる。この転写されたRNAをmRNA(メッセンジャーRNA)と呼ぶ。タンパク質の翻訳はリボソームで行われる。 - 4.樹状突起
神経細胞が他の神経細胞から信号(入力)を受け取るのに特化した構造で、樹木の枝のように細かく分岐している。神経細胞同士のつながりであるシナプスが形成される。 - 5.リボソーム
細胞の小器官の1つ。mRNAから情報を読み取り、タンパク質へ翻訳する。転写されたmRNAはリボソームに結合する。そこにtRNA(トランスファーRNA)が遺伝子情報に対応したアミノ酸を運び、タンパク質が作られる。 - 6.TRAP法
TRAP:Translating Ribosomal Affinity Purificationの略。リボソームを構成するタンパク質を細胞種特異的に標識し、そのタンパク質に結合している物質を結合したまま取り出す手法。
参考文献:Doyle JP, Dougherty JD, Heiman M, Schmidt EF, Stevens TR, Ma G, Bupp S, Shrestha P, Shah RD, Doughty ML, et al. 2008. Application of a translational profiling approach for the comparative analysis of CNS cell types. Cell 135: 749–762 - 7.CAGEscan法
CAGE:Cap Analysis of Gene Expressionの略。キャップ構造を持つmRNAのcDNAの5’端を定量的にシーケンス(遺伝子配列を読む)する手法。理研ライフサイエンス技術基盤研究センター ゲノミックス微量技術開発ユニットの シャルル ・プレシ ユニットリーダーが開発した。
参考文献:Plessy C, Bertin N, Takahashi H, Simone R, Salimullah M, Lassmann T, Vitezic M, Severin J, Olivarius S, Lazarevic D, et al. 2010. Linking promoters to functional transcripts in small samples with nanoCAGE and CAGEscan. Nat Methods 7: 528–534. - 8.アデノ随伴ウイルス
分裂期・非分裂期いずれの細胞にも感染し、遺伝子を細胞内に取り込ませることができるウイルス。病原性は今のところ確認されておらず、細胞内に外来遺伝子を導入する道具として広く利用される。
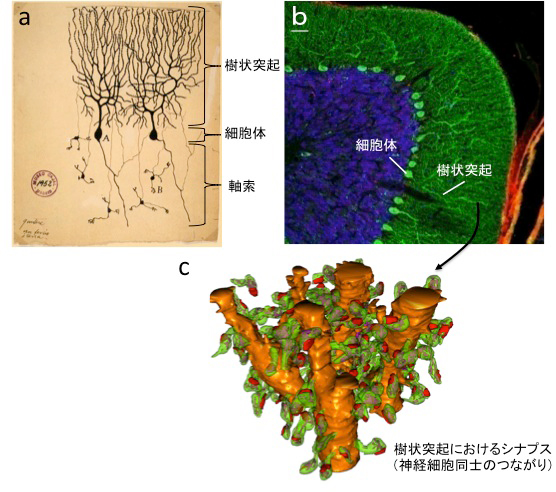
図1 プルキンエ細胞の形態
- a.スペインの神経細胞学者サンティアゴ・ラモン・イ・カハルによるプルキンエ細胞(スケッチの中のAで示されている細胞)のスケッチ(Santiago Ramón y Cajal 1899. Instituto Ramón y Cajal, Madrid, Spainから引用)。樹木の枝のように分岐している部分が樹状突起、核のある部分が細胞体、そこから伸びているのが軸索である。スケッチの中のBで示されている神経細胞は顆粒細胞と呼ばれるが、その形態は、プルキンエ細胞とは極めて異なっていることが分かる。
- b.小脳の皮質部分。緑;プルキンエ細胞、青;顆粒細胞
- c.プルキンエ細胞の樹状突起にあるシナプスの構造。電子顕微鏡から再構成したモデル図。赤・緑の部分が、シナプスが存在するスパインという構造。
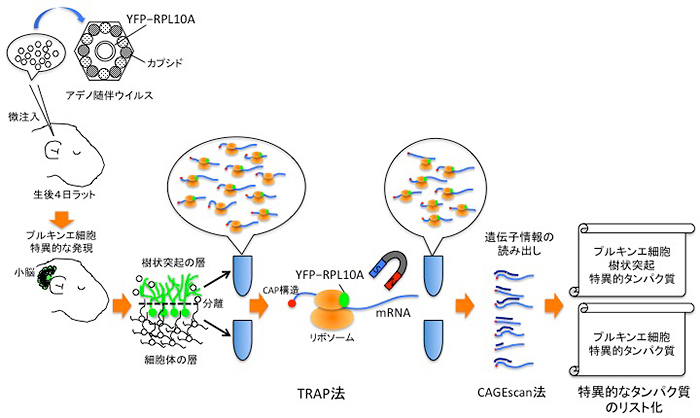
図2 今回用いた研究手法の概略
翻訳中のmRNAをラットのプルキンエ細胞から選択的に抽出するため、リボソームタンパク質(RPL10A)と蛍光タンパク質(YFP)を融合させたプローブタンパク質(YFP-RPL10A)をプルキンエ細胞特異的に発現させる。プローブタンパク質をコードする遺伝子を、ラットのプルキンエ細胞を標的とするよう設計したアデノ随伴ウイルスに挿入し、生後4日のラットの小脳に注入する。数週間後、YFPの蛍光を目印に、プローブタンパク質がプルキンエ細胞だけで強く発現していることを確認する。極小メスを用いて、プルキンエ細胞の樹状突起部分と細胞体部分を分離する。それぞれの試料からプローブタンパク質で標識されたリボソームを磁力により選択的に抽出する。これらのリボソームには、プルキンエ細胞のそれぞれの部位でタンパク質へと翻訳されていたmRNAが結合している。mRNAのCAP構造を認識しながら高感度で定量的にmRNA配列を読み出す「CAGEscan法」を用いて、それぞれの細胞内部位で作られているタンパク質をカタログ化した。
