理化学研究所(理研)生命医科学研究センター 骨関節疾患研究チームの池川 志郎 チームリーダー(研究当時)、ゲノム解析応用研究チームの寺尾 知可史 チームリーダー、小井土 大 客員研究員らの研究チームは、後縦靭帯骨化症(OPLL)の発症に関わる遺伝子「CCDC91」を発見し、分子レベルでその作用を明らかにしました。
本研究成果は、現在のところ有効な治療法のない難病であるOPLLの原因解明、治療法の開発、ならびに予防医学研究に貢献すると期待できます。
OPLLは、背骨の後ろを縦に走る後縦靭帯が骨に変化し、脊髄や神経を圧迫して、手や足のしびれや痛み、運動障害などを引き起こす疾患です。OPLLの発症には遺伝的要因が関与すると考えられ、日本を中心に原因遺伝子の探索が行われてきました。池川チームリーダーらは2014年に、ゲノムワイド関連解析(GWAS)[1]を用いて、OPLLの発症しやすさ(疾患感受性)に関わる複数のゲノム領域を世界に先駆けて発見しています。
今回、研究チームは、GWASで決定していた領域の一つからOPLL発症に関わる遺伝子CCDC91を同定しました。そして、CCDC91遺伝子の組織特異的な転写産物が、靭帯で非翻訳RNA[2]として働き、MIR890というマイクロRNA(miRNA)[3]と結合することで、骨形成に関係する遺伝子を制御していることを突き止めました。
本研究は、科学雑誌『American Journal of Human Genetics』オンライン版(3月28日付)に掲載されました。
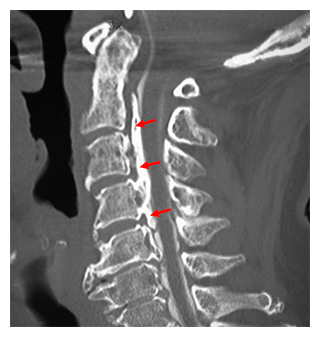
頚椎(背骨の首の部分)を側面から見た後縦靭帯骨化症のCT像(赤矢印は骨化した後縦靭帯)
背景
骨、関節をはじめとする運動器の疾患には、多くの遺伝性難病が含まれています。池川チームリーダーらは、運動器の遺伝性難病の原因遺伝子の同定を出発点として、原因遺伝子とその変異の機能解析を通じて画期的な診断法・治療法を開発するとともに、骨格や運動器の成長・発達・維持のメカニズムの解明を目指し、研究を続けてきました。これまでに、数多くの運動器疾患の原因遺伝子を発見し、その分子病態を明らかにしてきました注1)。
「後縦靭帯骨化症(OPLL: ossification of the posterior longitudinal ligament of the spine)」は、背骨の椎体の後面を縦に走る後縦靭帯が骨に変化し、そのすぐ後ろにある脊髄や神経を圧迫して、手や足のしびれや痛み、運動障害などさまざまな症状を引き起こす疾患です(図1)。50歳前後で発症することが多い、極めて頻度の高い疾患で、日本での患者数は100万人以上と考えられています注2)。しかし、現在のところ根本的な治療法はなく、厚生労働省が定める指定難病となっています。
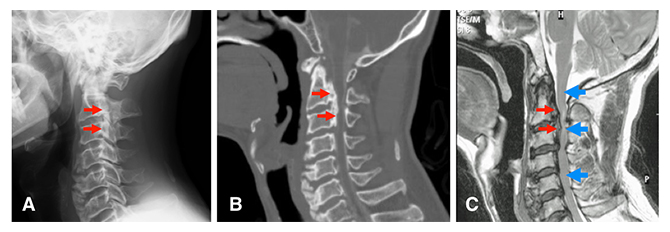
図1 後縦靭帯骨化症(OPLL)の画像所見
同一患者の頚部(首)の側面像。左が腹側、右が背側。
- (A)単純X線像。赤矢印で示した部分が骨化した靭帯。
- (B)同じ部位のCT像。第2頚椎椎体から第3頚椎椎体の背側にかけて、骨化した後縦靭帯(赤矢印)が見られる。
- (C)同じ部位のMRI像。青矢印で示した部分が椎体の背側にある脊髄。骨化した靭帯により圧迫されて、特に上から2番目の青矢印周辺で細くなっている。
過去の疫学研究から、OPLLの発症・進行には遺伝的要因が関与すると考えられ、日本を中心に原因遺伝子の探索が行われてきました。2014年、池川チームリーダーらは日本の患者を対象としたOPLLのゲノムワイド関連解析(GWAS)を行い、発症しやすさ(疾患感受性)に関わるゲノム領域を複数発見しました注3)。本研究では、OPLLの疾患感受性遺伝子[4]とその多型(個人差)の同定に挑みました。
- 注1)池川 志郎.整形外科領域のゲノム解析. 整形外科71(6):716-9, 2020.
- 注2)Sasaki E, et. al. Prevalence and symptom of ossification of posterior longitudinal ligaments in the Japanese general population. J Orthop Sci. 19:405-411,2014. doi: 10.1007/s00776-014-0552-0.
- 注3)2014年7月28日プレスリリース「後縦靭帯骨化症(OPLL)の発症に関わる6つのゲノム領域を発見」
研究手法と成果
小井土 客員研究員らは2022年、DNA配列の変化による非翻訳RNA発現量の変化を機械学習によって予測するMENTR(Mutation Effect prediction on NcRNA TRanscription)法を開発しました注4)。研究チームはまず、このMENTR法を2014年のOPLLのGWAS結果に適用しました。すると、軟骨細胞、骨芽細胞、間葉系幹細胞[5]などのOPLL関連細胞において、6個のCAGEタグ[6](mRNAの5'末端の約20塩基の配列)の発現が計12個のOPLLの疾患感受性(リスク)遺伝子多型と相関することが分かりました。それらのリスク遺伝子多型のうちrs35098487は「CCDC91(coiled coil domain 91)遺伝子」領域内に存在し、他のCAGEタグ「p4@CCDC91」の発現に影響していました。
CCDC91遺伝子には、22個の転写産物アイソフォーム[7]が存在することが知られています。しかし、p4@CCDC91の配列はそれら既知のアイソフォームのどれとも一致せず、「新しいアイソフォーム」の転写開始点の塩基配列であることが分かりました。そこで、リアルタイムPCR法[8]と3'-RACE[9]というin vitro(試験管内で)の実験手法を用いて、この新しいアイソフォームの全長を同定しました。このアイソフォームは、大多数の既知のアイソフォームと同じ翻訳開始点を持っていますが、翻訳の読み枠には終止コドン[10]がなく、翻訳領域の途中にポリA(アデノシンが複数連続した配列)が付加された「ノンストップmRNA[11]」でした(図2)。一般に、ノンストップmRNAはノンストップmRNA decay[11]という機構により転写後すぐに分解され、消失するとされています。しかし、この新しいアイソフォームは脳、腎臓、腱、靭帯などの組織で強く発現していました。
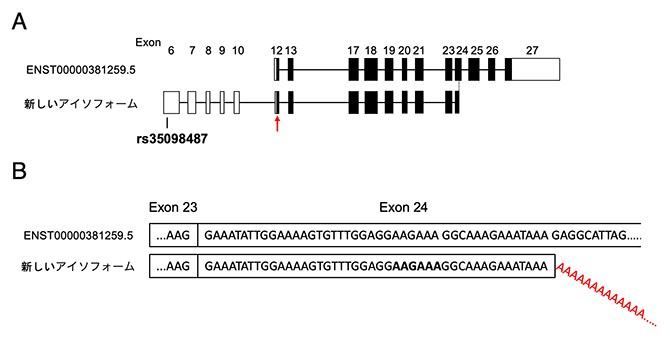
図2 CCDC91遺伝子転写産物の新しいアイソフォームの構造
- (A)公共データベースに載っているCCDC91遺伝子転写産物の標準的アイソフォームのENST00000381259.5(上)と今回発見された新しいアイソフォーム(下)のエクソン構造の図。エクソンの番号は、公共データベースに載っている全てのエクソンについて、5'末端側(図の左側)から順番につけた。白は非翻訳エクソン(UTR)、黒は翻訳エクソンを表す。二つのアイソフォームの転写開始点(赤矢印)は同じだが、新しいアイソフォームは翻訳領域の途中で転写が終了している。OPLLのリスク遺伝子多型のrs35098487は、新しいアイソフォームの最も5'末端側の非翻訳エクソン(5'-UTR)に存在し、既知のエクソン上にはなかった。
- (B)アイソフォームの3'末端の塩基配列。太字はポリA付加シグナルの塩基配列、赤字はポリAの配列を示す。新しいアイソフォームは、エクソンの途中で転写が終了している。ポリA付加部位の5'末端側(左側)には終止コドン(TAA、TGA、TAG)が存在しないため、新しいアイソフォームは「ノンストップmRNA」だと考えられる。
rs35098487は新しいアイソフォームの5'-UTR(5'非翻訳領域)部分に存在しました(図2A)。5'-UTRは遺伝子の転写に関わることが多いため、この遺伝子多型の転写への影響を実験で調べました。すると、rs35098487のOPLLリスクアレル[12]は、非リスクアレルに比べてより強く核内タンパク質と結合し、強い転写活性を示しました。
骨芽細胞は骨形成の主役となる細胞で、間葉系幹細胞から分化します。新しいアイソフォームの発現をOPLLに関係するさまざまな組織・細胞で実験的に調べたところ、骨芽細胞分化で中心的な役割を果たすRUNX2遺伝子と並行して、新しいアイソフォームの発現が骨芽細胞の分化の初期に亢進することが分かりました。間葉系幹細胞やMG-63などの骨形成株化細胞で、新しいアイソフォームの発現をsiRNA[13]で阻害すると、RUNX2遺伝子をはじめとする骨形成関連遺伝子群の発現が低下しました。一方、新しいアイソフォームを過剰に発現させると、骨形成関連遺伝子群の発現、ならびに骨形成が上昇しました。
次に、RNA22 V2とmiRDBによるin silico解析[14]から、新しいアイソフォームはマイクロRNA(miRNA)との結合部位を持ち、MIR890というmiRNAと結合することが予測され、ルシフェラーゼアッセイ[15]でそれが証明されました。MIR890はRUNX2遺伝子の3'-UTR(3'非翻訳領域)と結合し、RUNX2遺伝子の機能を抑制しました。
以上の結果から、CCDC91遺伝子によるOPLLの発症のしやすさのメカニズムは次のように考えられます(図3)。
- (1)CCDC91遺伝子転写産物の新しいアイソフォームは、非翻訳RNAとしてMIR890と直接結合し、MIR890とRUNX2遺伝子の結合を阻害することで、骨芽細胞の分化の初期過程において骨形成を亢進させる。
- (2)リスク遺伝子多型のrs35098487は、新しいアイソフォームの発現を上昇させる。
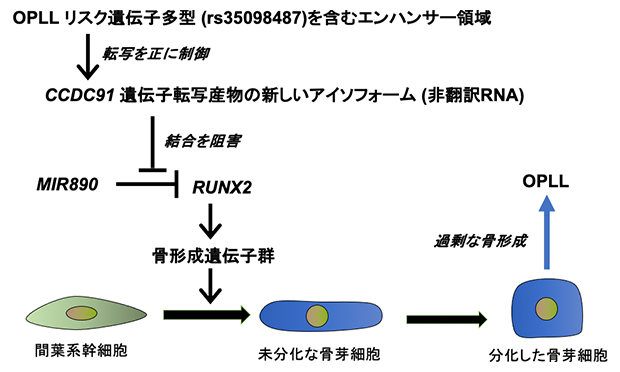
図3 CCDC91遺伝子転写産物の新しいアイソフォームとOPLLリスク遺伝子多型の役割
CCDC91遺伝子転写産物の新しいアイソフォームは、非翻訳RNAとして働く。GWASで相関を示したOPLLリスク遺伝子多型(rs35098487)は、新しいアイソフォームをコードする遺伝子のエクソン1に存在する。エクソン1のゲノムはエンハンサー(遺伝子の転写量を増加させる作用を持つDNA領域)活性を持ち、新しいアイソフォームの発現を正に制御する。そして、新しいアイソフォームはmiRNAと結合するが、そのうちの一つのMIR890は、骨形成のマスター転写因子であるRUNX2遺伝子に結合し、RUNX2遺伝子の機能を阻害する。しかし、新しいアイソフォームの発現が増えると、RUNX2遺伝子は脱阻害され、その下流の骨形成遺伝子群の発現を上昇させ、骨芽細胞の分化を亢進させる。分化した骨芽細胞は過剰な骨形成を引き起こし、OPLLが発症する。
- 注4)2022年11月22日プレスリリース「非翻訳RNAの発現を予測するAIの開発」
今後の期待
これまでさまざまな疾患でGWASが行われてきましたが、実際に疾患感受性遺伝子の同定に成功する例はごく少数でした。しかし今回、AIを活用した統計学的解析による予測と骨代謝学・分子遺伝学に関する専門的な実験による検証により、それが可能になりました。今後、同様のin silicoとin vitroの研究手法の融合アプローチにより、さまざまな疾患感受性遺伝子が発見されると期待できます。
今回の結果は、靭帯細胞に特異的に発現する非翻訳RNAのネットワークが、骨形成の制御を通じて、OPLLの発症・進展に重要な役割を担うことを示しています。今後、CCDC91-MIR890経路の骨形成における役割を詳しく調べることで、OPLLの病態の理解が進み、新しいタイプの治療薬が開発されると期待できます。
補足説明
- 1.ゲノムワイド関連解析(GWAS)
疾患の感受性遺伝子を見つける方法の一つ。ヒトのゲノム全体をカバーする一塩基多型(Single Nucleotide Polymorphism: SNP)を用いて、疾患を持つ群と疾患を持たない群とで、SNPの頻度に差があるかどうかを統計学的に比較する解析方法。GWASはGenome-Wide Association Studyの略。 - 2.非翻訳RNA
DNAから転写されるRNAのうち、メッセンジャーRNA(mRNA)はタンパク質に翻訳される。タンパク質に翻訳されるmRNAに対して、タンパク質に翻訳されないRNAの総称を非翻訳RNAという。非翻訳RNAは、ヒトの発生や恒常性の維持などにおいて、組織や細胞種特異的に作用するさまざまなタイプがあることが報告されている。 - 3.マイクロRNA(miRNA)
細胞内に存在する長さ21~23塩基程度の1本鎖RNA。数百~数千の塩基の一次転写産物から段階的に切り出されて作られる。タンパク質へは翻訳されず、標的となるmRNAの分解、翻訳抑制を通して、遺伝子機能の制御に働く。 - 4.疾患感受性遺伝子
単一遺伝子病の原因遺伝子のように、遺伝子に変異があると必ず発症するというものではなく、変異があると発症しやすくなったり、逆に発症しにくくなったりする遺伝子を指す。リスク遺伝子ともいう。 - 5.間葉系幹細胞
軟骨、骨、脂肪、腱、靭帯などの細胞への分化能を持つ未分化細胞。 - 6.CAGEタグ
CAGE法によって得られた、mRNAの5'末端の約20塩基の配列。転写開始点、転写量など、遺伝子の転写に関するさまざまな情報が得られる。CAGEはCap Analysis of Gene Expressionの略。 - 7.転写産物アイソフォーム
同じ遺伝子から転写された塩基配列の異なる一群のmRNAのこと。選択的スプライシングなどのため、通常、一つの遺伝子から複数の異なるmRNAが生成される。 - 8.リアルタイムPCR法
PCR法では、増幅対象のDNA、DNAポリメラーゼおよび大量のプライマーと呼ばれるオリゴヌクレオチドをあらかじめ混合し、これにDNAポリメラーゼが働くと、結合したプライマーの3'末端を起点として1本鎖部分と相補的なDNAが合成される。DNAが合成された後、再び高温にしてDNA変性から繰り返す。PCR法は、DNA鎖長の違いによる変性とアニーリングの違いを利用して、温度の上下を繰り返すだけでDNA合成を繰り返し、DNAを増幅する技術である。リアルタイムPCR法は蛍光プローブを共存させ、反応の進行を蛍光シグナルの増減を通じてリアルタイムで確認することができる。PCRはPolymerase Chain Reactionの略。 - 9.3'-RACE法
PCR法を応用して、部分的な配列が分かっているmRNAの末端までの配列を同定する方法。5'側の末端を決める5'-RACE法と3'側の末端を決める3'-RACE法がある。RACEは、Rapid amplification of cDNA endsの略。 - 10.終止コドン
タンパク質の中のアミノ酸の並び方は、そのタンパク質の遺伝子(DNA)の中の塩基配列に対応している。三つの塩基がひとまとまりになって一つのアミノ酸に対応しており、この3塩基の並びをコドン(codon)と呼ぶ。64通りのコドンが存在するが、そのうちの61種類のコドンがアミノ酸を意味している。例えばTTTはフェニルアラニンである。残り三つのコドン(TAA、TGA、TAG)はタンパク質合成終了を意味する「終止コドン(stop codon)」であり、mRNAの翻訳される領域の最後に現れる。 - 11.ノンストップmRNA、ノンストップmRNA decay
翻訳の読み枠(reading frame)に終止コドンがなく、翻訳領域の途中で転写が終了し、ポリA(アデノシンが複数連続した配列)が付加されてしまったmRNAをノンストップmRNA(non-stop mRNA)という。この異常なmRNAを転写後に検出し、分解して、翻訳されないようにする機構をノンストップmRNA decayという。 - 12.リスクアレル
疾患感受性遺伝子多型のアレル(対立遺伝子)のうち、疾患感受性(リスク)を高める方のアレルのこと。 - 13.siRNA
siRNAはsmall interfering RNAの略で、21~25塩基対の二本鎖RNA。合成したsiRNAを細胞に取り込ませることにより、相補的な配列を持つ遺伝子の発現を抑制できる。 - 14.RNA22 V2とmiRDBによるin silico解析
ゲノム情報解析のためのオンライン・ツール。RNA22 V2は、ゲノム上のマイクロRNAの結合部位を予測するプログラム。miRDBは、マイクロRNAのターゲット予測と機能アノテーションのためのオンラインデータベース。なお、in silicoは、in vivo(生体内で)やin vitro(試験管内で)などに準じて作られた用語で、「シリコン内で」(コンピュータで)を意味する。 - 15.ルシフェラーゼアッセイ
細胞での転写活性測定法。遺伝子の転写活性は、定量性に優れたレポータータンパク質(ルシフェラーゼ、CAT、βガラクトシダーゼなど)に置き換えて測定することができる。転写調節機能(プロモーター/エンハンサーなど)の機能解析では、ルシフェラーゼは最もポピュラーなレポーターとして広く使用される。実験では、目的のプロモーター領域のDNAをレポーター遺伝子につないだプラスミド(ルシフェラーゼレポーター)を培養細胞に強制発現させ、レポーター活性からプロモーターDNAの転写活性を測定する。
研究チーム
理化学研究所 生命医科学研究センター
骨関節疾患研究チーム
チームリーダー(研究当時) 池川 志郎(イケガワ・シロウ)
副チームリーダー(研究当時)郭 竜(ゴウ・ロン)
(現 西安交通大学(中国)医学部 教授)
研究員 中島 正宏(ナカジマ・マサヒロ)
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(テラオ・チカシ)
客員研究員 小井土 大(コイド・マサル)
(東京大学大学院 新領域創成科学研究科 メディカル情報生命専攻 助教)
研究支援
本研究は、厚生労働科学研究費補助金難治性疾患政策研究事業「脊柱靭帯骨化症に関する調査研究」(研究代表者:筑波大学医学医療系整形外科の山崎 正志 教授)、全国脊柱靭帯骨化症患者家族連絡協議会(増田 靖子 会長)、日本学術振興会(JSPS)科学研究費助成事業若手研究「多因子疾患のエピスタシス効果を同定するためのゲノム配列の深層学習戦略」(20K15773、研究代表者:小井土 大)、基盤研究(B)「運動器疾患のMulti disease GWAS解析」(22H03207、研究代表者:池川 志郎)、基盤研究(A)「エンハンサーの遺伝的発現制御の解明による免疫疾患解析」(20H00462、研究代表者:寺尾 知可史)、日本医療研究開発機構(AMED)難治疾患実用化研究事業「シングルセル統合ゲノミクス解析が解き明かす強皮症の病態基盤の開発」(JP21kk0305013、研究代表者:寺尾 知可史)、ゲノム医療実現推進プラットフォーム・先端ゲノム研究開発「先天的/後天的構造多型に着目した免疫/精神疾患病態解明に関する研究開発」(JP21tm0424220、研究代表者:寺尾 知可史)、革新的がん医療実用化研究事業「体細胞モザイクのがん発症および予後因子としての意義解明の開発」(JP21ck0106642、研究代表者:寺尾 知可史)による支援を受けて行われました。
原論文情報
- Masahiro Nakajima, Masaru Koido, Long Guo, Chikashi Terao, and Shiro Ikegawa, "A novel CCDC91 isoform associated with ossification of the posterior longitudinal ligament of the spine works as a non-coding RNA to regulate osteogenic genes", American Journal of Human Genetics, 10.1016/j.ajhg.2023.03.004
発表者
理化学研究所
生命医科学研究センター 骨関節疾患研究チーム
チームリーダー(研究当時)池川 志郎(イケガワ・シロウ)
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(テラオ・チカシ)
客員研究員 小井土 大(コイド・マサル)
 池川 志郎
池川 志郎
 寺尾 知可史
寺尾 知可史
 小井土 大
小井土 大
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
