2024年11月27日
理化学研究所
東京都医学総合研究所
科学技術振興機構(JST)
FANTOMウェブリソースの最新アップデート
-ノンコーディングRNA・シスエレメントの新データ公開-
理化学研究所(理研)生命医科学研究センター 生命医科学大容量データ技術研究チームの信定 知江 研究員、粕川 雄也 チームリーダー、東京都医学総合研究所 ゲノム医学研究センターの川路 英哉 副センター長らの共同研究グループは、国際共同研究「FANTOM(Functional ANnoTation of Mammalian Genome)プロジェクト」の成果を基に、FANTOMウェブリソースにおけるゲノム上のノンコーディングRNA(ncRNA)[1]の機能情報の拡張と、シスエレメント[2]に関する新たなデータベース注1)を公開しました。
本研究成果は、遺伝子発現制御機構をはじめとする基礎研究に加え、疾患、薬などを対象とした生命科学分野の研究への活用が期待されます。
今回のFANTOMウェブリソースの更新では(1)iPS細胞における長鎖ncRNA[3]の機能情報、(2)クロマチン相互作用による核内長鎖ncRNAの機能情報、(3)今回新たに同定した転写活性に関わるゲノム領域であるシスエレメントの情報、について拡充されました。
本研究は、科学雑誌『Nucleic Acids Research』(2025年データベース特集号)の掲載に先立ち、オンライン版(11月27日付:日本時間11月27日)に掲載されました。
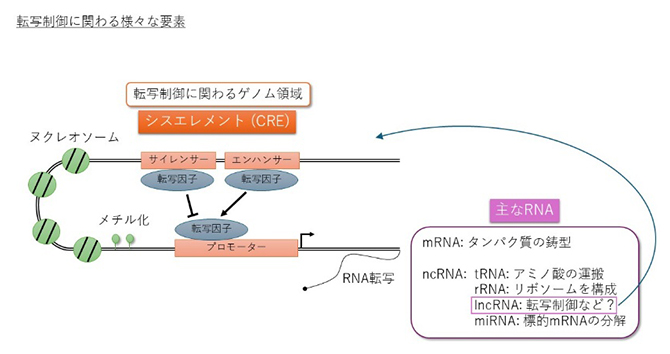
転写制御に関わるさまざまな要素
背景
理研主導で行われている国際共同研究「FANTOM(Functional ANnoTation of Mammalian Genome)プロジェクト」は、20年以上にわたり、新しい技術を取り入れながら、ヒトやマウスを中心とした哺乳動物の大規模転写解析を行い、転写に関するさまざまなデータを提供してきました。
大規模転写解析で決定されるRNA配列には、タンパク質に翻訳されるメッセンジャーRNAや、タンパク質に翻訳されないノンコーディングRNA(ncRNA)など多様なRNAが含まれます。ncRNAのうち、特に約200塩基以上から成る長鎖ncRNAは、ヒトにおいて約28,000種類が見つかっているにもかかわらず、実験的に機能が解明されているものは全体の1%もありません。近年FANTOMプロジェクトではその長鎖ncRNAの機能を明らかにすることに取り組んでおり、最近の成果として長鎖ncRNAの機能不全による遺伝子発現、形質への影響や、長鎖ncRNAのターゲット配列を報告してきました注2、3)。
また、第5期のFANTOMプロジェクト(FANTOM5)では、CAGE法[4]による転写開始点の解析から、転写制御に関わるゲノム上のプロモーター[5]やエンハンサー[5]領域を推定してきました。エンハンサー領域ではその両端において双方向に転写されるエンハンサーRNA(enhancer RNA:eRNA)の存在が知られていたため、FANTOM5ではこの双方向性のeRNAの存在を手掛かりにエンハンサー領域を決定していました。しかし近年、単方向に転写される領域を持つエンハンサーがあることが報告され、今までの解析では見過ごされているエンハンサーが存在することが考えられました。また、FANTOM5以降、第6期のFANTOMプロジェクト(FANTOM6)をはじめとして多くの新たなデータセットが報告されており、転写制御に関わるシスエレメント領域の再検討が求められていました。
- 注2)Yip,C.W., Hon,C.-C., Yasukawa., Sivaraman,D.M., Ramilowski,J.A., Shibayama,Y., Agrawal,S., Prabhu,A.V., Parr,C., Severin,J., et al. (2022) Antisense-oligonucleotide-mediated perturbation of long non-coding RNA reveals functional features in stem cells and across cell types. Cell Reports, 41, 111893.
- 注3)Agrawal,S., Buyan,A., Severin,J., Koido,M., Alam,T., Abugessaisa,I., Chang,H.Y., Dostie,J., Itoh,M., Kere,J., et al. (2024) Annotation of nuclear lncRNAs based on chromatin interactions. PLOS ONE, 19, e0295971.
研究手法と成果
FANTOMウェブリソースの更新として、最近の成果である「iPS細胞における長鎖ncRNAの機能情報」と「クロマチン相互作用による核内長鎖ncRNAの機能情報」について探索したり参照したりできるインタフェース群を開発しました注4)。これらは理研で開発された「ZENBU-Reports」というシステムを用いて開発されています(図1A)。また、解析に用いた元のデータについてはFANTOMのウェブサイト注5)から入手できます。
転写制御に関わるシスエレメント領域の再解析については、東京都医学総合研究所の川路副センター長らが開発した、CAGEデータからプロモーターとエンハンサーを同定する新たな手法を使い、FANTOM5で取得したデータに加えて、新たにFANTOM6で取得したデータや公共のデータベースから収集したデータを用いて統一的な解析を行いました。これにより、ヒトで6,298個のCAGEデータセットから447,315個のヒトのシスエレメント領域を、マウスの1,264個のCAGEデータセットから288,877個のマウスのシスエレメント領域を同定しました。
同定したシスエレメントについて、ゲノム上の位置情報に加え、発現レベルを基にした活性情報、近傍遺伝子や一塩基多型(SNP)[6]などの情報を収集し、新しい統合的なシスエレメントデータベースであるfanta.bioとして公開しました(図1B)。fanta.bioのデータは前述のサイトの他、UCSCゲノムブラウザ・データベース[7]のTrack Hub[7]を介して他の公共データと並べて閲覧することもできます。また、ChIP-Seq[8]の情報を統合したChIP-Atlasデータベース注6)と連携し、シスエレメント領域中の結合転写因子の情報が参照できます。さらに、ENCODEプロジェクト注7)で同定されたシスエレメントとの関連情報など、外部データベースの情報にもリンクしています。これらにより、fanta.bioはシスエレメントの統合的な情報を提供するデータベースになっています。
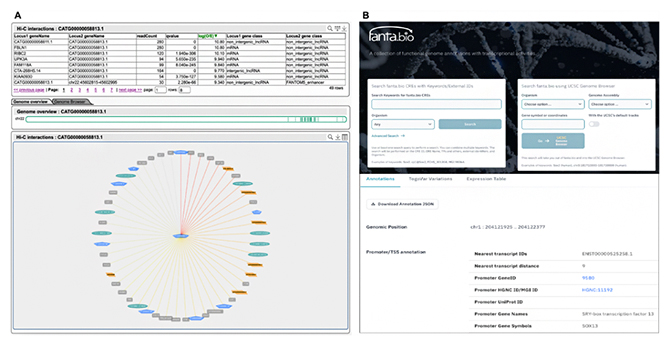
図1 長鎖ncRNAの機能情報とシスエレメントのデータベース
- (A)クロマチン相互作用による核内長鎖ncRNAの機能情報のデータ(一部)をZENBU-Reportsで表示したところ。
- (B)シスエレメントのデータベースであるfanta.bioのトップページ(上)とシスエレメントの情報を記載したページ(下)。
- 注4)今回更新の内容をまとめたFANTOMウェブサイト内のページ(英語)
- 注5)FANTOM6のデータファイル公開ページ(英語)
- 注6)ChIP-Atlasデータベース(英語)
- 注7)ENCODEプロジェクト(英語)
今後の期待
FANTOMプロジェクトは最新技術を取り込みながら、さまざまなアプローチでデータの拡充を続けており、今後もFANTOMウェブリソースを通じてその成果を公開し、転写制御に関わる研究の重要なデータリソースとして国内から世界中の研究コミュニティーへ貢献し続けられることが期待されます。
本研究成果は、ヒトやマウスの長鎖ノンコーディングRNA(ncRNA)の機能やシスエレメントの包括的な情報を広く研究コミュニティーに提供するものです。
転写制御機構の研究は疾患の解明、薬の開発などの基礎となるものであり、生命科学分野における研究への活用が大いに期待されます。
補足説明
- 1.ノンコーディングRNA(ncRNA)
ゲノムから転写されるRNAのうち、タンパク質合成の鋳型となる領域を含まないRNAのこと。ncはnoncodingの略。 - 2.シスエレメント
転写制御に関わるゲノム上の領域。転写開始に関与するプロモーター、転写を促進するエンハンサー、転写活性を調節するインシュレーターなどがある。 - 3.長鎖ncRNA
タンパク質合成の鋳型とならないノンコーディングRNAの1種。一般に約200塩基以上のものを指す。 - 4.CAGE法
理研が開発した転写産物の5'末端(転写開始点)の塩基配列を決定する実験手法。転写量の測定値は転写活性の指標となる。CAGEはCap Analysis of Gene Expressionの略。 - 5.プロモーター、エンハンサー
ゲノムDNA上で転写される領域の近くにあり、遺伝子を発現させる機能を持つ部分をプロモーター(領域)、主に遺伝子の上流や下流に位置し、遺伝子の転写効率を変化させるDNAの配列のうち、転写効率を高める領域をエンハンサー(領域)という。 - 6.一塩基多型(SNP)
ヒトゲノムの配列上に個体間で一つの塩基の違いがある箇所。身長などの形質の差や、疾患に関連しているものなどが知られている。SNPはSingle Nucleotide Polymorphismの略。 - 7.UCSCゲノムブラウザ・データベース、Track Hub
米国カリフォルニア大学サンタクルーズ校(UCSC)が開発、維持しているゲノムデータベースおよびデータブラウザ。ヒトをはじめとしてさまざまな生物のゲノム地図を閲覧できる。世界中の研究者が標準的に用いるデータベースの一つであり、その公式ミラーサイトの一つが理研で運用されている注8)。Track Hub機能を使うことでゲノムの座標情報にひもづいた各種アノテーション情報を追加でき、fanta.bioではこの機能を使ってシスエレメント領域や関連情報を参照できるようにしている。 - 8.ChIP-Seq
タンパク質特異的に結合する抗体を用いて免疫沈降させることで、転写因子や特定のメチル化パターンを示すヒストンタンパク質とゲノムDNAとの相互作用を、シーケンシング技術を用いて調べる手法。転写因子結合部位やDNAメチル化パターンの同定ができる。
- 注8)2016年6月17日お知らせ「ゲノムブラウザ・データベースの公式ミラーを開設」
共同研究グループ
理化学研究所
生命医科学研究センター
生命医科学大容量データ技術研究チーム
チームリーダー 粕川 雄也(カスカワ・タケヤ)
研究員 信定 知江(ノブサダ・トモエ)
客員研究員イマド・アブケセーサ(Imad Abugessaisa)
技師 ジェシカ・セヴェリン(Jessica Severin)
技師 スコット・ウォーカー(Scott Walker)
上級テクニカルスタッフ 長谷川 哲(ハセガワ・アキラ)
テクニカルスタッフⅠ 近藤 敦(コンドウ・アツシ)
研究パートタイマーⅠ ニシャード・タラハト(Nishad Thalhath)
トランスクリプトーム研究チーム
チームリーダー ピエロ・カルニンチ(Piero Carninci)
遺伝子制御回路研究チーム
チームリーダー ジェイ・W・シン(Jay W. Shin)
上級研究員 チーワイ・イップ(Chi Wai Yip)
上級技師 安澤 加代子(ヤスザワ・カヨコ)
応用計算ゲノミクス研究チーム
チームリーダー ミヒル・デ・ホーン(Michiel de Hoon)
研究員 ソムヤ・アグラワァル(Saumya Agrawal)
遺伝子制御ゲノミクス研究チーム
チームリーダー ヂョン・チョウ・ホン(Chung Chau Hon)
応用ゲノム解析技術研究チーム
専門技術員 田上 道平(タガミ・ミチヒラ)
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(テラオ・チカシ)
客員研究員 小井土 大(コイド・マサル)
バイオリソース研究センター 統合情報開発室
室長 桝屋 啓志(マスヤ・ヒロシ)
開発研究員 高田 豊行(タカダ・トヨユキ)
東京都医学総合研究所 ゲノム医学研究センター
副センター長 川路 英哉(カワジ・ヒデヤ)
研究員 井手 聖(イデ・サトル)
熊本大学 生命資源研究支援センター
教授 沖 真弥(オキ・シンヤ)
研究支援
本研究は、科学技術振興機構(JST)統合化推進プログラム「統合的な転写制御データ基盤の構築(研究代表者:粕川雄也、JPMJND2202)」、日本医療研究開発機構(AMED)ゲノム創薬基盤推進研究事業「RNA標的医薬創出に資する、疾患RNA分子完全長一次構造に関するデータ基盤の構築(研究代表者:川路英哉、23kk0305024)」、同ゲノム医療実現バイオバンク利活用プログラム「先天的/後天的構造多型に着目した免疫/精神疾患病態解明に関する研究開発(研究開発代表者:寺尾知可史、21tm0424220)」「炎症性関節炎の統合ゲノミクス解析(研究開発代表者:寺尾知可史、23tm0424225)」、同免疫アレルギー疾患実用化研究事業「先天的・後天的ゲノム情報と臨床情報を用いた関節リウマチの層別化基盤の構築(研究開発代表者:寺尾知可史、23ek0410114)」による助成を受けて行われました。
原論文情報
- Tomoe Nobusada, Chi Wai Yip, Saumya Agrawal, Jessica Severin, Imad Abugessaisa, Akira Hasegawa, Chung Chau Hon, Satoru Ide, Masaru Koido, Atsushi Kondo, Hiroshi Masuya, Shinya Oki, Michihira Tagami, Toyoyuki Takada, Chikashi Terao, Nishad Thalhath, Scott Walker, Kayoko Yasuzawa, Jay Shin, Michiel de Hoon, Piero Carninci, Hideya Kawaji, Takeya Kasukawa, "Update of the FANTOM web resource: enhancement for studying noncoding genomes", Nucleic Acids Research, 10.1093/nar/gkae1047
発表者
理化学研究所
生命医科学研究センター 生命医科学大容量データ技術研究チーム
研究員 信定 知江(ノブサダ・トモエ)
チームリーダー 粕川雄也(カスカワ・タケヤ)
東京都医学総合研究所 ゲノム医学研究センター
副センター長 川路 英哉(カワジ・ヒデヤ)
 信定 知江
信定 知江
 粕川 雄也
粕川 雄也
 川路 英哉
川路 英哉
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
東京都医学総合研究所 事務局研究推進課
Tel: 03-5316-3109
Email: koho [at] igakuken.or.jp
科学技術振興機構 広報課
Tel: 03-5214-8404
Email: jstkoho [at] jst.go.jp
JST事業に関する問い合わせ
科学技術振興機構 情報基盤事業部 NBDC事業推進室
川口 貴史(カワグチ・タカフミ)
Tel: 03-5214-8491
Email: info [at] biosciencedbc.jp
※上記の[at]は@に置き換えてください。
