理化学研究所(理研)生命医科学研究センター ゲノム免疫生物学理研白眉研究チームのニコラス・パリッシュ 理研白眉研究チームリーダー、小嶋 将平 基礎科学特別研究員らの共同研究グループは、「転移因子多型[1]」が皮膚病変などの遺伝的リスクであることを明らかにしました。
本研究成果は、これまで見過ごされてきたゲノム多型である転移因子多型に光を当てたもので、患者一人一人に合った診断・予防・治療を行う「ゲノム医療」の発展に貢献すると期待できます。
疾患の遺伝的リスクを調べるゲノムワイド関連解析(GWAS)[2]には、一塩基多型[2]を用いるのが一般的で、技術的な問題から転移因子多型はこれまで用いられてきませんでした。
今回、共同研究グループはヒトゲノムデータから転移因子多型を正確に同定するソフトウェアを開発し、転移因子多型を用いたGWASを実施しました。バイオバンク・ジャパン(BBJ)[3]により収集されたおよそ18万人における42疾患のGWASの結果、五つの転移因子多型が三つの疾患(ケロイド、2型糖尿病、前立腺がん)と強く関連することが分かりました。さらに、ヒト線維芽細胞を用いた転移因子のノックアウト(欠損)実験を行い、皮膚病変であるケロイド[4]の重症化の原因遺伝子の一つであるNEDD4に存在する転移因子の挿入が、ケロイドの遺伝的リスクであることを明らかにしました。
本研究は、科学雑誌『Nature Genetics』オンライン版(5月11日付:日本時間5月12日)に掲載されました。
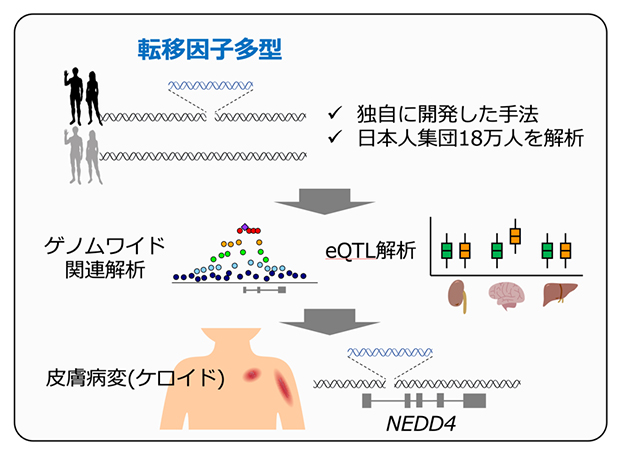
転移因子多型を遺伝統計解析に取り入れ、ケロイドのリスクとなる変異を特定
背景
ゲノムワイド関連解析(GWAS)やeQTL[5]解析といった遺伝統計解析には、一塩基多型(SNP)を用いるのが一般的ですが、長鎖配列の挿入・欠損に由来する多型も異常な遺伝子発現や疾患の原因となる可能性があります。しかし、このような多型は、解析手法の技術的な問題から遺伝統計解析に用いられることはありませんでした。従って、遺伝統計解析に一塩基多型以外の変異を取り入れれば、これまで見過ごされてきた遺伝的な疾患リスクの発見につながる可能性があります。
「転移因子(トランスポゾン)」はゲノムに存在し、ゲノムの至る所に挿入される長鎖配列です。個人間での転移因子の有無である「転移因子多型」は、ヒトの長鎖配列の挿入多型のおよそ25%を占めます。しかし、これまで転移因子多型を正確に同定する手法がなかったため、転移因子が遺伝子発現や疾患に与える影響は不明のままでした。
このような背景から本研究では、転移因子多型を正確に同定するソフトウェアを作成し、遺伝統計手法に転移因子多型を取り入れることで、①ヒト集団間における違い、②遺伝子発現への影響、③疾患リスクを調べました。
研究手法と成果
共同研究グループはまず、転移因子多型を正確に同定するソフトウェアを作成しました。検出感度(効率)は80%以上、偽陽性率は6%以下、多型の量の決定精度(正確性)は94%以上と、ヒトゲノムに存在する転移因子多型の大半を正確に検出できます。この同定精度は転移因子多型を遺伝統計解析に用いるのに十分であり、DNAマイクロアレイ[6]データを用いた転移因子多型の統計的推定(インピューテーション[7])も可能です。
開発したソフトウェアを、国際1000人ゲノムプロジェクト[8]により収集された多様なヒト集団由来の2,504人に適用し、転移因子多型を調べました。その結果、日本人集団を含む東アジア人集団において、Alu[9]と呼ばれる転移因子の挿入はアメリカ人集団・ヨーロッパ人集団・南アジア人集団とほぼ同じでしたが、LINE-1[9]やSVA[9]と呼ばれる転移因子の挿入はアメリカ人集団・ヨーロッパ人集団・南アジア人集団よりも多いことが分かりました(図1)。このことから、ヒト集団間において転移因子の転移活性は異なると考えられます。
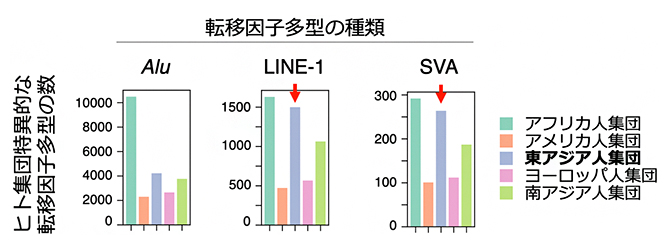
図1 ヒト集団特異的な転移因子多型の数
五つのヒト集団において転移因子多型を解析し、特定のヒト集団にのみ見つかった転移因子多型の数を数えた。青色のバーで示す東アジア人集団では、アメリカ人集団、ヨーロッパ人集団や南アジア人集団と比較し、LINE-1やSVAの多型が多い。
次に、転移因子多型を含むeQTLを解析しました。国際プロジェクトGTEx[10]により収集された838人において、全ゲノムデータから転移因子多型を統計的に推定し、49のヒト組織においてeQTL解析を行いました。その結果、遺伝子発現の変化と相関する1,073のeQTLが見つかりました(図2a)。一塩基多型と転移因子多型のどちらがeQTLに多く見つかるかを検定した結果、転移因子多型の方がより高頻度にeQTLに見つかりました(図2b)。このことから、転移因子多型は遺伝子発現へ頻繁に影響すると考えられます。
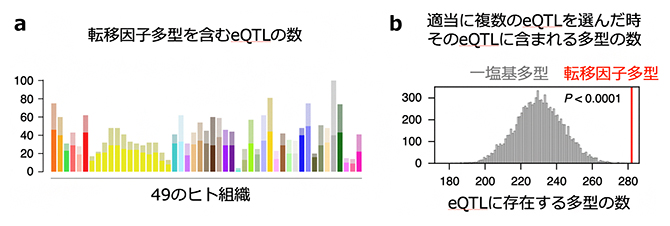
図2 転移因子多型を含むeQTL
- (a)49のヒト組織において検出された、転移因子多型を含むeQTLの数。合計1,073のeQTLが見つかった。
- (b)転移因子多型は一塩基多型と比較して、eQTLに高頻度に見つかる。精巣組織において検出されたeQTLに転移因子多型が多く存在するかを確かめるため、パーミューテーション検定を行った。本検定では、遺伝子からの距離、周辺遺伝子数などを考慮の上、ランダムにeQTLを複数個選択し、そのeQTLにどのくらいの一塩基多型が含まれているかを調べた。その結果、一塩基多型と比較して転移因子多型が精巣のeQTLに高頻度に見つかることが明らかとなった。P値は、1万回のパーミューテーションを行ったときの経験的確率を示す。
最後に、バイオバンク・ジャパン(BBJ)により収集された日本人集団由来の被験者およそ18万人において、DNAマイクロアレイデータから転移因子多型を統計的に推定し、42疾患のGWASを行いました。その結果、五つの転移因子多型(Alu三つとLINE-1二つ)が三つの疾患(ケロイド、2型糖尿病、前立腺がん)と強く関連することが明らかになりました。検出された転移因子多型のうち、一つ(Alu)は東アジア人集団に特異的な多型であり、人類史において近年に挿入されたヒト集団特異的な転移因子も疾患に関わると考えられます。
転移因子多型が疾患リスクとなるかを検証するため、ケロイドと呼ばれる皮膚病変と強い関連が見られた転移因子であるLINE-1の挿入に着目しました。このLINE-1の挿入は、ケロイド重症化の原因遺伝子の一つであるNEDD4注1)のイントロン(タンパク質に翻訳されない領域)に見つかりました(図3左)。ゲノム編集技術[11]により、LINE-1挿入をノックアウトアウト(欠損)したヒト線維芽細胞を作製し、LINE-1挿入がNEDD4の発現へ与える影響を調べた結果、LINE-1挿入はNEDD4の発現を上昇させることが明らかになりました(図3右)。このことから、LINE-1挿入がNEDD4発現を上昇させ皮膚病変の重症化を引き起こす、ケロイドのリスク因子であると考えられます。
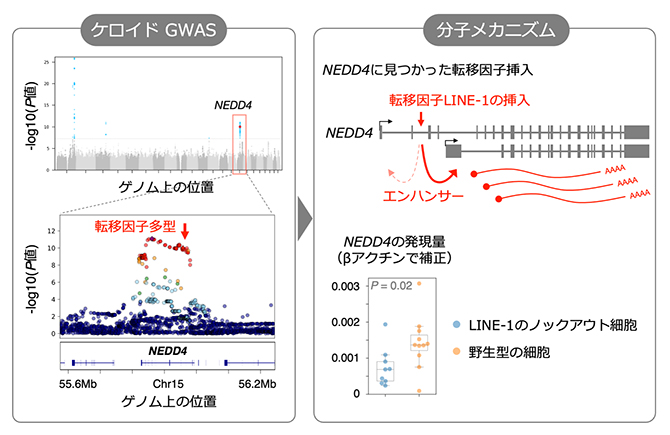
図3 ケロイド重症化の原因遺伝子の一つであるNEDD4の発現を上昇させる転移因子挿入
- (左)バイオバンク・ジャパンにより収集されたおよそ18万人のゲノムデータを使用したGWASから、NEDD4のイントロン(タンパク質に翻訳されない領域)に存在する転移因子挿入(LINE-1の挿入)がケロイドの発生や病態と関連することが明らかになった。
- (右)NEDD4に存在するLINE-1挿入はNEDD4の転写産物(mRNA)を増加させるエンハンサーとして機能する。ヒト線維芽細胞におけるLINE-1挿入のノックアウト(欠損)実験により、LINE-1の挿入はNEDD4の発現を上昇させることが分かった。P値はt-testの結果を示す。
- 注1)Fujita M. et al, NEDD4 Is Involved in Inflammation Development during Keloid Formation. J. Invest. Dermatol., 139(2), 333-341 (2019).
今後の期待
本研究成果は、これまで見過ごされてきたゲノム変異に光を当てる研究で、本研究で開発したソフトウェアがさまざまな疾患バイオバンクにおいて用いられることで、疾患の原因となるゲノム変異の特定を加速させると期待できます。また、このような疾患の遺伝要因の特定は、患者一人一人に合った診断・予防・治療を行う「ゲノム医療」の発展に貢献すると期待できます。
また、日本人集団から見つかった転移因子多型の情報をバイオサイエンスデータベースセンター注2)(NBDC、Research ID: hum0014.v28)において、GWASの解析結果を理研が独自に構築した日本人集団ゲノム関連解析情報データベース「Jenger注3)」においてそれぞれ公開しました。今回の解析結果をもとに多くの研究が展開され、さらなる研究成果につながることも期待できます。
補足説明
- 1.転移因子多型
転移因子はゲノムに存在し、複製されゲノムの異なる場所に挿入される配列で、「動く遺伝子」や「トランスポゾン」などと呼ばれる。ゲノムの至る所に挿入される変異源で、遺伝子を壊したり遺伝子発現を変えたりする場合がある。人類史において近年に起きた挿入に由来する転移因子配列は一部のヒトのゲノムにしか見つからず、そのような転移因子挿入の個人間の違いを転移因子多型と呼ぶ。 - 2.ゲノムワイド関連解析(GWAS)、一塩基多型
一塩基多型とは、ゲノムの個人間の違いのうち、A、C、T、Gからなるヒトゲノム塩基配列の1カ所の違いを指す。ゲノムワイド関連解析(GWAS)は形質に対する遺伝的関連を知るための手法であり、一塩基多型を用いて解析するものが一般的である。疾患の有無や量的形質などを目的変数、多型の量的情報や各種共変量を説明変数にしてモデル化し、多型の関連を評価する。GWASはGenome-Wide Association Studyの略。 - 3.バイオバンク・ジャパン(BBJ)
日本人集団約27万人を対象とした疾患バイオバンク。日本医療研究開発機構のゲノム医療実現バイオバンク利活用プログラム「利活用を目的とした日本疾患バイオバンクの運営・管理」において運営され、DNAサンプルや血清サンプルを臨床情報と共に保管し、研究者へ提供している。2003年から東京大学医科学研究所内に設置されている。詳細はバイオバンク・ジャパンのウェブサイトを参照。 - 4.ケロイド
皮膚の傷が硬化して盛り上がり、かゆみや痛みの症状が出た状態のこと。外傷・にきび・手術による創傷などの皮膚の損傷が発生のきっかけとなる。ヒト線維芽細胞の増殖がケロイドの形成につながる。 - 5.eQTL
遺伝子の発現量の個人差に関与するゲノム領域。eQTLはexpression Quantitative Trait Locusの略。 - 6.DNAマイクロアレイ
遺伝的多型(主に一塩基多型)を検出するための分析器具。基板の上にDNAの部分配列が高密度に配置・固定されており、数十万から数百万の遺伝的多型の検出が可能である。 - 7.インピューテーション
DNAマイクロアレイで数十万から数百万カ所の一塩基多型の遺伝型を測定した後に、得られた遺伝型を用いて、実験的に測定していない遺伝的変異を推定する遺伝統計学的手法。被検者の全ゲノムシークエンス解析を行う必要がなく、時間や費用が抑えられるというメリットがある。 - 8.国際1000人ゲノムプロジェクト
ヒト集団間の遺伝的多様性を調べることを目指し、世界各地のヒトの全ゲノムシークエンスを行った国際研究計画。現在、多様なヒト集団由来の3,000人以上の高カバレッジ全ゲノムデータを公開している。 - 9.Alu、LINE-1、SVA
ヒトゲノムに存在し、現在も転移活性がある転移因子の種類。Aluは約300塩基長の大きさで、1人のゲノムにおよそ2,500コピーの多型が見られる。LINE-1は約6,000塩基長で1人のゲノムにおよそ500コピーの多型が見られ、SVAは約2,000塩基長で1人のゲノムにおよそ100コピーの多型が見られる。 - 10.GTEx
ヒトの組織特異的な遺伝子発現量とゲノム多型に関する大規模なデータ構築を目指している国際プロジェクト。死後のドナーから臓器や組織の提供を受け、ヒトの組織ごとの遺伝子発現量を解析しデータを集積している。GTExはGenotype-Tissue Expressionの略。 - 11.ゲノム編集技術
特定のDNA配列を切断できる人工ヌクレアーゼによりゲノムDNAを切断して、ノックアウトやノックインなどの遺伝子改変を行う技術。
共同研究グループ
理化学研究所
生命医科学研究センター
ゲノム免疫生物学理研白眉研究チーム
理研白眉研究チームリーダー Nicholas F. Parrish(ニコラス・パリッシュ)
基礎科学特別研究員 小嶋 将平(コジマ・ショウヘイ)
研究パートタイマー(研究当時)何 美玲(カ・ミレイ)
大学院生(研究当時)齋藤 優花(サイトウ・ユウカ)
テクニカルスタッフ(研究当時)Erica H. Parrish(エリカ・パリッシュ)
テクニカルスタッフ 藤井 麻美(フジイ・アサミ)
基礎科学特別研究員 Steven M. Heaton(スティーブン・ヒーテン)
研究員 小出 りえ(コイデ・リエ)
特別研究員(研究当時)Anselmo J. Kamada(アンセルモ・鎌田)
基盤技術開発研究チーム
チームリーダー 桃沢 幸秀(モモザワ・ユキヒデ)
テクニカルスタッフ(研究当時)遠藤 ミキ子(エンドウ・ミキコ)
テクニカルスタッフ(研究当時)髙田 定暁(タカタ・サダアキ)
テクニカルスタッフ(研究当時)水越 美咲(ミズコシ・ミサキ)
ゲノム解析応用研究チーム
チームリーダー 寺尾 知可史(テラオ・チカシ)
特別研究員 曳野 圭子(ヒキノ・ケイコ)
循環器ゲノミクス・インフォマティクス研究チーム
チームリーダー 伊藤 薫(イトウ・カオル)
客員研究員 小山 智史(コヤマ・サトシ)
ヒト免疫遺伝研究チーム
チームリーダー 石垣 和慶(イシガキ・カズヨシ)
理研-IFOMがんゲノミクス連携研究チーム
チームリーダー 村川 泰裕(ムラカワ・ヤスヒロ)
バイオリソース研究センター 細胞材料開発室
室長 中村 幸夫(ナカムラ・ユキオ)
上級技師 野口 道也(ノグチ・ミチヤ)
東京大学大学院 新領域創成科 学研究科メディカル情報生命専攻
複雑形質ゲノム解析分野
教授 鎌谷 洋一郎(カマタニ・ヨウイチロウ)
早稲田大学 理工学術院 先進理工学部
教授 浜田 道昭(ハマダ・ミチアキ)
大学院生 武田 淳志(タケダ・アツシ)
研究支援
本研究は、日本学術振興会(JSPS)科学研究費助成事業基盤研究(B)「ヘルペスウイルスのゲノムへの組み込みと再活性化:大規模解析から探る疾患との関連(研究代表者:Nicholas F. Parrish)」、同基盤研究(S)「ゲノム免疫:内在性ウイルスの抗ウイルス活性の動作原理解明と機能資源としての確保(研究代表者:朝長啓造、研究分担者:Nicholas F. Parrish)」、同若手研究「転移因子の挿入多型と疾患とのゲノムワイド関連解析(研究代表者:小嶋将平)」、公益財団法人痛風・尿酸財団研究助成「ゲノム転移因子由来のゲノム構造多型が引き起こす尿酸値異常:20万人の日本人ゲノムを用いたゲノムワイド関連解析(研究代表者:Nicholas F. Parrish)」、理化学研究所奨励課題「Ongoing virus integration in the diverse human genomes: comprehensive survey and functional implication(研究代表者:小嶋将平)」「Detection and experimental infection of endogenous viruses in human cell lines(研究代表者:野口道也)」、理化学研究所国際戦略に基づく第4期中長期計画における戦略的な研究パートナーとの国際連携事業(研究代表者:Nicholas F. Parrish)、日本医療研究開発機構(AMED)ゲノム研究バイオバンク事業「利活用を目的とした日本疾患バイオバンクの運営・管理(研究代表者:山梨裕司)」による助成を受けて行われました。
原論文情報
- Shohei Kojima*, Satoshi Koyama, Mirei Ka, Yuka Saito, Erica H. Parrish, Mikiko Endo, Sadaaki Takata, Misaki Mizukoshi, Keiko Hikino, Atsushi Takeda, Asami F. Gelinas, Steven M. Heaton, Rie Koide, Anselmo J. Kamada, Michiya Noguchi, Michiaki Hamada, Biobank Japan Project Consortium, Yoichiro Kamatani, Yasuhiro Murakawa, Kazuyoshi Ishigaki, Yukio Nakamura, Kaoru Ito, Chikashi Terao, Yukihide Momozawa, Nicholas F. Parrish*, "Mobile element variation contributes to population-specific genome diversification, gene regulation, and disease risk", Nature Genetics, 10.1038/s41588-023-01390-2
発表者
理化学研究所
生命医科学研究センター ゲノム免疫生物学理研白眉研究チーム
理研白眉研究チームリーダー Nicholas F. Parrish(ニコラス・パリッシュ)
基礎科学特別研究員 小嶋 将平(コジマ・ショウヘイ)
 Nicholas F. Parrish
Nicholas F. Parrish
 小嶋 将平
小嶋 将平
報道担当
理化学研究所 広報室 報道担当
お問い合わせフォーム
