理化学研究所(理研)生命機能科学研究センター発生エピジェネティクス研究チームの三浦尚研究員、平谷伊智朗チームリーダーらの共同研究チーム※は、マウスES細胞[1]の分化[2]に伴う染色体の時間的・空間的な構造変化が、トポロジカルドメイン(TAD)[3]と呼ばれる約1Mb(メガベース=100万塩基対[4])のDNAの塊を単位とする核内配置変化であることを、1細胞レベルで突き止めました。
本研究成果は、哺乳類染色体の三次元構造の構築原理に迫るものであり、染色体の構造変化と遺伝子発現[5]制御の統合的な理解にもつながると期待できます。
哺乳類細胞では、染色体の1本1本は、TADが数珠つながりになった形で核内に収納されています。核内での染色体の形や位置は細胞種によって異なり、細胞分化の際には染色体構造が変化すると考えられますが、TADのようなMbレベルの階層でどのように変化するかは、全く分かっていませんでした。
今回、共同研究チームは、マウスES細胞の分化に伴う染色体の三次元構造変化を調べ、これがTADを単位とする核内配置の変化であることを1細胞レベルで突き止めました。この核内配置の変化は染色体上のさまざまな領域で生じ、その領域の遺伝子発現の活性化とよく対応し、しかも核内配置変化が遺伝子発現の活性化よりも先に起きることも分かりました。このことから、染色体の三次元構造変化を調べることで、将来の遺伝子発現変化を予測できる可能性が示唆されました。
本研究は、英国の科学雑誌『Nature Genetics』の掲載に先立ち、オンライン版(8月12日付け:日本時間8月13日)に掲載されます。
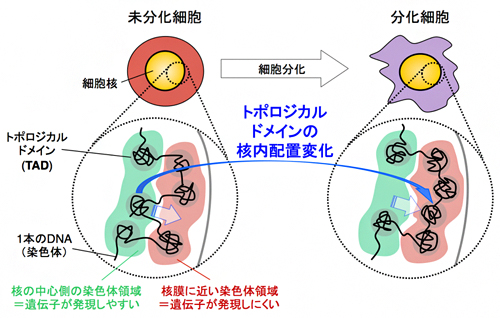
図 細胞分化に伴う哺乳類染色体の三次元構造変化
※共同研究チーム
理化学研究所 生命機能科学研究センター 発生エピジェネティクス研究チーム
チームリーダー 平谷 伊智朗(ひらたに いちろう)
研究員 三浦 尚(みうら ひさし)
基礎科学特別研究員 高橋 沙央里(たかはし さおり)
研究員 ラウィン・プーンパーム(Rawin Poonperm)
テクニカルスタッフⅠ 谷川 明恵(たにがわ あきえ)
三重大学大学院 生物資源学研究科 生命機能化学講座 分子細胞生物学分野
准教授 竹林 慎一郎(たけばやし しんいちろう)
※研究支援
本研究は、日本学術振興会(JSPS)科学研究費補助金若手研究「1細胞DNA複製タイミング解析による発生分化過程の核内コンパートメント動態予測(研究代表者:高橋沙央里)」、同新学術領域研究(研究領域提案型)「染色体機能ドメインの可塑性とその意義(研究代表者:竹林慎一郎)」、同新学術領域研究(研究領域提案型)「細胞分化にともなうクロマチンポテンシャルの変化とその分子基盤(研究分担者:平谷伊智朗)」、科学技術振興機構(JST)戦略的創造研究推進事業個人型研究(さきがけ)の研究領域「エピジェネティクスの制御と生命機能(研究総括:向井常博)」の研究課題「三胚葉分化直前の条件的ヘテロクロマチン形成の発生生物学的意義(研究者:平谷伊智朗)」による支援を受けて行われました。
背景
生命の設計図であるゲノムDNA[6]は、マウスでは40本、ヒトでは46本のDNA分子に分かれて細胞核内に存在しています。各々のDNA分子は数千万~数億塩基対の長さを持ち、タンパク質との複合体(クロマチン[7])を形成して、細胞核内で高度に折り畳まれて各々の染色体を形作っています。なお、かつては「染色体」は細胞周期[8]の分裂期[8]に現れる明瞭な構造体に付けられた用語でしたが、現在では、一般的に間期[8]の時期も含めたクロマチン構造のことを指します。本研究で言及する「染色体の形」とは、間期の染色体のことです。
染色体の構造には階層があります。最も小さい階層には、DNA分子の基本単位であるヌクレオチド[9](1塩基)や、クロマチンの基本単位であるヌクレオソーム[10](約146塩基対のDNAと8個のヒストンタンパク質)といった構造が存在します(図1)。より大きな階層になると、約1Mb(メガベース=100万塩基対)長のDNAが球状に折り畳まれた「トポロジカルドメイン(TAD)」と呼ばれる構造があり、複数のTADがさらに数珠つながりになって空間的にまとまった配置をとることが分かっています(図1)。この配置はTAD内に存在する遺伝子の発現と密接な関わりがあるとされ、遺伝子がよく転写[5]されているTADが集まったものをAコンパートメント[11]、転写されていないTADが集まったものをBコンパートメント[11]と呼んでいます(図1)。各階層の特徴的な構造が積み重なって形作られた染色体という巨大な三次元構造が、遺伝子の発現や細胞の分化とどのような関係にあるのかを解明することは、真核細胞の機能を理解する上で重要なテーマの一つです。
近年、Hi-C法[12]などゲノム全体にわたってクロマチンの核内構造を解析する手法の開発が進んだ結果、同一の生物種であればTADとその境界の位置は細胞種によらず一定であるのに対し、A/Bコンパートメント[11]の分布は細胞種に特異的であると考えられるようになりました。これが正しければ、細胞種が変わるとき、すなわち細胞分化の際には、Aコンパートメントに含まれていたTADがBコンパートメントに変わるといった変化が生じるはずです。しかし、これを詳しく調べた報告はなく、その実態は全く分かっていませんでした。
そこで、共同研究チームは、Hi-C法を用いてマウスES細胞の分化に伴う染色体の三次元構造変化の詳細な全ゲノム解析[13]を行いました。特に、核内コンパートメント[11]の分布変化という観点から詳しく解析を行い、これと遺伝子発現やDNA複製[14]といったゲノム機能の変化との関係を調べました。さらに、Hi-C法は細胞集団を対象とした解析手法であるため、得られた知見が1細胞レベルでも成り立つかについて、最近独自に開発した1細胞全ゲノム解析法であるscRepli-seq法[15]も取り入れて研究を進めました注1)。
注1)2019年2月26日プレスリリース「ゲノムDNA複製の真の姿を捉えた」
研究手法と成果
細胞分化に伴う染色体の三次元構造変化を1細胞レベルで経時的に追跡するには、培養下の細胞を均一に分化できる実験系が必要です。先行研究により、核内コンパートメントに大きな変化が見られる時期として、マウスの胚発生初期に起きるエピブラスト[16]の分化の時期が考えられました注2)。そこで、この時期の細胞分化過程を試験管内で再現するため、まずマウスES細胞を2日間かけてエピブラスト様細胞(EpiLC)[17]に分化させました。そして、このEpiLCにSFEBq法(無血清凝集浮遊培養法)[18]という方法論を適用することで、7日間でマウスES細胞をエピブラスト様の状態を経て、均一に神経前駆細胞へ分化誘導させる実験系の構築に成功しました(図2)。
この実験系を用いて、マウスES細胞分化過程において経時的にHi-C解析を行ったところ、分化前はAコンパートメントであった領域が、分化後にはBコンパートメントに変化(あるいはその逆)する様子が、ゲノム上のさまざまな領域で認められました(図3)。核内コンパートメントの分布はDNA複製タイミング[14](ゲノムDNA複製の際、どの領域から順に複製が進行するかという順序)とよく相関し、Aコンパートメントは細胞周期のS期[8]前半に複製される領域と、BコンパートメントはS期後半に複製される領域とそれぞれ一致する傾向を示すことが知られています。今回の実験系で再現した細胞分化の過程でも、核内コンパートメントとDNA複製タイミングは予想通り高い相関を示し、さらに、A/Bコンパートメントが入れ換わる領域と複製タイミングが変化する領域はゲノム上で重複しており、両者が協調的に変化していることが分かりました(図3、A/Bコンパートメントの赤と緑の色分けと、DNA複製タイミングの青と緑の色分けを比較)。また、核内コンパートメントが変化するゲノム領域は、複製タイミングに加えて、核内配置、特に核ラミナ[19](核内膜タンパク質)との距離が変化することも確認できました(図3、核ラミナ結合)。このことから、Hi-Cで観察される核内コンパートメント変化が、核内空間における染色体の物理的な動きであることが強く示唆されました。
このように、核内コンパートメント変化は、ゲノムのさまざまな形質の変化を伴います。AコンパートメントとBコンパートメントは、それぞれ遺伝子がよく転写されている領域と転写が抑えられている領域に対応しており、これらの変化のうち何が原因で何が結果であるかを明らかにするのは重要です。その糸口をつかむため、核内コンパートメント、複製タイミング、遺伝子発現の三者の変化の時間的な関係を調べたところ、特にBコンパートメントからAコンパートメントへの変化が、S期後半から前半への複製タイミング変化と遺伝子発現の活性化に先立って起きている例が多く観察されました(図4)。すなわち、三者の中では核内コンパートメントの変化が最も時間的に先行しており、これがその後に起きる複製タイミング変化や遺伝子発現の上昇を引き起こしている可能性が考えられました。
では、7日間にわたるES細胞分化の過程で、核内コンパートメントはいつどのように変化していくのでしょうか。これを調べるために、細胞集団のHi-C解析に加えて、1細胞ごとのDNA複製プロファイルを、最近独自に開発したscRepli-seq法を用いて全ゲノム解析しました。その結果、細胞集団中にはS期前半から後半を含むさまざまな細胞周期の細胞が存在し(図5a)、それらのDNA複製プロファイルは同じ培養日数の細胞集団を見るとグラフ上の一つの曲線(経路)で近似され、かつ培養0日から培養7日にかけて一定の傾向で変化していくことが認められました(図5b)。この結果から、個々の細胞においても、複製タイミングの変化に先行して起きる核内コンパートメント分布の変化が、細胞集団中で徐々に、しかし一様に起きている可能性が示されました(図5b)。また、核内コンパートメント分布と複製タイミングプロファイルは、分化後5日目に一過的にエピブラスト幹細胞(EpiSC)[20]に非常に近くなり、ES細胞が分化の過程でEpiSCに極めて近い状態を経て神経分化したと解釈できました(図5b、「5日」と「EpiSC」のグラフを比較)。
さらに、ゲノムDNA上でどのような領域が核内コンパートメント変化に関わっているかを調べるため、分化前後での細胞集団のHi-C解析データを詳細に比較しました。その結果、A/Bコンパートメントが入れ換わる現象は、コンパートメント内部に新しいコンパートメントが現れるのではなく、ほとんどの場合においてAコンパートメントとBコンパートメントの境界が移動することによって生じていることが分かりました(図6a、b)。 これらのコンパートメント境界は、ほぼ全てがTADとTADの間に存在しており、TADの構造を崩すような位置にコンパートメント境界が移動することはなく、変化の大半は約1Mb長のTAD一つ分の変化であることが分かりました(図6c)。また、体細胞からiPS細胞[21]へのリプログラミング[22]の過程についても同様の解析を行ったところ、やはりTAD一つ分を単位とする核内コンパートメント変化が観察されました(図6c)。最後に、細胞集団を対象としたHi-C解析でコンパートメント変化を示したTADについて、scRepli-seq解析を行ったところ、TADを単位とする複製タイミング変化が1細胞レベルでも多く観察できました(図6d)。
以上の結果を総合すると、細胞分化に伴うコンパートメント変化の実態は、AコンパートメントとBコンパートメントの境界に接しているTAD一つ分の核内配置変化であるといえます(図7)。コンパートメント変化は染色体上のさまざまな場所で起きており、これに引き続いて起きる複製タイミング変化や遺伝子発現変化の引き金となっている可能性も考えられます。また、TADは細胞集団を用いたHi-C実験で得られた概念であるため、個々の細胞にTADが存在するか否かはいまだに議論が分かれています。本研究結果は、TADを単位とする制御が1細胞レベルで確かに起きていることを示しており、個々の細胞におけるTADの存在を支持するものといえます(図7)。
注2)Takahashi, S., Kobayashi, S. & Hiratani, I. Epigenetic differences between naïve and primed pluripotent stem cells. Cell. Mol. Life Sci. 75, 1191–1203 (2018).
今後の期待
本研究により、マウスの細胞分化に伴う染色体の三次元構造変化、特にMbレベルの階層における変化の実態は、「TADを単位とする核内配置の変化である」と簡潔な表現で記述できるようになりました。この変化は遺伝子発現変化に先行していたことから、TADの核内配置制御の理解が、遺伝子発現制御の仕組みの理解にもつながると期待できます。
また技術的な評価として、scRepli-seq法が個々の細胞の染色体構造の推定に威力を発揮することがあらためて実証されました。DNA複製が染色体構造をよく反映しているため、scRepli-seq法は、染色体構造を指標とした細胞プロファイリング法として有用になる可能性があります。scRepli-seq法が細胞種の特定や均一性の判定など、さまざまな用途に応用できることを今後も実証していきたいと考えています。
原論文情報
- Hisashi Miura, Saori Takahashi, Rawin Poonperm, Akie Tanigawa, Shin-ichiro Takebayashi, Ichiro Hiratani, "Single-cell DNA replication profiling identifies spatiotemporal developmental dynamics of chromosome organization", Nature Genetics, 10.1038/s41588-019-0474-z
発表者
理化学研究所
生命機能科学研究センター 発生エピジェネティクス研究チーム
チームリーダー 平谷 伊智朗(ひらたに いちろう)
研究員 三浦 尚(みうら ひさし)
 平谷 伊智朗
平谷 伊智朗
 三浦 尚
三浦 尚
お問い合わせ先
理化学研究所 生命機能科学研究センター センター長室 報道担当
山岸 敦(やまぎし あつし)
Tel: 078-306-3095 / Fax: 078-306-3090
ayamagishi[at]riken.jp
報道担当
理化学研究所 広報室 報道担当
Tel: 048-467-9272 / Fax: 048-462-4715
お問い合わせフォーム
産業利用に関するお問い合わせ
補足説明
- 1.ES細胞(Embryonic Stem cells; ES cells)
胚性幹細胞とも呼ばれる。哺乳類生物の発生初期の胚盤胞期と呼ばれる時期の胚の一部、内部細胞塊と呼ばれる細胞群から作製する幹細胞細胞株のこと。 - 2.細胞分化
分化ともいう。多細胞生物の発生過程において、特殊化していない未分化の細胞が、より特殊化したタイプの細胞に変化するプロセスのことを指す。 - 3.トポロジカルドメイン(TAD; Topologically Associating Domain)
Hi-C法によって見出された染色体上に存在する約1Mb長の染色体ドメインのこと。相互作用頻度の高い一つながりのDNA領域に対応しており、TAD間には相互作用のギャップ(境界)が存在する。TADは、異なる細胞種間でも高度に保存されている普遍的な構造と考えられている。 - 4.塩基、塩基対
塩基とは、DNAを構成する主要な成分で、アデニン(A)、チミン(T)、グアニン(G)、シトシン(C)の4種類がある。ゲノム中で2本のデオキシリボ核酸(DNA)分子が2重らせん構造をとる際に、アデニン(A)とチミン(T)、グアニン(G)とシトシン(C)、という決まった組を作り、水素結合でつながった状態をとるが、その決まった各々の組のことを塩基対と呼ぶ。DNAの長さを表現するときに「~塩基対(base pairs: bp)のDNA」、あるいは単に「~塩基(bases : b)」という表現が用いられることが多い。Mbはメガベース、100万塩基(対)のこと。Kbはキロベース、1000塩基(対)のこと。 - 5.遺伝子発現、転写
ゲノムDNA上の遺伝子配列をもとにRNAが合成されることを転写、転写されたRNAがタンパク質に変換されることを翻訳と言う。このようにゲノムDNA上の遺伝子配列情報が、転写、翻訳のプロセスを経て細胞における構造や機能に変換される一連の過程のことを遺伝子発現と言う。しかし、RNA発現解析のことを遺伝子発現解析とも呼ぶように、遺伝子発現は転写と近い意味で使われることもある。 - 6.ゲノムDNA
生物の細胞が持っている全てのDNA配列情報のこと。真核生物の場合は核内にある染色体DNAを指すことが多い。 - 7.クロマチン
真核生物の核内でゲノムDNAが形成する高次構造体のこと。その主成分はDNAとヒストンと呼ばれるタンパク質。 - 8.細胞周期、分裂期、間期、S期
増殖中の真核細胞が繰り返す時間的なサイクル(期間)のことを細胞周期と呼ぶ。分裂期(M期)は細胞周期の中で細胞分裂を行う期間、間期は、分裂期以外の期間を指す。間期は、さらにG1期、S期、G2期に分けられる。S期は、DNA複製が行われる期間。 - 9.ヌクレオチド
DNAとRNA、すなわち核酸の構成要素。窒素を含む塩基と、五炭糖(デオキシリボース)、リン酸基よりなる。 - 10.ヌクレオソーム
全ての真核生物に共通するクロマチンの基本単位。4種類のヒストンタンパク質(H2A、H2、H3、H4)から構成されるヒストン8量体に146塩基対の2本鎖DNAが巻きついた構造をとる。 - 11.A/Bコンパートメント、核内コンパートメント
類似の性質を持つTAD同士は細胞核内で寄り集まって、核内でAおよびBコンパートメントという相互排他的な(互いにほぼ混ざり合わない)領域を核内空間に形成している。Aコンパートメントはよく転写されていて複製タイミングがS期前半の領域(ユークロマチン)とよく一致し、Bコンパートメントは転写が抑えられていて複製タイミングがS期後半の領域(ヘテロクロマチン)とよく一致する。 - 12.Hi-C法
3C(Chromosome Conformation Capture)法を発展させた全ゲノム解析手法のことで、細胞核内三次元空間におけるあらゆるゲノムDNA配列どうしの相対距離を測定して染色体の三次元的な構造を推定できる画期的な手法。 - 13.全ゲノム解析
ヒトやマウスなど、単一の生物種のゲノムを構成するすべてのDNA配列にわたって、特定の性質(RNA転写量、特定のDNA結合タンパク質の結合性など)を次世代シーケンサー(NGS; Next Generation Sequencer)を用いて網羅的に調べる解析のこと。 - 14.DNA複製、DNA複製タイミング
DNA複製は、ゲノムDNA複製、ゲノム複製と同義。細胞分裂における核分裂の前に、DNAポリメラーゼという酵素によって、ゲノムDNAが過不足なく倍加される過程。DNA複製タイミングとは、ゲノムDNA複製の時間的制御のこと。S期において、各ゲノム領域はそれぞれ特徴的な時期に複製され、その時間的制御は個々の細胞間でさほどゆらがない。 - 15.scRepli-seq法(single-cell DNA Replication sequencing法)
増殖中の細胞でゲノムDNAが倍加していく過程を1細胞レベルで全ゲノム解析する方法。 - 16.エピブラスト
胚盤葉上層とも言う。哺乳類や鳥類の胚発生胚初期に一過的に出現し、将来、成体を形成する全ての細胞に分化する多能性を持つ細胞集団。 - 17.エピブラスト様細胞(EpiLC; Epiblast-like cells)
マウスES細胞を特定の培養条件で培養して分化誘導することで生じる、初期エピブラストの細胞に近い遺伝子発現プロファイルを持った細胞。 - 18.SFEBq法(無血清凝集浮遊培養法)
ES細胞などを酵素によりバラバラに分散させ、それを3000個程度の細胞の塊に再凝集させたものを分化培養の材料に用いる。この細胞凝集塊を、血清や転写因子などの神経分化阻害効果のある成分を一切含まない特殊な培養液に浮遊させて数日培養することで、9割以上の細胞を中枢神経系の細胞に分化させることが可能である。SFEBqは、Serum-free Floating culture of Embryoid Body-like aggregates with quick reaggregationの略。 - 19.核ラミナ
ほぼ全ての真核細胞の核膜の内側に存在する、厚さ約30-100 nmの網状の構造。ラミンなどの中間径フィラメントと膜結合タンパク質から構成されており、遺伝子が転写されていないゲノム領域との親和性が高い。 - 20.エピブラスト幹細胞(EpiSC; Epiblast-derived stem cells)
マウス着床後のエピブラストから樹立された多能性幹細胞の一種。マウスES細胞とは遺伝子発現プロファイルや性質が大きく異なる。 - 21.iPS細胞(induced pluripotent stem cells)
人工多能性幹細胞とも呼ばれる。iPS細胞は体細胞をリプログラミングすることによって作られる多能性を持った細胞株であり、性質はES細胞とよく似ている。 - 22.リプログラミング
成体の細胞(体細胞)の核を未分化な状態に初期化(リセット)すること。
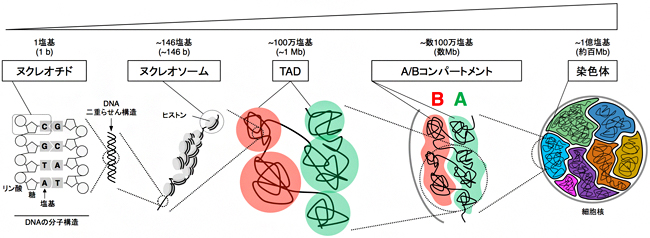
図1 哺乳類の染色体の階層構造
哺乳類の染色体の階層構造を大きさの順に右から示す。間期の染色体は、分裂期ほど密には折り畳まれていないが、それぞれの染色体は核内で別々の空間を占めている。本研究では、TADとA/Bコンパートメントで構成されるMbレベルの階層構造に着目し、細胞分化過程でこれらがどのように変化するのかを調べた。
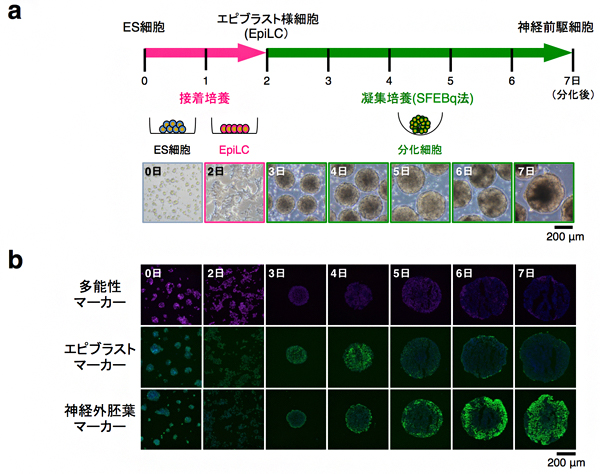
図2 マウスES細胞から神経前駆細胞への分化誘導
- a) 本研究で用いたマウスES細胞から神経前駆細胞への分化誘導法。マウスES細胞を2日間の接着培養によりエピブラスト様細胞(EpiLC)に分化させ、その後EpiLCを用いて凝集培養(SFEBq法)を5日間行うことで神経前駆細胞へと分化させた。
- b) 分化に伴い、多能性マーカータンパク質(Oct4)の発現は減少する一方、エピブラストマーカー(Eomes)の発現は分化後4日目に一過性に上昇してその後すぐに減少し、最後に、神経外胚葉マーカー(Sox1)の発現が一様に上昇した。この結果より、マウスES細胞はエピブラストの状態を経て比較的均一に神経前駆細胞に分化したと判断できた。
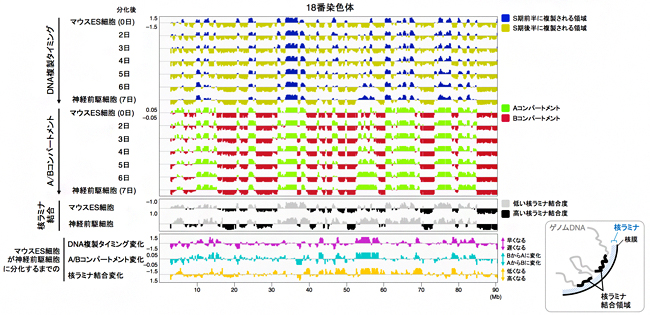
図3 DNA複製タイミング、A/Bコンパートメント、核ラミナ結合の協調的な変化
7日間にわたるES細胞分化過程におけるDNA複製タイミング変化、A/Bコンパートメント変化、核ラミナ結合の変化を示す。最下段には、前述の三つの性質について、分化前後の変化の度合いを示した。核ラミナ結合領域(黒)はBコンパートメント(赤)と一致する傾向を示すことから、Aコンパートメントは核の中心側、Bコンパートメントは核膜側に位置すると考えられる。
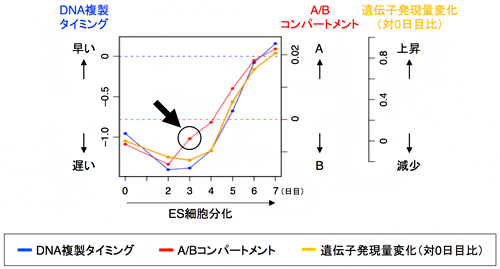
図4 分化に伴うA/Bコンパートメント、DNA複製タイミング、遺伝子発現の変化の順序
分化に伴いBからAへコンパートメント変化する一群のゲノム領域の挙動。BからAへのコンパートメント変化(赤)が、S期後半から前半へのDNA複製タイミング変化(青)と遺伝子発現の上昇(黄)に先行していることが分かる(矢印)。グラフはいずれも一群のゲノム領域の平均値を示す。
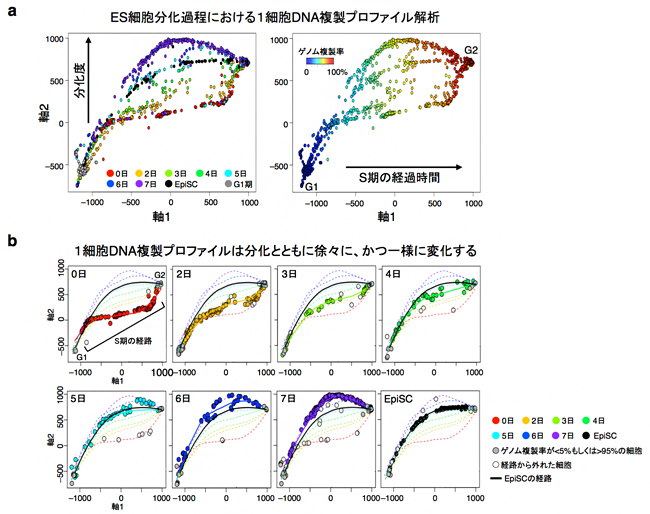
図5 ES細胞分化に伴う1細胞DNA複製プロファイルの変化
- a) SPRING法注3)というデータ解析手法を用いて、ES細胞分化過程(分化後0~7日)とエピブラスト幹細胞(EpiSC)の1細胞DNA複製プロファイル(scRepli-seqデータ)のばらつきを、二次元のグラフで可視化した(G1期、S期を含めて合計884細胞)。結果的に、縦軸(軸2)は分化の度合いを示し(左)、横軸(軸1)は各細胞のゲノム複製率の分布からS期の経過時間を示していた(右)。
- b) 7日間の分化に伴う日ごとの1細胞DNA複製プロファイルが描く曲線(S期の経路)。S期の経路は、分化に伴い一定の傾向で変化していくが、細胞集団中で見ると一様に変化していく。分化後5日目には、EpiSCと非常に近くなる。点線は日ごとの近似曲線(経路)を示す。
注3)Weinreb C. et al., Gene expression SPRING: a kinetic interface for visualizing high dimensional single-cell expression data. Bioinformatics 34,1246-1248 (2018).
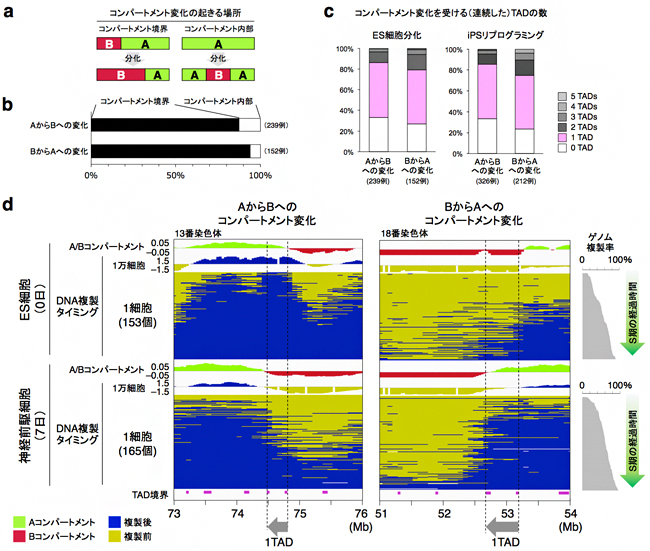
図6 A/Bコンパートメント変化=その境界におけるTAD一つ分の変化
- a) A/Bコンパートメント変化の起きる場所は、その境界もしくは内部、二通りの可能性が考えられる。
- b) A/Bコンパートメント変化の大半はコンパートメント境界で起きる。
- c) A/Bコンパートメント変化を受ける(連続した)TADの数。変化の大半は1TAD単位で起きている。これはES細胞分化(左)のみならず、体細胞からiPS細胞へのリプログラミング(右)の過程でも見られた。
- d) A/Bコンパートメント境界に位置する1TAD単位の変化はscRepli-seq法による1細胞DNA複製プロファイル解析でも観察された。AからB(左)、BからAのコンパートメント変化(右)を各1例ずつ示す。
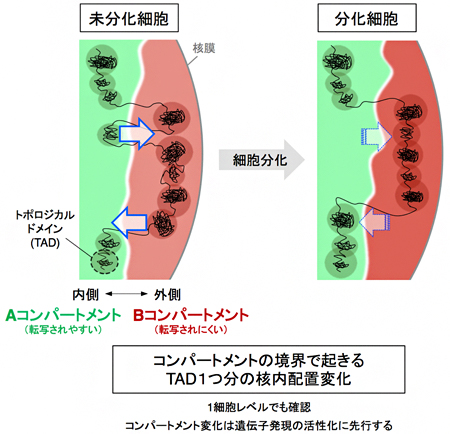
図7 分化に伴うTADを単位とするA/Bコンパートメント変化のモデル
未分化な細胞が分化するとき、特にA/Bコンパートメントの境界に位置する一つ分のTADの核内配置が変わり、それまでとは別のコンパートメントに移動することが1細胞レベルでも観察できた。配置が変わったTAD内部では、配置変化に引き続いて遺伝子発現の変化が引き起こされ、このようなTADが一定以上存在すると細胞分化が決定される可能性が考えられた。
